Работа финансировалась грантом РФФИ ¹ 06-04-48031-а.
Последовательности праймеров:
CSN1S1f: 5'-GAAGAAGCAGCAAGCTGG-3'
CSN1S1r: 5'-TGCATGTTCTCATAATAACC-3'
CSN3f: 5'-GAAATCCCTACCATCAATACC-3'
CSN3r: 5'-CCATCTACGCTAGTTTAGATG-3'
При амплификации в полимеразной цепной реакции (ПЦР) с использованием в качестве матрицы тотальной ДНК быков черно-пестрой породы получили фрагменты длиной 319 п.н. для гена CSN1S1 и 270 п.н. для гена CSN3. Олигонуклеотидное мечение ДНК-зондов для выполнения скринга геномных библиотек проводили при помощи набора «Рандом-прайм» («Ферментас», Литва) с использованием 2 Mbq a-32P-dATP. Состав реакционной смеси: ДНК-матрица — 15-30 нг; раствор гексануклеотидов-праймеров (10 мкМ) — 2 мкл; бидистиллированная H2O — до 30 мкл. Смесь денатурировали нагреванием в термоблоке до 95 °С в течение 3-5 мин и быстро охлаждали на льду. Затем добавляли: ½10 буфер для кленовского фрагмента ДНК-полимеразы — 5 мкл; смесь dNTPs (кроме dATP) (0,1 мМ) — 10 мкл; a-32P-dATP — 2 Mbq; кленовский фрагмент ДНК-полимеразы I (10 ед/мкл) — 0,5 мкл; бидистиллированную H2O — до 50 мкл. Полученную смесь инкубировали в течение 1 ч при 37 °С, реакцию останавливали добавлением 1 мкл 0,5 М ЭДТА.
Предгибридизацию реплик гридированной библиотеки CHORI-240 Сегмент I (http://bacpac.chori.org/bovine240.htm) проводили инкубированием при 65 °С в растворе Church Buffer (0,342 М Na2HPO4, 0,158 М NaH2PO4, 7 % SDS, 1 % BSA, pH 7,2) в течение 1 ч. Затем в раствор добавляли по 25 мкл меченого ДНК-зонда на библиотеку и инкубировали при той же температуре в течение 16 ч.
Отмывки проводили по следующей схеме: ½2 стандартный солевой раствор (SSC), 0,1 % SDS при 65 °С в течение 7 мин; ½2 SSC, 0,1 % SDS при комнатной температуре в течение 7 мин; ½0,5 SSC, 0,1 % SDS при комнатной температуре в течение 7 мин; ½0,1 SSC, 0,1 % SDS при комнатной температуре в течение 7 мин. Затем фильтры заворачивали в полиэтиленовую пленку Sarah Wrap и помещали в кассеты.
Анализ результатов гибридизации проводили методом авторадиографии на рентгеновской пленке Kodak или прямым сканированием на приборе Molecular Imager FX-Scan («Bio-Rad», США). Полученные изображения обрабатывали при помощи пакета компьютерных программ Quantity-One («Bio-Rad», США).
При проверке клонов на содержание нуклеотидных последовательностей кластера казеиновых генов КРС с помощью ПЦР матрицей служили последовательности геномных клонов (условия ПЦР и праймеры приведены в табл. 1). Длину вставки в BAC-клонах CH240-130F23 и CH240-035O12 определяли после обработки рестриктазой NotI и электрофоретического разделения фрагментов в агарозном геле.
Результаты. При гибридизации фильтров с последовательностями генов CSN1S1 и CSN3 было выявлено соответственно 6 и 11 BAC-клонов. Клоны CH240-130F23 и CH240-035O12, как оказалось, содержат гены CSN1S1, CSN1S2, CSN2, STATH и CSN3 (табл. 2). Пять клонов выявлены как позитивные при ПЦР-амплификации с использованием праймеров, специфических для генов CSN2, STATH, CSN1S2 и CSN3 (см. табл. 2).
1. Праймеры и условия полимеразной цепной реакции (ПЦР) при проверке клонов на содержание последовательностей кластера казеиновых генов крупного рогатого скота |
||||
Локус |
Идентификационный номер |
Последовательность праймеров |
Температура |
Длина фрагмента, п.н. |
CSN1S1 |
X59856 |
f 5'-atccaaaggcttcaactgct-3' |
59 |
178 |
CSN1S2 |
M94327 |
f 5'-tctcctcctaggattggaaaga-3' |
59 |
199 |
CSN2 |
X14711 |
f 5'-acagcctcccacaaaacatc-3' |
60 |
224 |
STATH |
AY154893 |
f 5'-tgaagaggaacaccggctta-3' |
60 |
212 |
CSN3 |
AY380229 |
f 5'-atttatggccattccaccaa-3' |
60 |
166 |
По данным M. Rijnkels с соавт., расстояние между генами CSN1S1 и CSN3 у КРС — примерно 250 т.п.н (4). Размер вставки BAC-клонов в геномной библиотеке КРС CHORI-240 Сегмент I составляет в среднем 180 т.п.н. и варьирует от 100 до 270 т.п.н. (http://bacpac.chori.org/bovine240.htm). Проведенная нами рестрикция ДНК-клонов CH240-130F23 и CH240-035O12 позволила установить размеры вставки — соответственно 263 и 265 т.п.н. То есть клоны CH240-130F23 и CH240-035O12 включают все кодирующие последовательности кластера и около 15 т.п.н. 5'-фланкирующей области. Вероятно, предполагаемый локус-контролирующий район (LCR) кластера может быть обнаружен в пределах клона CH240-130F23, поскольку такого рода последовательности обычно расположены в 5'-фланкирующих участках на расстоянии до 35 т.п.н. от стартового кодона дистального гена (8).
2. Результаты полимеразной цепной реакции (ПЦР) с использованием праймеров, специфических для казеиновых генов крупного рогатого скота |
|||||
Клон |
CSN1S1 |
CSN2 |
STATH |
CSN1S2 |
CSN3 |
CH240-046B08 |
- |
- |
- |
- |
- |
CH240-035O12 |
+ |
+ |
+ |
+ |
+ |
CH240-115O19 |
- |
+ |
+ |
+ |
+ |
CH240-130F23 |
+ |
+ |
+ |
+ |
+ |
CH240-124F17 |
- |
- |
- |
- |
- |
CH240-126C05 |
- |
- |
- |
- |
- |
CH240-258E02 |
- |
- |
- |
- |
- |
CH240-200N21 |
- |
+ |
+ |
+ |
+ |
CH240-254P04 |
- |
- |
- |
- |
- |
CH240-260O08 |
- |
- |
- |
- |
- |
CH240-274P22 |
- |
+ |
+ |
+ |
+ |
CH240-270N05 |
- |
- |
- |
- |
- |
CH240-250M04 |
- |
+ |
+ |
+ |
+ |
CH240-262B19 |
- |
- |
- |
- |
- |
CH240-200L09 |
- |
- |
- |
- |
- |
CH240-233L10 |
- |
- |
- |
- |
- |
CH240-215M02 |
- |
- |
- |
- |
- |
Следовательно, полученный контиг содержит не только все известные гены казеинового кластера КРС, но и значительную часть 3'-фланкирующей области.
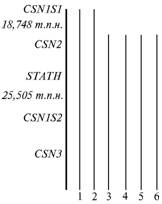
Согласно имеющимся сведениям о геноме КРС, гены STATH и CSN1S2 расположены в пределах контига NW_936323 соответственно в положениях 821-1090 и 26595-38541 п.н., в то время как гены CSN2 и CSN1S1 — в контиге NW_973412 в положениях 418 544-426 774 и 445 522-463 059 п.н.; информация о локализации гена CSN3 отсутствует (http://www.ncbi.nlm.nih.gov/genome/guide/cow/index.html). Таким образом, расстояние между генами STATH и CSN1S2 составляет 25505 п.н., между генами CSN2 и CSN1S1 — 18748 п.н. (рис.). На основании известного размера вставки в клонах библиотеки CHORI-240 и приблизительной оценки расстояния между генами CSN1S1 и CSN3 в 200 т.п.н. (4) можно заключить, что представленный в работе контиг содержит до 70 т.п.н. 3'-фланкирующей области. Максимальное покрытие контига (½7) наблюдается в центральной и 3'-дистальной части кластера (см. рис.).
|
Контиг BAC-клонов из библиотеки CHORI-240, включающий гены CSN1S1, CSN2, STATH, CSN1S2 и CSN3. Геномные BAC-клоны: 1 — CH240-035O12, 2 — CH240-130F23, 3 — CH240-115O19, 4 — CH240-200N21, 5 — CH240-274P22, 6 — CH240-250M04. |
Итак, посредством скрининга гридированной BAC-библиотеки CHORI-240, содержащей 6,1 геномного эквивалента крупного рогатого скота, с использованием в качестве ДНК-зондов фрагментов генов CSN1S1 и CSN3 выявлены 17 протяженных геномных клонов со средним размером вставки 180 т.п.н. Методом полимеразной цепной реакции с праймерами, специфическими для генов, входящих в состав кластера, показано, что клоны CH240-130F23 и CH240-035O12 содержат гены CSN1S1, CSN1S2, CSN2, STATH и CSN3 и еще пять BAC-клонов содержат гены CSN2, STATH, CSN1S2 и CSN3. То есть все известные гены кластера представлены в контиге из семи полученных геномных клонов.
Л И Т Е Р А Т У Р А
- R i j n k e l s M., M e e r s h o e k E., d e B o e r H.A. e.a. Physical map and localization of the human casein gene locus. Mam. Genome, 1997, 8(4): 285-286.
- R i j n k e l s M., M e e r s h o e k E., d e B o e r H.A. e.a. Structure and expression of the mouse casein gene locus. Mam. Genome, 1997, 8(1): 9-15.
- R i j n k e l s M., K o o i m a n P.M., d e B o e r H.A. e.a. Organization of the bovine casein gene locus. Mam. Genome, 1997, 8(2): 148-152.
- R i j n k e l s M., E l n i t s k i L., M i l l e r W. e.a. Multispecies comparative analysis of a mammalian-specific genomic domain encoding secretory proteins. Genomics, 2003, 82(4): 417-432.
- M i l e n k o v i c D., M a r t i n P., G u e r i n G. e.a. A specific pattern of splicing for the horse alphaS1-Casein mRNA and partial genomic characterization of the relevant locus. Gen., Sel., Evol., 2002, 34(4): 509-519.
- F u j i w a r a Y., M i w a M., N o g a m i M. e.a. Genomic organization and chromosomal localization of the human casein gene family. Hum. Genet., 1997, 99(3): 368-373.
- P a u l o i n A., R o g e l - G a i l l a r d C., P i u m i F. e.a. Structure of the rabbit alphas1- and beta-casein gene cluster, assignment to chromosome 15 and expression of the alphas1-casein gene in HC11 cells. Gene, 2002, 283(1-2): 155-162.
- L i Q., H a r j u S., P e t e r s o n K.R. Locus control regions: coming of age at a decade plus. Trends in Genetics, 1999, 15(10): 403-408.
ГНУ ВПО Всероссийский НИИ генетики и разведения сельскохозяйственных животных Россельхозакадемии, |
Поступила в редакцию 29 мая 2007 года |
CONSTRUCTION OF THE CONTIGUUM OF GENOMIC CLONES OF CASEIN GENES CLUSTER IN CATTLE
K.A. Fomichev, T. Malevski, S. Kaminski, A.L. Sazanova, A.A. Sazanov
S u m m a r y
The authors constructed the contiguum of cattle casein genes cluster in the form of overlapping BAC-clones. By means of screening gridded BAC-library CHORI-240 including 6.1 of genome equivalent KPC with using as DNA-sonde of genes fragments CSN1S1 and CSN3 the 17 long genome clones were revealed with average size of insertion of 180 kb. By the PCR method with primers specified for cluster genes it was shown that the clones CH240-130F23 and CH240-035O12 contain the genes CSN1S1, CSN1S2, CSN2, STATH and CSN3 and else five BAC-clones contain the genes CSN2, STATH, CSN1S2 and CSN3. That is all known cluster genes are presented in contiguum from seven obtained genomic clones