doi: 10.15389/agrobiology.2019.6.1177rus
УДК 636.294:636.086.2/.3:579
Исследование выполнено при поддержке гранта РНФ для реализации научного проекта № 17-76-20026 «Микробиоценоз рубца Rangifer tarandus Арктических регионов России как фундаментальная основа получения перспективных биотехнологий для сельскохозяйственных животных».
МЕСТО ОБИТАНИЯ КАК ОПРЕДЕЛЯЮЩИЙ ФАКТОР ФОРМИРОВАНИЯ МИКРОБИОМА РУБЦА У СЕВЕРНЫХ ОЛЕНЕЙ В АРКТИЧЕСКОЙ РОССИИ
Л.А. ИЛЬИНА1, К.А. ЛАЙШЕВ2, Е.А. ЙЫЛДЫРЫМ1, В.А. ФИЛИППОВА1, Т.П. ДУНЯШЕВ1, А.В. ДУБРОВИН1,
Д.В. СОБОЛЕВ1, Н.И. НОВИКОВА1, Г.Ю. ЛАПТЕВ1, А.А. ЮЖАКОВ2, Т.М. РОМАНЕНКО3, Ю.П. ВЫЛКО3
Северный олень (Rangifer tarandus) географически изолирован от других подвидов жвачных семейства Cervidae, что предполагает формирование специфических микробных сообществ рубца у этого вида. Известно, что приуроченность к определенным экологическим условиям может оказывать значимое влияние на состав микробиоты рубца жвачных. В настоящей работе с применением молекулярно-биологического анализа впервые показано, что одним из ключевых факторов, влияющих на состав микробиома рубца у северного оленя, служит место обитания. Цель работы состояла в оценке региональных и половозрастных особенностей микробиома рубца северных оленей из различных природно-климатических зон Российской Федерации с использованием T-RFLP-анализа (terminal restriction fragment length polymorphism) и количественной ПЦР. Объектом исследования были 58 особей северных оленей ненецкой породы — телята (4-8 мес) и взрослые особи. Содержимое рубца северных оленей отбирали в летне-осенний период 2017 года (n ≥ 3 из каждой возрастной группы) в Ямало-Ненецком автономном округе (АО) (п.г.т. Харп, лесотундровая природно-климатическая зона), Ненецком АО (п. Нельмин-Нос, тундровая природно-климатическая зона) и Мурманской области (ст. Лопарская, тундровая природно-климатическая зона). Также для анализа были отобраны образцы пастбищной растительности, составлявшие основу рациона северных оленей в каждом регионе. Общее количество бактерий, архей и грибов класса Neocallimastigales в содержимом рубца анализировали методом количественной ПЦР, состав бактериального сообщества — методом Т-RFLP. Основным детерминантом всех компонент микробного сообщества рубца северных оленей оказались региональные условия обитания, что, по всей видимости, обусловлено различиями в составе пастбищного рациона и эпизоотической ситуацией в стаде. Наименьшее сходство с другими регионами по составу содержимого рубца было выявлено у образцов из Мурманской области. Половозрастные различия животных оказались менее значимыми, однако вносили определенный вклад в соотношение микроорганизмов в рубце. Выявленные изменения микробиоты были неодинаковы у особей из разных регионов. Так, у северных оленей старше 2 лет из Мурманской области и Ямало-Ненецкого АО наблюдалось достоверное увеличение численности бактерий (p < 0,05), в отличие от особей из Ненецкого АО. Вне зависимости от региона отбора образцов доминирующими в содержимом рубца были представители филума Firmicutes,в меньшей степени детектированы бактерии филумов Bacteroidetes, Proteobacteria, Actinobacteria, доля других выявленных таксонов (Tenericutes, Fusobacteria, Cyanobacteria) в сообществе была минорной. В целом, отмечены значимые изменения представленности ряда таксонов в связи с показателями питательности пастбищного рациона северных оленей. Помимо рубцовых микроорганизмов, традиционно связанных с деградацией целлюлозы, статистически значимую связь установили между содержанием клетчатки в пастбищных кормах и численностью членов семейств Veillonellaceae (r = -0,75), Nostocaceae (r = 0,52), Rivulariaceae (r = -0,88). Значимой взаимосвязи количества условно-патогенных микроорганизмов из филумов Fusobacteria, Tenericutes (семейство Mycoplasmataceae) и Proteobacteria (семейства Enterobacteriaceae, Campylobacteraceae) с показателями питательности кормов и представленностью других групп микроорганизмов мы не отмечали. Для углубленного изучения выявленных закономерностей, их причин и механизмов требуются дополнительные исследования.
Ключевые слова: T-RFLP-анализ, количественная ПЦР, микробиом рубца, северный олень, Rangifer tarandus, Российская Арктика.
растительноядных жвачных. Географическая изоляция этого вида от других подвидов семейства Cervidae (1) предполагает не только анатомические и морфологические особенности в строении пищеварительной системы, но и формирование специфических микробных сообществ рубца (2-4). Интерес к исследованию сложных симбиотических сообществ рубца северных оленей также связан с изучением адаптационных приспособлений организма этих животных к неблагоприятным факторам, в частности возможности эффективно использовать скудные растительные ресурсы пространств тундры, лесотундры и северной тайги (5). На формирование микробных сообществ в рубце жвачных животных влияет комплекс взаимообусловленных факторов: разнообразие источников растительной клетчатки, взаимодействия между микроорганизмами различных групп, таксономическое разнообразие ферментных систем, молекулярное строение ферментов, физиология микроорганизмов, экологические аспекты (6-8). В связи с этим адаптационные приспособления северных оленей должны рассматриваться с учетом условий их местообитания, доступности питательных ресурсов и других факторов (9).
Приуроченность к определенным экологическим условиям оказывает влияние на состав микробиоты рубца у представителей жвачных (10). Однако такие исследования сосредоточены преимущественно на анализе представленности отдельных групп микроорганизмов, например целлюлозолитических. Так, существенное различие микробных сообществ рубца показано для двух географически отделенных подвидов северных оленей Норвегии — Rangifer tarandus (Eurasian tundra reindeer, R. tarandus tarandus) материковой части страны и R. tarandus platyrhynchus (Svalbard reindeer, R. tarandus platyrhynchus) высокоарктического архипелага Шпицберген, расположенного между Норвегией и Северным Полюсом. При этом у R. tarandus platyrhynchus, которые в течение 8-10-месячного зимнего периода вынуждены питаться кормом с высоким содержанием лигнина, отмечено в 6-14 раз большее содержание целлюлозолитических бактерий, чем у R. tarandus tarandus (1). Однако бактерии, связанные с процессами ферментации растительных кормов, были сходными по видовому составу: Pepto-streptococcus anaerobius, Lachnospira multiparus, Butyrivibrio fibrisolvens, Eubac-terium ruminantium, Selenomonas ruminantium, Fibrobacter succinogenes, Eubacterium pyruvovorans и Fusocillus sp. Также, по данным T.H. Aagnes с соавт. (11), подвид R. tarandus tarandus имеет серьезные ограничения в переваривании клетчатки.
Молекулярно-генетические технологии, такие как T-RFLP-анализ (terminal restriction fragment length polymorphism) и NGS-секвенирование (next generation sequencing), открыли новые возможности в изучении структуры микробных сообществ, что позволяет дать глубокую характеристику их биоразнообразия, выявляя не только доминирующие таксоны, но и другие компоненты, в том числе некультивируемые микроорганизмы (12-15). Однако проведенные до настоящего времени исследования микробиома рубца северных оленей единичны (16, 17).
В настоящей работе с применением молекулярно-биологического анализа впервые показано, что одним из ключевых факторов, влияющих на формирование микробиома рубца северного оленя (Rangifer tarandus), служит место обитания, что, вероятно, связано с особенностями кормовой базы животных.
Цель работы состояла в оценке региональных и половозрастных особенностей микробиома рубца северных оленей, обитающих в различных природно-климатических зонах Российской Федерации, с использованием T-RFLP-анализа и количественной ПЦР.
Методика. Объектом исследования были 58 особей северных оленей (Rangifer tarandus) ненецкой породы — телята (4-8 мес) и взрослые особи (самцы и самки). Содержимое рубца северных оленей отбирали в летне-осенний период 2017 года (n ≥ 3 из каждой возрастной группы) в Ямало-Ненецком автономном округе (АО) (п.г.т. Харп, лесотундровая природно-климатическая зона), Ненецком АО (п. Нельмин-Нос, тундровая природно-климатическая зона) и Мурманской области (ст. Лопарская, тундровая природно-климатическая зона).
Общее количество бактерий, архей и грибов класса Neocallimastigales в содержимом рубца анализировали методом количественной ПЦР (qRT-PCR) с использованием набора реактивов для ПЦР в реально времени (ПЦР-РВ в присутствии интеркалирующего красителя EVA Green (ЗАО «Синтол», Россия) и праймеров F — 5'-ACTCCTACGGGAGGCA-GCAG-3', R — 5'-ATTACCGCGGCTGCTGG-3' (бактерии), F — 5'-AG-GAATTGGCGGGGGAGCAC-3', R — 5'-GCCATGCACCWCCTCT-3' (археи), F — 5'-GCACTTCATTGTGTGTACTG-3', R — 5'-GGATGAAACTCG-TTGACTTC-3' (грибы) на детектирующем амплификаторе DT Lite-4 (НПО «ДНК-Технология», Россия) в следующем режиме: первый цикл — 3 мин при 95 °С (1 повтор); второй цикл — 13 с при 95 °С, 13 с при 57 °С, 30 с при 72 °С (40 повторов).
Бактериальное сообщество рубца изучали при помощи метода Т-RFLP (18). ДНК из образцов извлекали набором для выделения ДНК Genomic DNA Purification Kit («Fermentas», Литва) в соответствии с инструкциями производителя. Полимеразную цепную реакцию проводили на амплификаторе Verity («Life Technologies», США) с бактериальными праймерами 63F (5'-CAGGCCTAACACATGCAAGTC-3') с меткой на 5'-конце (флуорофор WellRed D4, «Beckman Coulter», США) и 1492R (5'-TAC-GGHTACCTTGTTACGACTT-3'), амплифицирующими фрагмент гена 16S pРНК с 63-й по 1492-ю позицию, используя режим: первый цикл — 3 мин при 95 °С (1 повтор); второй цикл — 30 с при 95 °С, 40 с при 55 °С, 60 с при 72 °С (35 повторов); третий цикл — 5 мин при 72 °С. Итоговую концентрацию полученной тотальной ДНК определяли при помощи флуориметра Qubit 2 («Invitrogen», США) с применением наборов Qubit dsDNA BR Assay Kit («Invitrogen», США) согласно рекомендациям производителя.
Ампликоны фрагмента гена 16S pРНК, меченные флуоресцентной меткой, очищали в соответствии со стандартной методикой (19). По 30-50 нг ДНК обрабатывали рестриктазами MspI, HaeIII, HhaI («Fermentas», Литва) 2 ч при температуре 37 °С. Рестрикционные продукты осаждали с помощью этанола, затем вносилось 0,2 мкл маркера молекулярной массы Size Standart-600 («Beckman Coulter», США) и 10 мкл формамида Sample Loading Solution («Beckman Coulter», США). Анализ проводился на секвенаторе CEQ 8000 («Beckman Coulter», США); погрешность прибора CEQ 8000 — не более 5 %. Высоту пиков, их площадь определяли при помощи программного обеспечения Fragment Analysis («Beckman Coulter», США), в результате чего выделяли подтипы (филотипы) и рассчитывали их относительное количество в составе микробного сообщества. Таксономическую принадлежность бактерий устанавливали с использованием базы данных (http://mica.ibest.uidaho.edu/trflp.php).
Образцы пастбищной растительности, составлявшие основу рациона северных оленей, отбирали в каждом регионе и проводили их ботаническое описание. Кроме того, определяли соотношение различных видов растений в рационе и его питательность.
Статистическую обработку результатов осуществляли методом дисперсионного анализа с использованием программного обеспечения Microsoft Excel 2010. Определяли средние значения (M), стандартные ошибки средних (±SEМ), достоверность различий оценивали по t-критерию Стьюдента. Расчет коэффициентов корреляции Пирсона и оценку сходства бактериальных сообществ методом главных компонент (Principal Component Analysis, PCA) на основе коэффициента Брея-Кертиса, учитывающего количество и относительную численность определенных таксонов, осуществляли с помощью программы Past (http://folk.uio.no/ohammer/past/).
Результаты. На рисунке 1 показаны регионы, где осуществляли отбор образцов. Усредненный состав и питательность летне-осеннего пастбищного рациона северных оленей представлены в таблице 1.
Физиология северных оленей, в том числе состояние рубцовой микрофлоры, испытывает гораздо меньшую антропогенную нагрузку, чем у одомашненных жвачных, в частности у крупного рогатого скота. Вместе с тем роль микробного сообщества рубца у жвачных в обеспечении питания особенно значима, поскольку их рацион состоит из крайне бедных по пищевой ценности растительных кормов (20). Известно, что пищеварительная система северных оленей имеет уникальные приспособления для микробиологической ферментации скудных растительных ресурсов в естественных природных условиях обитания, что может оказывать существенное селектирующее давление на структурно-функциональную организацию рубцового микробиома.
Рубец жвачных населен различными группами симбиотических микроорганизмов (4-7). В наших исследованиях у животных из Ямало-Ненецкого АО было отмечено достоверно большее (p < 0,05) количество бактерий и грибов-хитридиомицетов по сравнению с особями из других регионов (табл. 2). В рубце северных оленей из Мурманской области и Ненецкого АО зафиксировали достоверно большее (p < 0,05) количество архей по сравнению с животным из Ямало-Ненецкого АО.
Половозрастные различия животных по численности основных групп микроорганизмов оказались менее значимы по сравнению с региональными особенностями. Однако выявленные изменения микробиоты были неодинаковы для особей из разных регионов. Так, у северных оленей старше 2 лет из Мурманской области и Ямало-Ненецкого АО наблюдалось достоверное увеличение численности бактерий (p < 0,05) по сравнению с особями из Ненецкого АО. Достоверных изменений численности хитридиомицетов у животных из Мурманской области и Ненецкого АО с возрастом не наблюдали, тогда как у особей из Ямало-Ненецкого АО их отмечали.
Можно предположить, что обнаруженные нами закономерности изменения численности микроорганизмов в сообществе рубца северных оленей взаимосвязаны со структурой летнего пастбищного рациона и его питательностью, что согласуется с сообщениями зарубежных исследователей. Так, M.A. Olsen с соавт. (21) показали снижение в зимнее время численности жизнеспособных зооспор хитридиомицетов, которые обладают широким спектром многофункциональных полисахаридных ферментов (22-23), в содержимом рубца у Norwegian reindeer, обитающих на естественных пастбищах. Аналогичные результаты получены для метаногенных архей, численность которых снижалась в рубце у R. tarandusввесенний период по сравнению с осенним (24). При этом для северных оленей R. tarandus platyrhynchus архипелага Шпицберген показано отсутствие существенных изменений в рубце количества метаногенов, бактерий и простейших в связи со сменой состава растительности естественных пастбищ в осенний и весенний периоды (25).
Анализ сходства бактериальных сообществ рубца у исследованных нами особей на основе метода главных компонент (рис. 2) показал, что все образцы подразделялись на три основные группы, соответствующие отдельным регионам: Мурманской области, Ямало-Ненецкому и Ненецкому АО. Обнаруженные закономерности предположительно обусловливались различиями в составе пастбищного рациона и эпизоотической ситуацией в стаде, поскольку метеорологические параметры в регионах были сходными для летне-осеннего периода, в который проводилось исследование. Интересно, что половозрастные особенности животных, определенные методом главных компонент, показали менее значительное разделение образцов на подгруппы. В целом, наиболее значимые различия детектировали в составе бактериального сообщества рубца между группами молодняка (до 2 лет) и взрослыми особями (старше 2 лет) (см. рис. 2). Стоит отметить, что существенных изменений в составе микробного сообщества рубца у важенок (самок) и хоров (самцов) мы не обнаружили.
Вне зависимости от региона отбора образцов у исследованных нами особей R. tarandus доминирующими в содержимом рубца были представители филума Firmicutes,в меньшей степени детектировали бактерии филумов Bacteroidetes, Proteobacteria, Actinobacteria,доля других выявленных таксонов (Tenericutes, Fusobacteria, Cyanobacteria) в сообществе была минорной, значительное количество бактериальных последовательностей не удалось идентифицировать до уровня филума.
Наибольший интерес у исследователей при изучении микробных сообществ рубца жвачных связан с микроорганизмами — продуцентами ферментов, не синтезируемых организмом хозяина, прежде всего целлюлозолитическими. Эти микроорганизмы, благодаря использованию различных ферментов (целлюлаз, гемицеллюлаз, ксиланаз, гликозидгидролаз и т.д.) участвуют в деградации растительной биомассы с образованием метаболитов, в том числе летучих жирных кислот (ЛЖК): пропионата, ацетата, бутирата, изобутирата, валерата, изовалерата и 2-ме-тилбутировой, гексановой и гептановой кислот (6, 7).
Представители бактериальных филумов Firmicutes и Bacteroidetes составляют преобладающую часть микробного сообщества в рубце жвачных (26, 27), что предполагает важную экологическую роль этих микроорганизмов, вероятно, благодаря их широкому метаболическому потенциалу, в том числе ферментации растительных полисахаридов. Высокая пропорциональная доля филумов Firmicutes/Bacteroidetes,по данным ряда авторов, характерна для различных жвачных (28, 29). Тем не менее в рубце североамериканского лося Alces alces показано присутствие лишь 33 % бактерий филума Firmicutes (29).
Сообщалось, что у северных оленей представленность бактерий этих таксонов варьирует в зависимости от местообитания. В частности, у подвида R. tarandus tarandus из материковой части Норвегии представленность бактерий филума Firmicutes (71 %) была выше, чем у особей R. tarandus platyrhynchus (55 %), обитающих на архипелаге Шпицберген (1). Анализ микробиоты рубца у исследованных нами особей R. tarandus показал, что наименьшая процентная доля представителей филума Bacteroidetes присутствовала у животных из Ненецкого АО, а наибольшая — у северных оленей из Ямало-Ненецкого АО (рис. 3). Общая доля бактерий филума Firmicutes достоверно не различалась у взрослых особей из разных регионов, тогда как у молодняка из Мурманской области численность этих микроорганизмов была достоверно ниже (p < 0,05), чем у особей из Ненецкого и Ямало-Ненецкого АО.
Данные о представленности в рубце северных оленей бактерий, взаимосвязанных с процессами ферментации растительной клетчатки, согласуются с результатами оценки питательности растений с пастбищ. Состав и питательность многокомпонентных проб, повторяющих состав усредненного летнего пастбищного рациона из трех регионов Арктической зоны России, различались. В целом, корма северных оленей характеризовались высоким содержанием сырой клетчатки и низким — других питательных веществ. По составу микробиома рубца северные олени из Мурманской области имели наименьшее сходство с особями из других регионов, что, вероятно, обусловлено особенностями пастбищного рациона животных в этом регионе — составом растительности и меньшими показателями питательности (см. табл. 1).
Сообщалось, что пропорция филумов Firmicutes и Bacteroidetes в рубце жвачных зависит от типа питания. Так, представители филума Bacteroidetes в основном связаны с наличием в рационе животных легко сбраживаемых углеводов (таких как крахмал) и белков, тогда как ряд представителей филума Firmicutes (бактерии родов Ruminococcus, Butirivibrio, Clostridium и др.) — с ферментацией растительной целлюлозы (7, 10, 30). Различие ферментативной активности этих микроорганизмов может оказывать влияние и на метаболизм хозяина через продукцию ЛЖК в результате ферментации растительных полисахаридов.
Интересно, что у взрослых особей по сравнению с молодыми отмечалось более широкое таксономическое разнообразие бактерий, связанных с ферментацией клетчатки, в том числе семейств Eubacteriaceae, Clostridiaceae, Lachnospiraceae филума Firmicutes (p ≤ 0,05), что свидетельствует о повышении способности микробного сообщества ферментировать растительные полисахариды.
Мы обнаружили статистически значимую корреляцию между составом микроорганизмов рубца R. tarandus, участвующих в ферментации растительных полисахаридов, и показателями питательности рационов. В частности, количество клетчатки было достоверно отрицательно связано с представленностью в рубце животных семейств Bacteroidaceae (r = -0,75, p < 0,05), Rivulariaceae (r = -0,88, p < 0,05), Veillonellaceae (r = -0,75, p < 0,05) и достоверно положительно — с присутствием семейств Lachnospiraceae (r = 0,89, p < 0,05), Clostridiaceae (r = 0,51, p < 0,05), Nostocaceae (r = 0,52, p < 0,05), Eubacteriaceae (r = 0,46, p < 0,05), Prevotellaceae (r = 0,56, p < 0,05).
Представленность некоторых целлюлозолитических микроорганизмов (например, семейств Clostridiaceae, Lachnospiraceae, Eubacteriaceae) была положительно взаимосвязана (p < 0,05) с долей бактерий из семейств Nostocaceae, Enterococcaceae, Lactobacillaceae и отрицательно (p < 0,05) — с представителями семейств Prevotellaceae, Bacteroidaceae, Veillonellaceae, Streptococcaceae. Интересно, что у исследованных нами северных оленей наблюдалась обратная закономерность между содержанием в рубце утилизирующих кислоты бактерий семейств Veillonellaceae и целлюлозолитических микроорганизмов, таких как Lachnospiraceae (r = -0,60, p < 0,05), Clostridiaceae (r = -0,55, p < 0,05), Eubacteriaceae (r = -0,60, p < 0,05). Бактерии, утилизирующие кислоты, относятся к физиологически важной для жвачных группе микроорганизмов, поскольку они позволяют поддерживать в рубце необходимую кислотность благодаря способности метаболизировать образуемые в результате сбраживания моносахаров, олиго- и полисахаридов кислоты (включая уксусную, пропионовую, масляную, молочную) (31).
Выявленные нами закономерности подтверждают мнение о том, что в процессе эволюции в рубце животных сформировались определенные связи между микроорганизмами и позволяют уточнить взаимосвязь между присутствием ряда микроорганизмов в рубце северных оленей.
Значимой взаимосвязи между содержанием условно-патогенных микроорганизмов из филумов Fusobacteria, Tenericutes (семейство Mycoplasmataceae), Proteobacteria (семейства Enterobacteriaceae, Campylobacteraceae) и других микроорганизмов мы не обнаружили. Однако для углубленного изучения этой проблемы необходимы дополнительные исследований. Прямой закономерности, характеризующей возрастные изменения в содержании условно-патогенных и патогенных бактерий в рубце у R. tarandus, мы также не обнаружили. Вероятно, выявленные при этом различия были связаны с другими факторами — особенностями пастбищного рациона и эпизоотической ситуацией в стаде.
Таким образом, выполненные нами исследования показали, что у особей северного оленя (Rangifer tarandus) ненецкой породы старше 2 лет из Мурманской области и Ямало-Ненецкого АО (но не Ненецкого АО) численность бактерий достоверно увеличивается (p < 0,05) по сравнению с показателями у телят. Независимо от региона в содержимом рубца доминирует филум Firmicutes,менее представлены бактерии филумов Bacteroidetes, Proteobacteria, Actinobacteria,доля других выявленных таксонов (Tenericutes, Fusobacteria, Cyanobacteria) минорна. Выявлены значимые изменения для ряда таксонов микроорганизмов в связи с показателями питательности пастбищного рациона в зависимости от региона. Установлена статистически значимая связь между содержанием клетчатки в пастбищных кормах и численностью членов семейств Veillonellaceae (r = -0,75), Nostocaceae (r = 0,52), Rivulariaceae (r = -0,88). Для условно-патогенных микроорганизмов из филумов Fusobacteria, Tenericutes (семейство Mycoplasmataceae) и Proteobacteria (семейства Enterobacteriaceae, Campylobacteraceae) подобных закономерностей не отмечено. В целом полученные результаты свидетельствуют о том, что условия обитания — главный фактор, оказывающий влияние на микробное сообщество рубца северных оленей из различных регионов Российской Арктики. Вероятнее всего, это связано с особенностями кормовых рационов животных. Другие факторы (пол, возраст) оказывают меньшее влияние, хотя и вносят определенный вклад в соотношение микроорганизмов в рубце. Результаты, полученные с использованием метагеномных методов анализа (благодаря их высокой разрешающей способности) позволяют открыть новые закономерности и уточнить аспекты взаимодействия и совместного обитания симбионтов в сложной экосистеме рубца северных оленей R. tarandus.
1. Усредненный состав и питательность летнего пастбищного рациона северных оленей (Rangifer tarandus) ненецкой породы в трех зонах Арктической России в летне-осенний период (M±SEM, 2017 год) |
|||
Показатель |
Ямало-Ненецкий АО |
Мурманская |
Ненецкий АО |
Показатели питательности |
|||
Растворимые углеводы (сахара), г/кг |
20,80±0,80 |
13,85±0,52 |
66,86±3,50 |
Массовая доля сухого вещества, % |
76,74±1,60 |
73,28±1,73 |
82,04±1,46 |
Сырой жир, г/кг |
16,90±0,61 |
12,39±0,84 |
15,46±0,54 |
Сырой протеин, г/кг |
95,90±4,70 |
54,96±3,10 |
64,03±3,50 |
Сырая зола, г/кг |
36,40±1,10 |
24,99±2,20 |
23,95±1,80 |
Сырая клетчатка, г/кг |
142,80±7,30 |
134,62±6,50 |
160,55±8,60 |
Компоненты рациона, % |
|||
Лишайники рода Кладония (Cladonia) |
5 |
10 |
10 |
Лишайники рода Нефрома (Nephroma) |
5 |
− |
− |
Береза обыкновенная (Betula pendula) |
5 |
20 |
20 |
Ива северная (Sаlix borеalis) |
5 |
20 |
15 |
Ива полярная (Salix polaris) |
15 |
− |
− |
Голубика обыкновенная (Vaccinium uliginosum) |
10 |
− |
5 |
Береза карликовая (Bеtula nаna) |
25 |
20 |
20 |
Смесь многолетних трав |
30 |
30 |
30 |
Примечание. АО — автономный округ. Прочерки означают отсутствие компонента в рационе. |
|||
2. Количество микроорганизмов в сообществе рубца у особей северных оленей (Rangifer tarandus) ненецкой породы в возрасте до 2 лет (I) и старше 2 лет (II), обитающих в различных регионах Российской Арктики (M±SEM, 2017 год) |
|||
Регион, возраст |
Общее количество бактерий |
Общее количество архей |
Количество грибов класса Neocallimastigales |
Ямало-Ненецкий АО: |
|
|
|
I |
1,56×109±5,38×107 d |
1,20×108±3,94×106 d |
2,81×106±8,24×104 b |
II |
2,58×109±1,84×106 c |
1,42×108±6,72×106 d |
4,53×106±1,95×105 a |
Мурманская область: |
|
|
|
I |
8,10×108±4,01×103 e |
1,03×109±5,25×104b |
2,07×105±8,09×102 c |
II |
1,75×109±2,87×108 c, d |
7,79×108±2,87×106c |
2,07×105±8,09×102 c |
Ненецкий АО: |
|
|
|
I |
6,37×109±4,72×108 a |
8,74×107±6,02×106 e |
3,24×105±9,50×103 c |
II |
4,41×109±2,12×107 b |
2,15×109±1,56×108 a |
3,39×105±5,10×104 c |
Примечание. АО — автономный округ. |
|||
Рис. 1. Регионы отбора образцов содержимого рубца северных оленей (Rangifer tarandus) ненецкой породы: 1 — Ямало-Ненецкий автономный округ, 2 — Мурманская область, 3 — Ненецкий АО (2017 год).
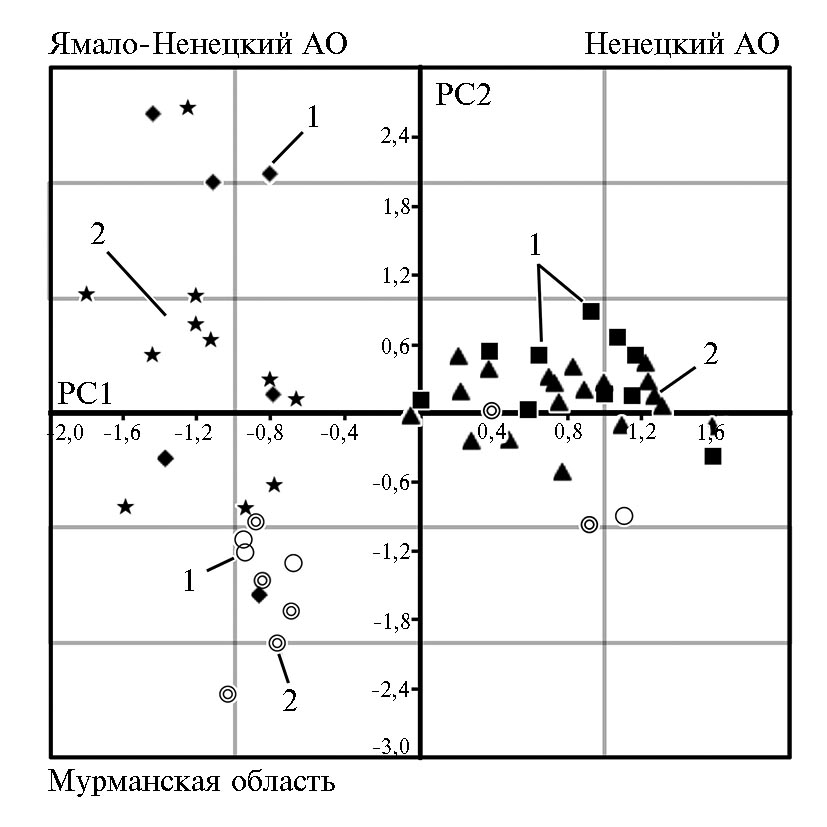
Рис. 2. Сравнительный анализ бактериального сообщества рубца северных оленей (Rangifer tarandus) ненецкой породы, обитающих в различных регионах Российской Арктики, с применением метода главных компонент (Principal Component Analysis) по результатам T-RFLP-анализа: 1 — молодняк (до 2 лет), 2 — взрослые особи (старше 2 лет) (2017 год).
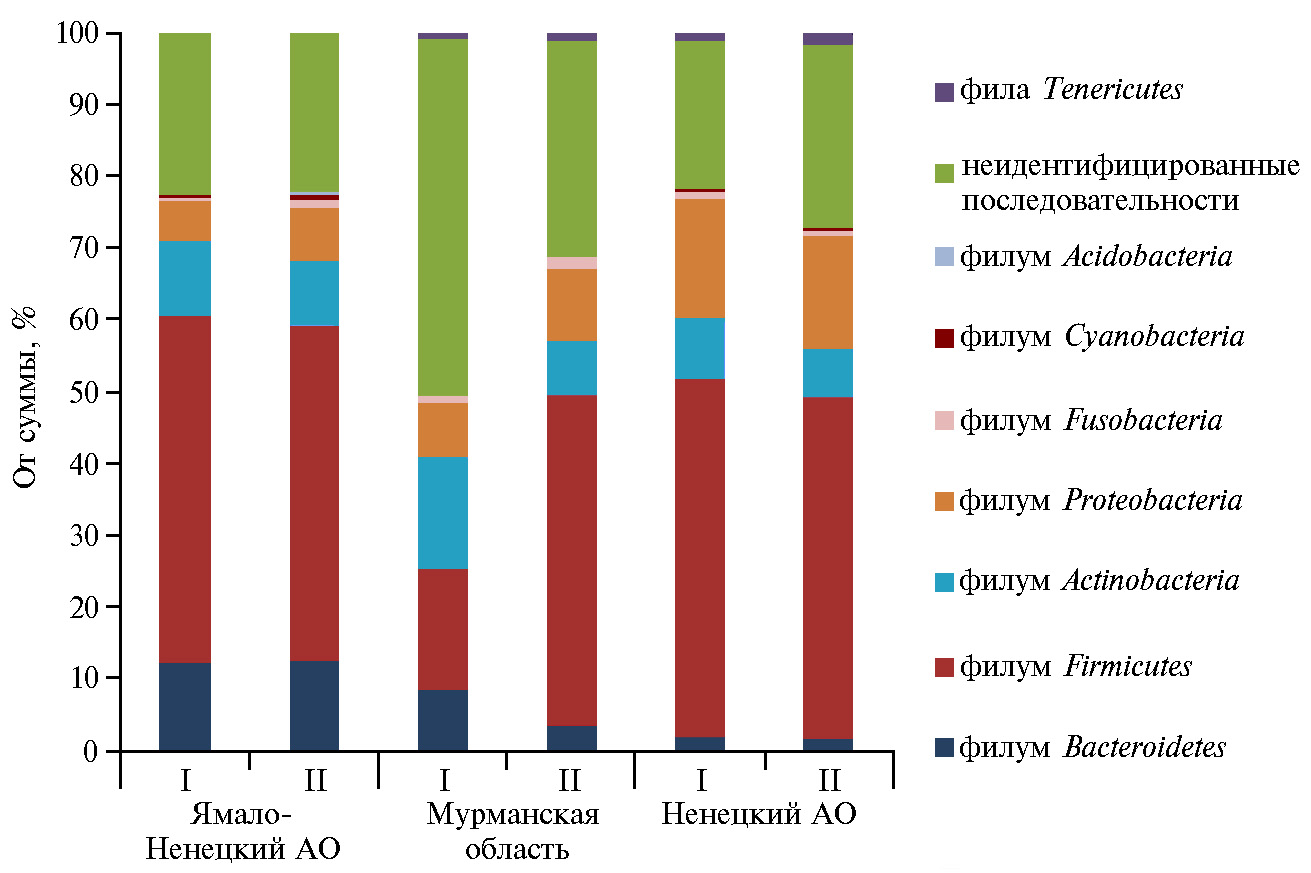
Рис. 3. Соотношение бактериальных филумовв рубце северных оленей (Rangifer tarandus) ненецкой породы, обитающих в различных регионах Российской Арктики (2017 год): I — в возрасте до 2 лет, II — старше 2 лет.
ЛИТЕРАТУРА
- Sundset M.A., Præsteng K.E., Cann I.K.O., Mathiesen S.D., Mackie R.I. Novel rumen bacterial diversity in two geographically separated sub-species of reindeer. Microb. Ecol., 2007, 54(3): 424-438 (doi: 10.1007/s00248-007-9254-x).
- Malmuthuge N., Guan L.L. Gut microbiome and omics: a new definition to ruminant production and health. Animal Frontiers, 2016, 6(2): 8-12 (doi: 10.2527/af.2016-0017).
- Matthews C., Crispie F., Lewis E., Reid M., O’Toole P.W., Cotter P.D. The rumen microbiome: a crucial consideration when optimising milk and meat production and nitrogen utilisation efficiency. Gut Microbes, 2019, 10(2): 115-132 (doi: 10.1080/19490976.2018.1505176).
- Hackmann T.J., Spain J.N. Invited review: ruminant ecology and evolution: perspectives useful to ruminant livestock research and production. Journal of Dairy Science, 2010, 93(4): 1320-1334 (doi: 10.3168/jds.2009-2071).
- Morgavi D.P., Kelly W.J., Janssen P.H., Attwood G.T. Rumen microbial (meta)genomics and its application to ruminant production. Animal, 2013, 7(1): 184-201 (doi: 10.1017/S1751731112000419).
- The ruminant animal: Digestive physiology and nutrition /D.C. Church (ed.). Prentice Hall, New Jersey, 1993.
- Hungate R.E. The rumen and its microbes. Academic Press, NY, 1966.
- Grilli D.J., Fliegerová K., Kopečný J., Lama S.P., Egea V., Sohaefer N., Pereyra C., Ruiz M.S., Sosa M.A., Arenas G.N., Mrázek J. Analysis of the rumen bacterial diversity of goats during shift from forage to concentrate diet. Anaerobe, 2016, 42: 17-26 (doi: 10.1016/j.anaerobe.2016.07.002).
- Mathiesen S.D., Mackie R.I., Aschfalk A., Ringø E., Sundset M.A. Microbial ecology of the gastrointestinal tract in reindeer — changes through season. In: Microbial ecology of the growing animal; Biology of the growing animals. V. 3 /W. Holzapfel, P. Naughton (eds.). Elsevier Press, Oxford: 73-100.
- Henderson G., Cox F., Ganesh S., Jonker A., Young W., Global Rumen Census Collaborators, Janssen P.H. Rumen microbial community composition varies with diet and host, but a core microbiome is found across a wide geographical range. Sci. Rep., 2015, 5: 14567 (doi: 10.1038/srep14567).
- Aagnes T.H., Sørmo W., Mathiesen S.D. Ruminal microbial digestion in free-living, in captive lichen-fed, and in starved reindeer (Rangifer tarandus tarandus) in winter. Appl. Environ. Microbiol., 1995, 61(2): 583-591.
- de la Fuente G., Belanche A., Girwood S.E., Pinloche E., Wilkinson T., Newbold C.J. Pros and cons of Ion-Torrent next generation sequencing versus Terminal Restriction Fragment Length Polymorphism T-RFLP for studying the rumen bacterial community. PLoS ONE, 2014, 9(7): e101435 (doi: 10.1371/journal.pone.0101435).
- Wang L., Xu Q., Kong F., Yang Y., Wu D., Mishra S., Li Y. Exploring the goat rumen microbiome from seven days to two years. PLoS ONE, 2016, 11(5): e0154354 (doi: 10.1371/journal.pone.0154354).
- Peng S., Yin J., Liu X., Jia B., Chang Z., Lu H., Jiang N., Chen Q. First insights into the microbial diversity in the omasum and reticulum of bovine using Illumina sequencing. J. Appl. Genetics, 2015, 56(3): 393-401 (doi: 10.1007/s13353-014-0258-1).
- Elekwachi C.O., Wang Z., Wu X., Rabee A., Forster R.J. Total rRNA-Seq analysis gives insight into bacterial, fungal, protozoal and archaeal communities in the rumen using an optimized RNA isolation method. Front. Microbiol., 2017, 8: 1814 (doi: 10.3389/fmicb.2017.01814).
- Gruninger R.J., Sensen C.W., McAllister T.A., Forster R.J. Diversity of rumen bacteria in Canadian cervids. PLoS ONE, 2014, 9(2): e89682 (doi: 10.1371/journal.pone.0089682).
- Zielińska S., Kidawa D., Stempniewicz L., Łoś M., Łoś J.M. New insights into the microbiota of the Svalbard Reindeer Rangifer tarandus platyrhynchus. Front. Microbiol., 2016, 7: 170 (doi: 10.3389/fmicb.2016.00170).
- Лаптев Г.Ю., Новикова Н.И., Ильина Л.А., Йылдырым Е.А., Нагорнова К.В., Думова В.А., Солдатова В.В., Большаков В.Н., Горфункель Е.П., Дубровина Е.Г., Соколова О.Н., Никонов И.Н., Лебедев А.А. Нормы содержания микрофлоры в рубце крупного рогатого скота. СПб, 2016.
- Маниатис Т., Фрич Э., Сэмбрук Дж. Молекулярное клонирование. М., 1984.
- Забродин В.А., Лайшев К.А., Дубовик И.К. Развитие северного оленеводства в рамках осуществления Арктических интересов России. Известия Санкт-Петербургского государственного аграрного университета, 2015, 40: 108-112.
- Olsen M.A., Aagnes T.H., Mathiesen S.D. The effect of timothy silage on the bacterial population in rumen fluid of reindeer (Rangifer tarandus tarandus) from natural summer and winter pasture. FEMS Microbiol Ecol., 1997, 24(2): 127-136 (doi: 10.1111/j.1574-6941.1997.tb00429.x).
- Orpin C.G., Mathiesen S.D., Greenwood Y., Blix A.S. Seasonal changes in the ruminal microflora of the high-arctic Svalbard reindeer (Rangifer tarandus platyrhynchus). Appl. Environ. Microbiol., 1985, 50(1): 144-151.
- Wang T.Y., Chen H.L., Lu M.J., Chen Y.C., Sung H.M., Mao C.T., Cho H.Y., Ke H.M., Hwa T.Y., Ruan S.K., Hung K.Y., Chen C.K., Li J.Y., Wu Y.C., Chen Y.H., Chou S.P., Tsai Y.W., Chu T.C., Shih C.A., Li W.H., Shih M.C. Functional characterization of cellulases identified from the cow rumen fungus Neocallimastix patriciarum W5 by transcriptomic and secretomic analyses. Biotechnol. Biofuels, 2011, 4: 24 (doi: 10.1186/1754-6834-4-24).
- Fonty G., Joblin K.N. Rumen anaerobic fungi: their role and interactions with other rumen microorganisms in relation to fiber digestion. In: Physiological aspects of digestion and metabolism in ruminants /T. Tsuda, Y. Sasaki, R. Kawashima (eds.). Academic Press, Toronto, ON, 1990: 665-680 (doi: 10.1016/B978-0-12-702290-1.50033-3).
- Sundset M.A., Edwards J.E., Cheng Y.F., Senosiain R.S., Fraile M.N., Northwood K.S., Præsteng K.E., Glad T., Mathiesen S.D., Wright A.D.G. Molecular diversity of the rumen microbiome of Norwegian reindeer on natural summer pasture. Microb. Ecol., 2009, 57(2): 335-348 (doi: 10.1007/s00248-008-9414-7).
- Schofield B.J., Lachner N., Le O.T., McNeill D.M., Dart P., Ouwerkerk D., Hugenholtz P., Klieve A.V. Beneficial changes in rumen bacterial community profile in sheep and dairy calves as a result of feeding the probiotic Bacillus amyloliquefaciens H57. J. Appl. Microbiol., 2018, 124(3): 855-866 (doi: 10.1111/jam.13688).
- Weimer P.J. Redundancy, resilience, and host specificity of the ruminal microbiota: implications for engineering improved ruminal fermentations. Front. Microbiol., 2015, 6: 296 (doi: 10.3389/fmicb.2015.00296).
- Samsudin A.A., Evans P.N., Wright A.G., Jassim R.A. Molecular diversity of the foregut bacteria community in the dromedary camel (Camelus dromedarius). Environmental Microbiology, 2011, 13(11): 3024-3035 (doi: 10.1111/j.1462-2920.2011.02579.x).
- Ishaq S.L, Wright A.D. Insight into the bacterial gut microbiome of the North American moose (Alces alces). BMC Microbiol., 2012, 12: 212 (doi: 10.1186/1471-2180-12-212).
- McCann J.C., Elolimy A.A., Loor J.J. Rumen microbiome, probiotics, and fermentation additives. Veterinary Clinics of North America: Food Animal Practice, 2017, 33(3): 539-553 (doi: 10.1016/j.cvfa.2017.06.009).
- Nocek J.E. Bovine acidosis: implications on laminitis. Journal of Dairy Science, 1997, 80: 1005-1028 (doi: 10.3168/jds.S0022-0302(97)76026-0).