doi: 10.15389/agrobiology.2019.5.990rus
УДК 635.21:632.4:577.21
Работа поддержана грантом РНФ № 18-16-00073. Отбор образцов и их описание выполнены при финансовой поддержке Министерства образования и науки Российской Федерации (Соглашение № 14.607.21.0178, RFMEFI60717X0178).
АНАЛИЗ МИКОБИОТЫ В ПОРАЖЕННЫХ ЛИСТЬЯХ КАРТОФЕЛЯ (Solanum tuberosum L.)
С ИСПОЛЬЗОВАНИЕМ МЕТАГЕНОМНЫХ ПОДХОДОВ
А.А. КИЧКО1, Т.С. АКСЕНОВА1, В.М. ШАПКИН1, А.О. ЗВЕРЕВ1, А.В. ХЮТТИ2, Е.Е. АНДРОНОВ1, 3, 4
Проблема поражения картофеля заболеваниями, вызванными грибами и грибоподобными организмами, актуальна для всех регионов мира, где возделывается эта культура, поскольку именно они наносят растениям самый значительный ущерб (A. Bernreiter, 2017). Традиционные подходы для идентификации патогенов картофеля нацелены на детекцию конкретного патогена и не учитывают ни другие (часто неизвестные) патогены, ни полезную микробиоту филлосферного сообщества, нарушения которой также могут стать одной из причин заболеваний. В нашей работе впервые исследована патогенная и непатогенная грибная и грибоподобная микрофлора с применением метагеномного подхода. Впервые показано, что реальная картина имеет комплексный и не вполне однозначный характер (например, основной патоген может быть минорным компонентом всего сообщества). Таксономический анализ сообществ, полученных по обоим вариантам праймеров, выявил в образце с поражением типа альтернариоз патогенный гриб рода Alternaria. Использование праймеров для амплификации региона ITS1 позволило обнаружить наличие оомицета Phytophthora и диагностировать присутствие фитофторы при обоих типах поражения. Целью нашей работы была максимально полная идентификация видового состава грибов и грибоподобных организмов, участвующих в фитопатогенезе листьев картофеля, с применением высокопроизводительного секвенирования и двух вариантов универсальных праймеров. Для анализа сообществ из образцов листьев картофеля сорта Никулинский, пораженных заболеваниями альтернариозного и фитофторозного типов, выделили ДНК, которую в дальнейшем использовали для создания ампликонных библиотек фрагментов ITS1 и ITS2 и высокопроизводительного секвенирования на приборе Illumina MiSeq («Illumina, Inc.», США). В процессе биоинформатической обработки данных при помощи программного обеспечения Illumina и программного пакета PIPITS (H.S. Gweon с соавт., 2015), получили 187 ОТЕ (операционные таксономические единицы) и 113 филотипов для библиотеки ITS1, 249 ОТЕ и 127 филотипов для ITS2. Последующее аннотирование ОТЕ и таксономический анализ сообществ проводились в программе QIIME (J.G. Caporaso с соавт., 2010), коэффициенты разнообразия внутри сообщества рассчитывались при помощи программного пакета PAST (Ø. Hammer с соавт., 2001). Сравнительная характеристика сообществ грибов и грибоподобных организмов, полученных для обоих видов поражения с использованием различных универсальных праймеров для ITS1 и ITS2, показала, что из выбранных праймеров только первая пара подходит для детекции фитофторы и дает более выравненную картину сообщества. Инструментов автоматического аннотирования результатов секвенирования оказалось недостаточно для объективной идентификации альтернарии в образцах, в связи с чем мы использовали методы ручного поиска по последовательностям в программе BLASTn (S.F. Altschul с соавт., 1990). Поскольку пара праймеров для ITS2 не учитывала присутствие фитофторы в образцах, дальнейший сравнительный анализ сообществ, характерных для двух типов поражения, проводился с использованием данных только по библиотеке ITS1. Результаты таксономического анализа показали, что на пораженных участках при обоих типах патогенеза формировалось довольно богатое сообщество грибов и грибоподобных организмов, причем в случае с поражением по типу фитофтороза доля соответствующего патогена в сообществе составляет около 30 %, а в варианте с альтернариозом — всего 2,07 %, тогда как значительная часть (около 15 %) приходится на фитофтору, что не исключает так называемого вторичного поражения. Таким образом, в сообществах, сформировавшихся на участках, пораженных заболеванием, доля патогенов составляет не более 30 %, что свидетельствует о выраженной динамике таксономического состава в зоне поражения. Очевидно, что методы высокопроизводительного секвенирования имеют весьма высокий потенциал в фундаментальных и прикладных исследованиях заболеваний растений, имеющих микробиологическую природу.
Ключевые слова: грибы, грибоподобные организмы, патогены, картофель, фитофтороз, альтернариоз, высокопроизводительное секвенирование.
Картофель — важная сельскохозяйственная культура, активно возделываемая во многих регионах мира, поэтому проблема потери ее урожая вследствие поражения различными заболеваниями актуальна как в России, так и за рубежом. Болезни растений могут быть вызваны бактериями, вирусами, грибами и грибоподобными организмами. Наиболее тяжелым течением характеризуются микозы и поражения, возбудителями которых становятся представители грибоподобной микрофлоры, в частности оомицеты (1). Вызываемые ими патологии выражаются в появлении гнилей, пятнистостей, налетов, заплесневении плодов и семян (2).
Лист растения — это экологическая ниша, заселенная сообществом микроорганизмов, в том числе грибами и грибоподобными организмами филлопланы, эндофитами и фитопатогенами. Нарушение равновесия в таком сообществе в результате изменения погодных условий, добавления в почву химических веществ или по другим причинам может привести к доминированию патогена и развитию заболевания (3, 4). К полезным обитателям филлосферы, которые повышают устойчивость к вредоносными представителям микрофлоры, относят диазотрофов, антагонистов и бактерий, обеспечивающих рост растения (3). Патогенные организмы, населяющие филлоплану, представляют потенциальную угрозу для хозяина и могут относиться как к биотрофам, так и к некротрофам (3, 4). Сапрофитные организмы, поражающие ослабленные растения, например грибы рода Fusarium, могут развиваться как вторичная инфекция, маскируя первичный патоген.
Одно из наиболее опасных заболеваний картофеля в регионах с влажным и умеренным климатом — фитофтороз, вызываемый оомицетом Phytophthora infestans (Mont.) de Bary (5). Этот патоген поражает листовую поверхность, снижая ассимиляционную активность растения в период формирования клубней, и провоцирует их дальнейшее загнивание в процессе хранения (6). В конце 1840-х годов в Европе, особенно в Ирландии, распространение фитофтороза привело к потере урожая картофеля, что вызвало «Великий ирландский голод» (Great Irish famine, Irish potato famine) (7). В России и Европе, ущерб от поражения фитофторой (в зависимости от почвенно-климатических условий конкретного региона) может составлять от 10-12 до 50 % всего урожая картофеля (8, 9). Несовершенные грибы рода Alternaria вызывают альтернариоз картофеля, который представляет серьезную угрозу для регионов с более засушливым климатом, характеризующимся наличием кратковременных осадков и обильных ночных рос (4, 5). Альтернария также поражает поверхность листьев, снижая ассимиляционную деятельность растения и приводя к потерям урожая до 40 %. Кроме того, в картофеле снижается содержание крахмала, что повышает долю нетоварных клубней (5, 6, 10), а также происходит накопление микотоксинов и аллергенов (2).
Сапрофитные грибы рода Fusarium вызывают фузариозное увядание картофеля. А.Н. Смирнов с соавт. (11) показали, что патогенный микологический комплекс Fusarium—Alternaria становится одной из причин полегания картофеля в различных регионах России и может возникать как первичное и вторичное поражение после ризоктониоза, фитофтороза или бактериоза. Если ранее фузариоз и альтернариоз были более распространены в южных регионах, то сейчас эти заболевания стали характерны и для европейской части страны (4, 5, 11).
Альтернариоз и фитофтороз распространены во всех картофелеводческих регионах, проблемой борьбы с ними занимаются ученые в России и за рубежом. В частности, известны исследования филлопланы (3, 12), эндофитных (13, 14) и почвенных грибных сообществ (15, 16), а также непосредственно участков растений, пораженных микозами (17, 18); в результате были обнаружены возбудители фитофтороза, альтернариоза и других микологических заболеваний. Активно изучается воздействие на патогены различных агротехнических методов (15) и фунгицидов (8, 10, 19), которые могут использоваться в комбинации с другими средствами борьбы (20), например совместно с культуральной жидкостью бактерии Klebsiella planticola (21). Изучена устойчивости ряда сортов картофеля к возбудителям альтернариоза и фитофтороза (9, 22).
Для эффективной борьбы с патогенами важно правильно их идентифицировать (23). Известно, что возбудители альтернариоза — комплекс альтернариевых грибов, среди которых выделяют мелкоспоровые и крупноспоровые формы (10), различающиеся по экологическим свойствам, специфичности в отношении растения-хозяина, патогенности, токсикогенности, чувствительности к фунгицидам и географическому распространению (2, 4). Традиционно для видовой идентификации используют цитологические методы, микроскопию и выделение чистых культур (1, 24), определение по специфической симптоматике на больном растении (1, 25), метод влажных камер для стимулирования образования инфекционных структур, проверку патогенности на восприимчивом растении. Также применяют спектроскопию (25-27), масс-спектрометрию (1), диагностику с помощью биосенсоров (25), молекулярно-биологические методы, основанные на ПЦР (28-30), клонирование с последующим секвенированием по Сэнгеру с универсальными праймерами (17, 31), иммуноферментный анализ (32).
Однако, как отмечалось выше, важен анализ сообщества в целом, в том числе детекция нарушения равновесия в сообществах филлопланы. Эта задача выходит за рамки традиционных методов, ориентированных на идентификацию конкретного патогена и не учитывающих наличие других патогенных организмов и состояние нормальной непатогенной микрофлоры, функция которой в защите растения зачастую недооценивается. Высокопроизводительное секвенирование (3, 16, 33) дает возможность оценить не только известные и неизвестные патогены, для которых на сегодняшний день вообще не созданы диагностикумы, но и нормальную флору. Среди праймеров, разработанных для создания грибных ампликонных библиотек (23, 33, 34), мы выбрали две пары универсальных праймеров для регионов ITS1 и ITS2 (35, 36), позволяющих проводить таксономическую идентификацию большинства групп грибов и грибоподобных организмов до рода и даже вида (16, 37, 38).
В настоящей работе на растениях картофеля, пораженных заболеваниями типа альтернариоза и фитофтороза, впервые была исследована патогенная и непатогенная грибная и грибоподобная микрофлора с применением метагеномного подхода. Проведена оценка наличия или отсутствия основного патогена, а также таксономической структуры всего сообщества в зоне поражения. Впервые показано, что реальная картина имеет комплексный и не вполне однозначный характер (например, основной патоген может быть минорным компонентом всего сообщества). Таксономический анализ сообществ, полученных по обоим вариантам праймеров, выявил в образце с поражением типа альтернариоз патогенный гриб рода Alternaria. Использование праймеров для региона ITS1 позволило обнаружить оомицет Phytophthora и диагностировать присутствие фитофторы при обоих типах поражения.
Целью нашего исследования была максимально полная идентификация видового состава грибов и грибоподобных организмов, участвующих в фитопатогенезе листьев картофеля, с применением высокопроизводительного секвенирования и двух вариантов универсальных праймеров.
Методика. Образцы листьев картофеля (Solanum tuberosum L.) сорта Никулинский с видимым поражением фитопатогенами были собраны на опытном поле Всероссийского института генетических ресурсов растений им. Н.И. Вавилова (г. Санкт-Петербург—Пушкин, 59°42´37,78²С; 30°25´41,26²В) в 2017 году. Два типа поражения идентифицировали по морфологическим признакам.
ДНК выделяли непосредственно из пораженного участка листа с использованием набора реактивов AxyPrep Multisource Genomic DNA Miniprep Kit («Axygen», США) согласно инструкции производителя.
В каждом образце определяли таксономический состав сообщества грибов и грибоподобных организмов на основании анализа ампликонных библиотек межгенных транскрибирующихся спейсеров рибосомальных оперонов (ITS1 и ITS2). Целевой фрагмент амплифицировали методом ПЦР (T100 Thermal Cycler, «BIO-RAD Laboratories, Inc.», США) с использованием пар праймеров:для участка ITS1 — ITS1_30F-GTCCCTGCCC-TTTGTACACA/ITS1_217R-TTTCGCTGCGTTCTTCATCG (35), для ITS2—ITS3-GCATCGATGAAGAACGCAGC/ITS4-TCCTCCGCTTATTGATATGC (36) с последующим добавлением служебных последовательностей по протоколу «Illumina, Inc.» (США), содержащих линкеры и баркоды. ПЦР проводили с использованием полимеразы Phusion Hot Start II High-Fidelity polymerase («Thermo Fisher Scientific», США) по протоколу производителя.
ПЦР-продукты очищали по методике, рекомендованной «Illumina, Inc.», с использованием AM Pure XP («Beckman Coulter», США). Дальнейшую подготовку библиотек осуществляли в соответствии с инструкцией производителя («MiSeq Reagent Kit Preparation Guide», США). Библиотеки секвенировали на приборе Illumina MiSeq c использованием набора реактивов MiSeq® Reagent Kit v3 (600 cycle) с двусторонним чтением (2×300 н.) («Illumina, Inc.», США).
Выявленные последовательности обрабатывали с помощью программного обеспечения Illumina («Illumina, Inc.»), пакета QIIME (39) и программного пакета PIPITS (40).
На основе полученных данных выполняли таксономический анализ всех образцов и сравнивали варианты с праймерами ITS1 и ITS2. При этом оценивали представленность различных таксонов, а также богатство сообществ. Определяли показатель, отражающий число таксонов (richness — ожидаемое число филотипов), коэффициенты Симпсона (eveness — выравненность распределения по филотипам) и Шеннона, рассчитанные при помощи программы PAST (41).
Результаты. Поверхностные поражения листьев картофеля на основании морфологических признаков были отнесены к заболеваниям альтернариозного и фитофторозного типов (рис. 1).
Для видовой идентификации возбудителей, находящихся в зоне поражения, методом высокопроизводительного секвенирования мы использовали хорошо известные праймеры для региона ITS2 (36), а в качестве дополнительных — праймеры для ITS1 (35), проявляющие широкую специфичность (рис. 2). Оба типа поражений оценивали по составу и разнообразию грибного и грибоподобного сообщества, полученного при анализе данных по каждому из регионов.
Число прочтений (последовательностей) для образцов, полученных с парой праймеров для ITS1, в вариантах с альтернариозом и фитофторозом составило соответственно 55000 и 54000, c ITS2 — 36000 и 21000. Процессинг библиотек ITS1 и ITS2 проводился раздельно, поскольку выделение OTE (операционные таксономические единицы) может выполняться только для библиотек гомологов. При биоинформатической обработке результатов для ITS1 получили 187 ОТЕ и 113 филотипов, для ITS2 — 249 ОТЕ и 127 филотипов.
После автоматического аннотирования ОТЕ (банк данных UNITE, https://unite.ut.ee/, программный пакет PIPITS) довольно большая группа ОТЕ получила таксономический диагноз только на уровне царства. При этом в библиотеках для обоих вариантов праймеров не обнаружили значимого количества гриба Alternaria (ITS1 — 0,01 и 0,08 %, ITS2 — 0,07 и 0,04 % соответственно для вариантов с альтернариозом и фитофторозом), кроме того, в библиотеке ITS2 не детектировалась фито-фтора. По этой причине неаннотированные ОТЕ проверили вруч-ную на таксономическую принадлежность при помощи программы BLASTn (42). Проблема вынесения неверного таксономического диагноза ОТЕ упоминается в работе B. Halwachs с соавт. (43), которые также рекомендуют ручную коррекцию данных при помощи программ на основе алгоритма BLAST для уточнения результатов автоматической аннотации. Среди причин, по которым нужные ОТЕ оказываются неаннотированными, могут быть недостаточное число референсных последовательностей в базах данных или неодинаковое таксономическое разрешение ITS для различных групп грибов на уровне рода и вида (37).
Сообщества грибов и грибоподобных организмов в целом оценивали по автоматическому таксономическому диагнозу. При сравнении результатов, полученных по двум регионам, были обнаружены различия в таксономическом составе как между сообществами, характерными для двух типов поражений, так и между библиотеками, полученными с использованием праймеров на ITS1 и ITS2 (табл. 1). Для дальнейшего сравнения выбрали только те таксоны, доля которых хотя бы в одной из библиотек составляла более 1 % (см. табл. 1).
Уже на уровне крупных таксонов были заметны различия в специфичности выбранных нами праймеров (рис. 3). Так, широко специфичные праймеры на ITS1 захватывали представителей филы Oomycota, к которой относится фитофтора, но при этом значительное количество прочтений приходилось на растительные организмы. Это хорошо согласуется со спецификой праймеров для ITS1, описанной L. Xu (16). В свою очередь, выбранные нами праймеры для ITS2 захватывали только представителей царства грибов, не выявляя растительных гомологов. Однако они демонстрировали более узкую специфичность по сравнению с парой для ITS1, результатом чего стало отсутствие оомицетов в библиотеках, что делает эту пару праймеров не подходящей для идентификации фитофторы в пораженных образцах.
Для оценки доли патогенов в полученных сообществах результаты автоматического аннотирования по базе UNITE и проверки последовательностей в программе BLASTn (https://blast.ncbi.nlm.nih.gov/Blast.cgi) были суммированы и сведены в общую таблицу (табл. 2). На ее основе оценивалась эффективность выбранных праймеров для выявления искомых патогенов.
Стоит отметить, что в библиотеках ITS2 наблюдалась значительная неравномерность в представленности таксонов, например заметный перевес в сторону представителей порядка Capnodiales (Ascomycota) (см. табл. 1), которые преобладали при обоих вариантах поражений, что приводило к существенному снижению параметра разнообразия полученного сообщества. Это ясно видно при сравнении значений коэффициента Симпсона, характеризующего выравненность (eveness) распределения таксонов (табл. 3). Такие же соотношения выявлены и при анализе числа таксонов, или видового богатства (richness), а также коэффициента Шеннона. Интересно, что по данным других авторов (37), исследовавших одно и то же сообщество с использованием ITS1 и ITS2, библиотеки давали довольно сходные результаты при аннотировании полученных ОТЕ. Степень сходимости результатов была выше в отношении базидиомицетов и ниже в отношении аскомицетов, в частности для них в варианте с ITS1 выделялось большее количество кластеров. Наши данные, напротив, показывают перенасыщенность библиотек ITS2 представителями филы Ascomycota за счет порядка Capnodiales.
При сравнении сообществ на уровне семейств общими для обоих вариантов праймеров оказались представители Cladosporiaceae, Didymellaceae, Phaeosphaeriaceae, Cystofilobasidiaceae, Mrakiaceae, Bulleraceae, Bulleribasidiaceae. Характерными только для варианта с ITS1 были представители Sporidio-bolaceae, Filobasidiaceae и Peronosporales, к которым относится Phytophthora infestans (см. табл. 1).
Обе пары праймеров подошли для идентификации альтернарии, однако по непонятным причинам система автоматической идентификации не атрибутировала грибы этого вида и отнесла их к группе неидентифицированных организмов. Тем не менее с использованием ручной коррекции по данным BLASTn грибы рода Alternaria были выявлены, причем составляли заметную часть сообщества.
Поскольку сопоставление библиотек ITS1 и ITS2, а точнее использованных праймеров, продемонстрировало очевидное превосходство первых, в дальнейшем мы работали только с библиотекой ITS1
Данные таксономического анализа для образца с поражением альтернариозного типа показали, что в сообществе преобладали неидентифицированные представители царства Fungi (27,38 %). Довольно многочисленными были представители родов Itersonilia (18,4 %) и Phytophthora (15,47 %). На долю растительных ITS приходилось 16,68 %. Встречались грибы родов Bullera (4,41 %), Cystofilobasidium (2,7 %), Alternaria (2,07 %) (см. табл. 3), неатрибутированные на уровне семейства представители порядка Tremellomycetes (1,51 %) и аскомицеты (1,26 %), а также грибы рода Dioszegia (1,18 %). В незначительных количествах (менее 1 % в сообществе) были обнаружены представители семейств Bulleraceae и Bulleribasidi-aceae, родов Udeniomyces и Filobasidium (см. табл. 1). При фитофторозном типе поражения доминировали две группы организмов — неидентифицированные представители царства Fungi (32,85 %) и оомицеты рода Phytophthora (29,85 %). На представителей рода Itersonilia и растительные ITS в сообществе приходилось соответственно 8,86 и 8,00 %. Далее по убыванию шли представители порядка Hypocreales (3,23 %), родов Sporobolomyces (2,64 %), Bullera (2,01 %), Udeniomyces (1,59 %), Cystofilobasidium (1,46 %) и Filobasidium (1,40 %). Также в сообществе обнаружили ряд малочисленных таксонов, доля которых не превышала 1 % (см. табл. 1).
Данные по ITS1 показали наличие фитофторы в обоих образцах в количествах 15,47 и 29,85 % от численности сообщества соответственно для альтернариозного и фитофторозного типа поражения. Представители рода Alternaria были обнаружены в обоих вариантах: их доля составила 2,1 и 0,1 % соответственно для альтернариоза и фитофтороза (см. табл. 3).
Вне зависимости от варианта поражения существенное количество прочтений приходилось на неидентифицированные грибные организмы. В структуре сообщества, полученного для образца с альтернариозом, наблюдалось присутствие двух субдоминантных таксонов — родов Itersonilia и Phytophthora. В варианте с фитофторозом род Phytophthora выступал в качестве второго доминанта. Также в обоих сообществах присутствовали таксоны, либо не обнаруженные в другом сообществе, либо обнаруженные в нем в незначительных количествах. Для альтернариозного типа поражения это были неидентифицированные представители класса Tremellomycetes и грибы семейства Phaeosphaeriaceae; для фитофторозного — неидентифицированные представители порядка Hypocreales и родов Sporobolomyces, Udeniomyces и Filobasidium.
В работах, посвященных патогенам в грибных сообществах филлосферы и клубней картофеля, грибы рода Alternaria обнаруживаются на пораженных растениях в существенных количествах, однако стоит отметить, что в основном авторы используют метод культивирования изолятов, выращенных на специальных питательных средах (12, 17, 18). Исследования эндофитной микобиоты здорового растения, основанные на этом подходе, также подтверждают значительное присутствие грибов рода Alternaria (13). В то же время подобный анализ с применением высокопроизводительного секвенирования показывает малую долю патогена в сообществе (0,35 %) (14), а в филлосфере ослабленного растения, пораженного грибом Podosphaera, — более значительную (0,35-4,6 %). Эти данные демонстрируют преимущества метагеномного подхода в исследованиях подобного рода.
Таким образом, нами выявлены серьезные различия в таксономической специфичности двух пар праймеров ITS1 и ITS2. Праймеры ITS1 позволяют обнаружить не только больше таксонов грибов и грибоподобных организмов, но и продемонстрировать более высокие уровни выравненности (eveness) в распределении последовательностей по таксонам. Однако за счет более широкой специфичности пары праймеров на ITS1 захватывают растительные последовательности. Автоматический таксономический диагноз по базам данных не всегда позволяет объективно оценить присутствие некоторых таксонов в сообществе. Это может сильно искажать результаты анализа, особенно в случаях, когда нужно диагностировать определенный процесс или присутствие конкретного патогенного организма. По этой причине комбинирование нескольких инструментов для детектирования целевого организма может положительно повлиять на результаты исследования. Сообщество изученных нами пораженных листьев картофеля сорта Никулинский оказалось весьма богато. Интересно, что в случае фитофтороза доля патогена не превышала 29,9 %, что свидетельствует о динамике таксономического состава пораженных тканей. Вероятно, на первом этапе поражения в них почти исключительно присутствует главный патоген, а впоследствии они заселяются оппортунистической микрофлорой. Возможно, именно этим объясняется низкое число альтернарий в образце, соответствующем альтернариозу. Не исключено, что в описываемом случае присутствие фитофторы также может быть вторичным поражением.
Итак, результаты таксономического анализа показали, что на пораженных участках при обоих типах патогенеза формировалось довольно богатое сообщество грибов и грибоподобных организмов, причем доля основного патогена в сообществе при поражении по типу фитофтороза составляет около 30 %, по типу альтернариоза — всего 2,07 %, тогда как значительная часть (около 15 %) приходится на фитофтору (возможно, вследствие вторичного поражения). Методы высокопроизводительного секвенирования, несомненно, весьма перспективны для идентификации растительных патогенов, однако требуют значительной методической работы как на этапе подбора праймеров, так и при анализе библиотек, когда может произойти потеря некоторых важных таксонов.
1. Таксономический диагноз основных операционных таксономических единиц (ОТЕ) грибов и грибоподобных организмов, выделенных из листьев картофеля (Solanum tuberosum L.) сорта Никулинский со следами поражения фитофторозного и альтернариозного типов (опытные поля Всероссийского института генетических ресурсов растений им. Н.И. Вавилова, г. Санкт-Пе-тербург—Пушкин, 59°42´37,78²С; 30°25´41,26²В, 2017 год) |
||||
Таксономический диагноз ОТЕ |
Доля ОТЕ в сообществе, % |
|||
Altern_Its1 |
Phyth_Its1 |
Altern_Its2 |
Phyth_Its2 |
|
k__Fungi; Other; Other; Other; Other; Other |
29,45 |
32,85 |
7,15 |
1,08 |
k__Fungi; p__Ascomycota; c__Dothideomycetes; o__Capnodiales; f__Cladosporiaceae; g__Cladosporium |
0,03 |
0,79 |
0,69 |
2,48 |
k__Fungi; p__Ascomycota; c__Dothideomycetes; o__Capnodiales; Other; Other |
0,02 |
0,00 |
63,38 |
71,10 |
k__Fungi; p__Ascomycota; c__Dothideomycetes; o__Pleosporales; f__Didymellaceae |
1,02 |
0,77 |
6,03 |
21,57 |
k__Fungi; p__Ascomycota; c__Dothideomycetes; o__Pleosporales; f__Phaeosphaeriaceae |
1,59 |
0,23 |
2,29 |
0,13 |
k__Fungi; p__Ascomycota; c__Dothideomycetes; o__Pleosporales; Other; Other |
0,04 |
0,10 |
1,85 |
0,46 |
k__Fungi; p__Ascomycota; c__Sordariomycetes; o__Hypocreales; f__unidentified; g__unidentified |
0,00 |
3,23 |
0,00 |
0,00 |
k__Fungi; p__Ascomycota; Other; Other; Other; Other |
1,26 |
0,43 |
0,00 |
0,00 |
k__Fungi; p__Basidiomycota; c__Microbotryomycetes; o__Sporidiobolales; f__Sporidiobolaceae; g__Sporobolomyces |
0,82 |
2,64 |
0,00 |
0,00 |
k__Fungi; p__Basidiomycota; c__Tremellomycetes; o__Cystofilobasidiales; f__Cystofilobasidiaceae; g__Cystofilobasidium |
2,70 |
1,46 |
1,15 |
0,00 |
k__Fungi; p__Basidiomycota; c__Tremellomycetes; o__Cystofilobasidiales; f__Mrakiaceae |
0,00 |
0,00 |
6,35 |
0,11 |
k__Fungi; p__Basidiomycota; c__Tremellomycetes; o__Cystofilobasidiales; f__Mrakiaceae; g__Itersonilia |
18,40 |
8,86 |
0,00 |
0,00 |
k__Fungi; p__Basidiomycota; c__Tremellomycetes; o__Cystofilobasidiales; f__Mrakiaceae; g__Udeniomyces |
0,20 |
1,59 |
0,00 |
0,00 |
k__Fungi; p__Basidiomycota; c__Tremellomycetes; o__Filobasidiales; f__Filobasidiaceae; g__Filobasidium |
0,22 |
1,39 |
0,00 |
0,00 |
k__Fungi; p__Basidiomycota; c__Tremellomycetes; o__Tremellales; f__Bulleraceae |
0,03 |
0,00 |
3,44 |
0,79 |
k__Fungi; p__Basidiomycota; c__Tremellomycetes; o__Tremellales; f__Bulleraceae; g__Bullera |
4,41 |
2,01 |
0,00 |
0,00 |
k__Fungi; p__Basidiomycota; c__Tremellomycetes; o__Tremellales; f__Bulleribasidiaceae |
0,10 |
0,51 |
3,05 |
1,10 |
k__Fungi; p__Basidiomycota; c__Tremellomycetes; o__Tremellales; f__Bulleribasidiaceae; g__Dioszegia |
1,18 |
0,83 |
0,00 |
0,00 |
k__Fungi; p__Basidiomycota; c__Tremellomycetes; Other; Other; Other |
1,51 |
0,60 |
0,00 |
0,00 |
k__Plantae; p__unidentified; c__unidentified; o__unidentified; f__unidentified; g__unidentified |
16,68 |
8,00 |
0,00 |
0,00 |
k__Stramenopila; p__Oomycota; c__Oomycetes; o__Peronosporales; f__Peronosporales_fam_Incertae_sedis; g__Phytophthora |
15,47 |
29,85 |
0,00 |
0,00 |
Примечание. Данные получены при помощи метода выскопроизводительного секвенирования на платформе MiSeq («Illumina, Inc.», США) по результатам автоматического аннотирования по базе данных UNITE (https://unite.ut.ee/); k — царство, p — филум, с — класс, о — порядок, f — семейство, g — род. Altern и Phyt — соответственно образцы с поражениями альтернариозного и фитофторозного типов, Its1 и Its2 — праймеры, использованные для создания библиотеки. |
||||
2. Доля патогенов в сообществах грибов и грибоподобных организмов, |
|||
Сообщество |
Доля в сообществе, % |
||
по типу праймера |
по типу поражения |
Alternaria |
Phytophthora |
ITS1 |
Альтернариоз |
2,07 |
15,47 |
Фитофтороз |
0,11 |
29,85 |
|
ITS2 |
Альтернариоз |
4,57 |
0 |
Фитофтороз |
0,006 |
0 |
|
Примечание. Результаты автоматического аннотирования по базе данных UNITE (https://unite.ut.ee/), скорректированного по данным дополнительного поиска в BLASTn (https://blast.ncbi.nlm.nih.gov/Blast.cgi). |
|||
3. Индексы разнообразия сообществ грибов и грибоподобных организмов, выделенных из листьев картофеля (Solanum tuberosum L.) сорта Никулинский со следами поражения фитофторозного и альтернариозного типов (опытные поля Всероссийского института генетических ресурсов растений им. Н.И. Вавилова, г. Санкт-Петербург—Пушкин, 59°42´37,78²С; 30°25´41,26²В, 2017 год) |
||||
Критерий |
Altern_Its1 |
Phyth_Its1 |
Altern_Its2 |
Phyth_Its2 |
Число таксонов |
19 |
18 |
10 |
9 |
Коэффициент Симпсона |
0,81 |
0,77 |
0,54 |
0,43 |
Коэффициент Шеннона |
1,90 |
1,87 |
1,31 |
0,84 |
Примечание. Altern и Phyt — соответственно образцы с поражениями альтернариозного и фитофторозного типов, Its1 и Its2 — праймеры, использованные для создания библиотеки. |
||||

Рис. 1. Листья картофеля (Solanum tuberosum L.) сорта Никулинский со следами поражения фитофторозного (А) и альтернариозного (Б) типов (опытные поля Всероссийского института генетических ресурсов растений им. Н.И. Вавилова, г. Санкт-Петер-бург—Пушкин, 59°42´37,78²С; 30°25´41,26²В, 2017 год) (микроскоп Stemi 508 и камера для световой микроскопии AxioCam ERc 5s, «ZEISS», Германия).
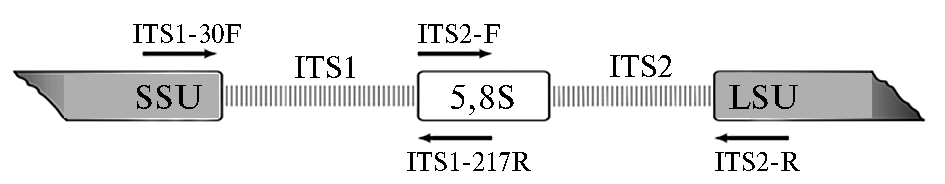
Рис. 2. Локализация праймеров для регионов ITS1 и ITS2 (35, 36), которые использовались при получении ампликонных библиотек для последующего описания сообществ, выделенных с пораженных участков листьев картофеля (Solanum tuberosum L.) сорта Никулинский: SSU — small subunit, LSU — large subunit (участки рДНК, кодирующие соответственно малую и большую рибосомальные субъединицы).
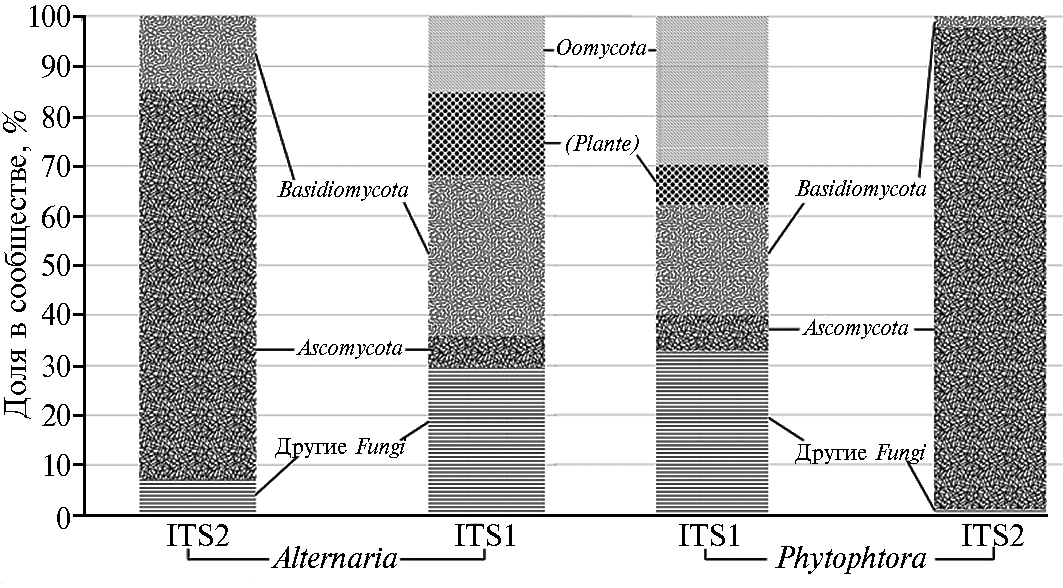
Рис. 3. Таксономическая структура сообществ грибов и грибоподобных организмов, выделенных из листьев картофеля (Solanum tuberosum L.) сорта Никулинский со следами поражения фитофторозного и альтернариозного типов, при использовании праймеров для регионов ITS1 и ITS2 (опытные поля Всероссийского института генетических ресурсов растений им. Н.И. Вавилова, г. Санкт-Петербург—Пушкин, 59°42´37,78²С; 30°25´41,26²В, 2017 год).
ЛИТЕРАТУРА
- Bernreiter A. Molecular diagnostics to identify fungal plant pathogens — a review of current methods. Revista Científica Ecuatoriana, 2017, 4: 26-35.
- Ганнибал Ф.Б., Орина А.С., Левитин М.М. Альтернариозы сельскохозяйственных культур на территории России. Защита и карантинрастений, 2010, 5: 30-32.
- Zhang Z., Luo L., Tan X., Kong X., Yang J., Wang D., Zhang D., Jin D., Liu Y. Pumpkin powdery mildew disease severity influences the fungal diversity of the phyllosphere. PeerJ, 2018, 6: e4559 (doi: 10.7717/peerj.4559).
- Козловский Б.Е., Филлипов А.В. Альтернариоз на картофеле становится более вредоносным. Защита икарантинрастений, 2007, 5: 12-13.
- Поштаренко А.Ю., Смирнов А.Н. Фитофтороз и альтернариоз картофеля и томата при аномальных погодных условиях в Московской области. Защита и карантин растений, 2011, 12: 40-42.
- Николаев А.В., Сезонова Н.П., Любимская И.Г., Ланге Ф. Урожайность и устойчивость к фитофторозу и альтернариозу сортов картофеля в условиях Костромской области. Аграрная наука Евро-Северо-Востока, 2014, 3(40): 19-24.
- Bourke A. The Visitation of God?: The Potato and the Great Irish Famine. Dublin, Lilliput Press, Ltd., 1993.
- Денисенков И.А. Эффективная защита картофеля от болезней различной этиологии в условиях Брянской области. Достижения науки и техники АПК, 2018, 32(3): 76-78 (doi: 10.24411/0235-2451-2018-10315).
- Киру С.Д., Костина Л.И., Рогозина Е.В., Ясько А.А., Чалая Н.А., Жигадло Т.Э. Итоги изучения генофонда картофеля и перспективы его использования в селекции. Труды по прикладной ботанике, генетике и селекции, 2013, 173: 91-101.
- Побединская М.А., Плуталов П.Н., Романова С.С., Кокаева Л.Ю., Николаев А.В., Александрова А.В., Еланский С.Н. Устойчивость возбудителей альтернариоза картофеля и томата к фунгицидам. Микология и фитопатология, 2012, 46(6): 401-408.
- Смирнов А.Н., Бибик Т.С., Приходько Е.С., Белошапкина О.О., Кузнецов С.А. Листостебельный комплекс фитопатогенных и сопутствующих грибов на картофеле в различных регионах России. Известия ТСХА, 2015, 3: 36-46.
- Kowalik M. Diversity of fungi colonizing and damaging leaves of pontic azalea Azalea pontica. Acta Mycologica, 2013, 48(2): 227-236 (doi: 10.5586/am.2013.024).
- Russo M.L., Pelizza S.A., Cabello M.N., Stenglein S.A., Vianna M.F., Scorsett A.C. Endophytic fungi from selected varieties of soybean (Glycine max L. Merr.) and corn (Zea mays L.) grown in an agricultural area of Argentina. Revista Argentina de Microbiología, 2016, 48(2): 154-160 (doi: 10.1016/j.ram.2015.11.006)
- Yang H., Ye W., Ma J., Zeng D., Rong Z., Xu M., Wang Y., Zheng X. Endophytic fungal communities associated with field-grown soybean roots and seeds in the Huang-Huai region of China. PeerJ, 2018, 6: e4713 (doi: 10.7717/peerj.4713).
- Qin S., Yeboah S., Xu X., Liu Y., Yu B. Analysis on fungal diversity in rhizosphere soil of continuous cropping potato subjected to different furrow-ridge mulching managements. Frontiers in Microbiology, 2017, 8: 845 (doi: 10.3389/fmicb.2017.00845).
- Xu L. Soil fungal communities associated with plant health as revealed by next-generation sequencing. PhD thesis. Slagelse, 2011.
- Youssuf G., Gashgari R.M. Mycobiota associated with superficial blemishes of potato tubers. Food Biotechnology, 2013, 27(2): 137-151 (doi: 10.1080/08905436.2013.781947).
- Mazur S., Kurzavińska H., Nadziakiewicz M., Nawrocki J. Redroot pigweed as a host for Alternaria alternata — the causal agent of Alternaria leaf blight in potato. Zemdirbyste-Agriculture, 2015, 102(1): 115-118 (doi: 10.13080/z-a.2015.102.015).
- Cwalina-Ambroziak B., Trojak A. Effectiveness of selected fungicides in potato protection against Phytophthora Infestans and Alternaria spp. Polish Journal of Natural Science, 2011, 26(4): 275-284.
- Cwalina-Ambroziak B., Damszel M.M., Głosek-Sobieraj M. The effect of biological and chemical control agents on the health status of the very early potato cultivar Rosara. Journal of Plant Protection Research, 2015, 55(4): 389-395 (doi: 10.1515/jppr-2015-0052).
- Приходько Е.С., Селицкая О.В., Смирнов А.Н. Влияние фунгицидов и культуральной жидкости ризобактерии Klebsiella planticola на развитие фитопатогена AlternariaAlternata на картофеле. Известия ТСХА, 2016, 5: 68-78.
- Vutto N.L., Gapeeva T.A., Pundik A.N., Tretyakova T.G., Volotovski I.D. Transgenic Belarusian-bred potato plants expressing the genes for antimicrobial peptides of the cecropin-melittin type. Russian Journal of Genetics, 2010, 46(12): 1626-1634 (doi: 10.1134/S1022795410120057).
- Raja H.A., Miller A.N., Pearce C.J., Oberlies N.H. Fungal identification using molecular tools: a primer for the natural products research community. Journal of Natural Products, 2018, 80(3): 756-770 (doi: 10.1021/acs.jnatprod.6b01085).
- Riley M.B., Williamson M.R., Maloy O. Plant disease diagnosis. Plant Health Instructor, 2002 (doi: 10.1094/PHI-I-2002-1021-01). Режим доступа: https://www.apsnet.org/edcenter/disimpact-mngmnt/casestudies/Pages/PlantDiseaseDiagnosis.aspx. Дата обращения 19.06.2019.
- Ray M., Ray A., Dash S., Mishra A., Achary K.G., Nayak S., Singh S. Fungal disease detection in plants: Traditional assays, novel diagnostic techniques and biosensors. Biosensors and Bioelectronics, 2017, 87: 708-723 (doi: 10.1016/j.bios.2016.09.032).
- Sankaran S., Mishra A., Ehsani R., Davis C. A review of advanced techniques for detecting plant diseases. Computers and Electronics in Agriculture, 2010, 72(1): 1-13 (doi: 10.1016/j.compag.2010.02.007).
- Kumbhar N.P., Patil S.B. A review for agricultural plant diseases detection using different techniques. International Journal of Electrical and Electronics Engineers, 2017, 9(1): 891-901.
- Гагкаева Т.Ю., Ганнибал Ф.Б., Гаврилова О.П. Метод ПЦР-диагностики фитопатогенных грибов родов Fusarium и Alternaria. В сб.: Высокопроизводительные и высокоточные технологии и методы фитосанитарного мониторинга. СПб, 2009: 4-14.
- Lau H.Y., Botella J.R. Advanced DNA-based point-of-care diagnostic methods for plant diseases detection. Front. Plant Sci., 2017, 8: 2016 (doi: 10.3389/fpls.2017.02016).
- Lees A.K., Sullivan L., Cullen D.W. A quantitative polymerase chain reaction assay for the detection of Polyscytalum pustulans, the cause of skin spot disease of potato. Journal of Phytopathology, 2009, 157(3): 154-158 (doi: 10.1111/j.1439-0434.2008.01459.x).
- Кокаева Л.Ю., Хуснетдинова Т.И., Березов Ю.И., Балабко П.Н., Еланский С.Н. Видовой состав грибов, ассоциированных с листьями картофеля. Защита картофеля, 2017, 2: 8-11.
- Павловская Н.Е., Солохина И.Ю., Лушников А.В. Создание тест-систем для идентификации фитопатогенов методом твердофазного иммуноферментного анализа. Биология в сельском хозяйстве, 2015, 4: 7-11.
- Schmidt P.A., Bálint M., Greshake B., Bandow C., Römbke J., Schmitt I. Illumina metabarcoding of a soil fungal community. Soil Biology and Biochemistry, 2013, 65: 128-132 (doi: 10.1016/j.soilbio.2013.05.014).
- Motooka D., Fujimoto K., Tanaka R., Yaguchi T., Gotoh K., Maeda Y., Furuta Y., Kurakawa T., Goto N., Yasunaga T., Narazaki M., Kumanogoh A., Horii T., Iida T., Takeda K., Nakamura S. Fungal ITS1 deep-sequencing strategies to reconstruct the composition of a 26-species community and evaluation of the gut mycobiota of healthy Japanese individuals. Frontiers in Mycrobiology, 2017, 8: 238 (doi: 10.3389/fmicb.2017.00238).
- Usyk M., Zolnik C.P., Patel H., Levi M.H., Burk R.D. Novel ITS1 fungal primers for characterization of the mycobiome. mSphere, 2017, 2(6): e00488-17 (doi: 10.1128/mSphere.00488-17).
- White T.J., Bruns T., Lee S., Taylor J. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics. In: PCR Protocols: Aguide to methods and applications /M.A. Innis, D.H. Gelfand, J.J. Sninsky, T.J. White (eds.). San Diego, Academic Press, 1990: 315-322 (doi: 10.1016/b978-0-12-372180-8.50042-1).
- Blaalid R., Kumar S., Nilsson R.H., Abarenkov K., Kirk P.M., Kauserud H. ITS1 versus ITS2 as DNA metabarcodes for fungi. Molecular Ecology Resources, 2013, 13(2): 218-224 (doi: 10.1111/1755-0998.12065).
- Seifert K.A. Progress towards DNA barcoding of fungi. Molecular Ecology Resources, 2009, 9(s1): 83-89 (doi: 10.1111/j.1755-0998.2009.02635.x).
- Caporaso J.G, Kuczynski J., Stombaugh J., Bittinger K., Bushman F.D., Costello E.K., Fierer N., Peña A.G., Goodrich J.K., Gordon J.I., Huttley G.A., Kelley S.T., Knights D., Koenig J.E., Ley R.E., Lozupone C.A., McDonald D., Muegge B.D., Pirrung M., Reeder J., Sevinsky J.R., Turnbaugh P.J., Walters W.A., Widmann J., Yatsunenko T., Zaneveld J., Knight R. QIIME allows analysis of high-throughput community sequencing data. Nature Methods, 2010, 7(5): 335-336 (doi: 10.1038/nmeth.f.303).
- Gweon H.S., Oliver A., Taylor J., Booth T., Gibbs M., Read D.S., Schonrogge K. PIPITS: an automated pipeline for analyses of fungal internal transcribed spacer sequences from the Illumina sequencing platform. Methods in Ecology and Evolution, 2015, 6(8): 973-980 (doi: 10.1111/2041-210X.12399).
- Hammer Ø., Harper D.A.T., Ryan P.D. PAST: Paleontological statistics software package for education and data analysis. Palaeontologia Electronica, 2001, 4(1): 9.
- Altschul S.F., Gish W., Miller W., Myers E.W., Lipman D.J. Basic local alignment search tool. Journal of Molecular Biology, 1990, 215(3): 403-410 (doi: 10.1016/S0022-2836(05)80360-2).
- Halwachs B., Madhusudhan N., Krause R., Nilsson R.H., Moissl-Eichinger C., Högenauer C., Thallinger G.G., Gorkiewicz G. Critical issues in mycobiota analysis. Frontiers in Mycrobiology, 2017, 8: 180 (doi: 10.3389/fmicb.2017.00180).