doi: 10.15389/agrobiology.2019.4.631rus
УДК 636.2:636.082.12
Исследования выполнены при поддержке РНФ, проект № 19-76-20012.
ГЕНЕТИЧЕСКИЕ РЕСУРСЫ ЖИВОТНЫХ: РАЗВИТИЕ ИССЛЕДОВАНИЙ АЛЛЕЛОФОНДА РОССИЙСКИХ ПОРОД КРУПНОГО РОГАТОГО СКОТА — МИНИОБЗОР
Н.А. ЗИНОВЬЕВА1, А.А. СЕРМЯГИН1, А.В. ДОЦЕВ1, О.И. БОРОНЕЦКАЯ2, Л.В. ПЕТРИКЕЕВА2, А.С. АБДЕЛЬМАНОВА2,
G. BREM1, 3
В современной биологической науке изучение и сохранение биоразнообразия рассматривается как одна из важных и актуальных задач (L.F. Groeneveld с соавт., 2010). В ХХ столетии во всем мире в животноводстве использовалось ограниченное число пород, что привело к существенному снижению численности локальных пород, которые до недавнего времени активно вовлекались в сельскохозяйственное производство (B. Rischkowsky с соавт., 2007). Настоящий обзор описывает современное состояние знаний о генофонде крупного рогатого скота (КРС), при этом особое внимание уделено российским генетическим ресурсам. Дано краткое описание эволюции методов, применяемых при изучении генетического разнообразия. Обобщены результаты исследований аллелофонда пород крупного рогатого скота на основе анализа полиморфизма митохондриальной ДНК и микросателлитов (M.-H. Li с соавт., 2009; J. Kantanen с соавт., 2009; P.V. Gorelov с соавт., 2011; T.Yu. Kiseleva с соавт., 2014; A.A. Traspov с соавт., 2011; R. Sharma 2015). Дискутируются преимущества использования полиморфизмов единичных нуклеотидов (single nucleotide polymorphism, SNP) в режиме полногеномного сканирования для изучения популяционной структуры и генетических связей между породами (R. Fries с соавт., 2001; R. Martinez-Arias с соавт., 2001; C. Xing с соавт., 2005). Приведены данные о дивергенции пород на основании анализа их полногеномных SNP профилей (J.E. Decker с соавт., 2009; L.A. Kuehn с соавт., 2011; E.J. McTavish с соавт., 2013; J.E. Decker с соавт., 2014; J.E. Decker с соавт., 2016; T. Iso-Touru с соавт., 2016). Охарактеризован современный аллелофонд отечественных пород скота (N.A. Zinovieva с соавт., 2016; A. Yurchenko с соавт., 2018; A.A. Sermyagin с соавт., 2018). В сравнительных исследованиях пород Bos taurus Евразии установлена высокая генетическая специфичность якутского скота. Показано сохранение существенной доли аутентичных генетических компонентов в ряде российских пород (холмогорской, ярославской, красной горбатовской), что позволяет рассматривать их в качестве наиболее значимых национальных генетических ресурсов и обосновывает необходимость более глубокого изучения и сохранения этих пород. Отмечается, что использование даже такого мощного инструмента, как анализ множественных SNPs, не всегда позволяет однозначно интерпретировать полученные результаты с точки зрения демографической истории отечественных пород. Это обусловлено существенными изменениями в аллелофонде современных популяций как российских пород, так и пород, предположительно принимавших участие в их формировании. Информационная значимость результатов, полученных молекулярно-генетическими методами при изучении эволюции пород, может быть существенно повышена за счет вовлечения в исследования исторических образцов ДНК, например костного материала из краниологических и остеологических коллекций (О.И. Боронецкая с соавт., 2017). В настоящее время разработаны методы, позволяющие получать препараты, которые пригодны для широкого спектра молекулярно-генетических исследований как митохондриальной, так и ядерной ДНК, включая анализ на уровне индивидуальных генов и полных геномов (D.E. McHugh с соавт., 2000; A. Beja-Pereira с соавт., 2006; M. Gargani с соавт., 2015). Вовлечение в исследования исторических образцов позволит получить новые данные об эволюции отечественного аллелофонда пород и уточнить происхождение современных популяций. Результаты таких исследований найдут применение при разработке программ сохранения пород, а также в селекции при создании органических систем производства, основанных на использовании отечественных генетических ресурсов.
Ключевые слова: биоразнообразие, отечественные породы крупного рогатого скота, ДНК маркеры, исторические образцы ДНК.
Изучение и сохранение биоразнообразия становится все более актуальной задачей современной биологической науки (1). Сельскохозяйственные генетические ресурсы — это национальный и мировой биологический капитал, крайне необходимый для развития систем производства будущего (2). В XX столетии развитие животноводства во всем мире сконцентрировалось на использовании ограниченного числа пород, что привело к существенному снижению численности локальных пород, которые до недавнего времени активно вовлекались в сельскохозяйственное производство (3). Так, за последние полвека суммарная численность шести российских локальных пород молочного направления продуктивности (холмогорская, ярославская, бестужевская, костромская, красная горбатовская, суксунская) снизилась более чем в 10 раз — с 3,4 млн гол. в 1960 году до 312,6 тыс. гол. в 2015 году, при этом численность ярославской, костромской и бестужевской пород уменьшилась в 19-37 раз. Современная популяция красного горбатовского скота по численности сегодня составляет лишь около 0,6 % от показателя середины прошлого столетия (рис.).
С увеличением спроса на продукцию животноводства в мире в целом и в России в частности следует ожидать дальнейшей ориентации производства на использование нескольких основных пород и, как следствие, усиления тенденции снижения биоразнообразия.
Наряду с уменьшением численности локальных пород, наблюдается активное использование скрещивания оставшейся незначительной части чистопородного поголовья, что ставит локальные породы под угрозу исчезновения (3). С генетической точки зрения, использование кроссбридинга приводит к исчезновению целого ряда уникальных аллелей, прежде всего редких, что может стать причиной потери ценных признаков и свойств пород, включая компонентный состав продукции, устойчивость к заболеваниям, хорошую способность к адаптации к природно-климатическим условиям конкретных регионов и др. В этой связи важно оценить существующее состояние аллелофонда пород, идентифицировать популяции и индивидуумов — носителей исходных породоспецифических аллелей и уникальных аллельных сочетаний для разработки на основе полученной информации программ сохранения генетической аутентичности пород.
Настоящий обзор описывает современное состояние знаний в области исследования генофонда крупного рогатого скота, при этом особое внимание уделено отечественным генетическим ресурсам. Дискутируются возможности привлечения исторических образцов для изучения эволюции пород.
Для реконструкции демографической истории пород сельскохозяйственных животных нашли применение различные типы ДНК маркеров (7). В 1960-х годах главным инструментом для характеристики генетического разнообразия, популяционной структуры и генеалогических связей между породами был анализ групп крови и полиморфизма белков молока (8, 9). Выявление полиморфизмов митохондриальной ДНК (мтДНК) и высокополиморфных микросателлитов открыло новые возможности для более информативных генетических исследований (10). Исследование полиморфизма D-петли мтДНК показало наличие двух независимых очагов доместикации тауринового и зебувидного скота (11-13), что впоследствии подтвердилось при изучении микросателлитов (14, 15). Микросателлиты были использованы для верификации гибридного происхождения ближневосточных пород скота (15, 16), демонстрации различного исторического происхождения средиземноморских и североевропейских популяций крупного рогатого скота (17), уточнения классификации евразийских пород крупного рогатого скота (18), определения происхождения некоторых локальных популяций и пород (19, 20). Полиморфизмы митохондриальной ДНК и микросателлиты были успешно использованы для оценки разнообразия, установления генетических связей, характеристики популяционной структуры ряда российских пород крупного рогатого скота на глобальном (21) и региональном уровне (22-24). Результаты M.-H. Li и J. Kantanen (18) подтвердили экспансию высокопродуктивных пород, таких как красная датская, ангельнская, голштино-фризская и айрширская, в северную и восточную Европу, включая Россию. Авторы указывают на смешанное происхождение ярославской, истобенской и холмогорской пород скота, что, по их мнению, является следствием использования кроссбридинга и может отражать наличие множественных миграционных событий из соседних областей Европы, Азии и Ближнего Востока. Во всех вышеназванных породах отмечалось наличие заметного генетического компонента европейского черно-пестрого равнинного скота. Следует, однако, отметить, что это противоречит мнению ряда авторов, которые указывают на то, что холмогорский, ярославский и истобенский скот формировался лишь с незначительным влиянием голштино-фризского скота (25, 26). При сравнительных исследованиях патернального (Y-хромосомальные микросателлитные гаплотипы) и матернального (гаплотипы мтДНК) разнообразия J. Kantanen с соавт. (21) показали ускоренную потерю Y-хромосомальной изменчивости у локально разводимого скота, включая российские породы (холмогорская, ярославская, истобенская, суксунская, красная горбатовская), что, по мнению авторов, стало следствием интенсивной выбраковки племенных быков и отцовской формы кроссбридинга. На основании анализа мтДНК было показано сходство финских и северных российских пород скота. С использованием микросателлитов охарактеризовано состояние аллелофонда и генетической структуры сычевской и симментальской пород (22, 24), получены данные о неравновесии по сцеплению 29 микросателлитных локусов в шести отечественных популяциях скота (23).
Еще один метод анализа полиморфизма одновременно большого количества локусов, нашедший широкое применение в изучении генофондов сельскохозяйственных животных, — исследование полиморфизма фрагментов ДНК, фланкированных инвертированными повторами микросателлитных локусов (inter simple sequence repeats, ISSR маркеры). ISSR маркеры позволяют анализировать сходство и различия генофондов видов, пород (внутрипородных групп), проводить оценку их консолидированности, чистопородности и генеалогических связей (27, 28) В исследованиях российских и зарубежных пород крупного рогатого скота выявлен спектр ISSR локусов, которые могут использоваться для маркирования видов Bos taurus и Bos indicus, описания стандарта пород, их генетического профиля, породоспецифичного паттерна (29, 30).
Анализ полиморфизма мтДНК, микросателлитов и ISSR-маркеров позволил продвинуться в понимании истории происхождения и эволюции пород, однако в связи с недостаточной информативностью этих типов ДНК маркеров результаты исследований не всегда могли быть однозначно интерпретированы.
Развитие новых высокопроизводительных технологий генотипирования привело к широкому использованию в исследовании геномов животных ДНК маркеров на основе полиморфизмов единичных нуклеотидов (single nucleotide polymorphism, SNP). В настоящее время SNP — предпочтительный тип маркеров для геномной оценки, установления родства между индивидуумами, определения степени инбридинга и гибридизации, генетического картирования высокого разрешения и более полной характеристики генетических ресурсов (31, 32). Как главное преимущество SNP в качестве маркеров по сравнению с микросателлитами рассматривается их широкое распространение в геноме, ясный мутационный механизм с низким уровнем гомоплазии и более низкая степень мутабильности. Методическими преимуществами анализа SNP считается отсутствие особых требований к качеству ДНК (анализ SNP проводится, как правило, посредством получения коротких ампликонов длиной менее 100 п.н.), более низкая степень ошибочного генотипирования, возможность автоматизации процесса с применением технологий высокопроизводительного генотипирования и стандартизации получаемых данных (33-35). SNPs способны обеспечивать более широкое покрытие генома по сравнению с STR (short tandem repeats) и могут быть использованы в случае как нейтральных, так и находящихся под давлением отбора генов (36, 37). Несмотря на существующее многообразие методов изучения SNP (37), в настоящее время все большее распространение получает технология полногеномного анализа на платформе BeadArrayTM («Illumina, Inc.», США), позволяющая проводить одновременное тестирование от нескольких десятков до нескольких сотен тысяч таких SNPs (38). Доминированию SNP маркеров в исследовании геномов животных способствовала разработка и вывод на рынок коммерческих ДНК-чипов, позволяющих проводить одновременный анализ десятков и даже сотен тысяч SNPs (39).
Анализ SNP на полногеномном уровне с использованием Bovine SNP50 BeadChip («Illumina, Inc.», США) успешно применялся для уточнения исторического происхождения и характеристики современной структуры популяций крупного рогатого скота во всем мире (40-45). В масштабном исследовании J.E. Decker c соавт. (40) была сконструирована филогенетическая сеть, включающая 48 пород крупного рогатого скота (n = 372), что позволило точно описать генетические связи между породами и улучшить доказательную базу касательно истории доместикации и формирования пород. E.J. McTavish с соавт. (38) показали, что североамериканские породы, а также многие родственные породы Южной Европы имеют гибридное происхождение, проявляя геномные компонентыкак Bos taurus, так и B. indicus. Используя широкий спектр аналитических методов, J.E. Decker c соавт. (43) смогли четко разделить 134 породы домашнего крупного рогатого скота (n = 1543) между тремя группами: азиатскийBosindicus, евразийский B. taurus и африканский B. taurus. Было показано, что африканская группа B. taurus содержит большую долю предков африканского тура, что обусловливает ее дивергенцию от группы евразийской группы B. taurus. Показано влияние других видов, отличных от B. taurus и B. indicus, на формирование азиатских пород крупного рогатого скота; установлено заметное влияние шортгорнов на развитие европейских пород, выявлена интрогрессия африканского скота B. taurus у иберийских и итальянских пород скота (43).
Исследования российских пород скота на полногеномном уровне пока менее масштабны. Для оценки генетического разнообразия и структуры современных популяций пяти российских пород скота — бестужевской, холмогорской, костромской, красной горбатовской и ярославской были использованы 35874 полиморфных SNPs (46). Анализ полученных SNP профилей позволил четко дифференцировать российские породы друг от друга и от голштинской, используемой в качестве сравнения. Все исследованные российские породы имели сложное происхождение и проявляли наличие геномных компонентов других российских пород, а в некоторых случаях — наличие примеси голштинов. Исследование 274 индивидуумов 18 отечественных пород в сравнении с 135 мировыми породами (47) показало их разделение на 4 кластера, отражающих предковые взаимосвязи с другими породами. Было показано, что некоторые породы (например, холмогорская, якутская, ярославская) имеют специфические генетические профили, что делает их приоритетными для более глубоких исследований. Детальное изучение популяционной структуры и взаимосвязей девяти отечественных пород скота (бестужевской, черно-пестрой, калмыцкой, холмогорской, костромской, красной горбатовской, суксунской, якутской и ярославской) с другими 36 породами Bostaurus Евразии выполнено A.A. Sermyagin с соавт. (48). Анализ посредством многомерного шкалирования (multidimensional scaling, MDS) подтвердил отнесение всех исследованных отечественных пород к Bostaurus. По результатам анализа Neighbor-Net и Admixture все породы разделились на три группы. Якутский и калмыцкий скот сформировал отдельную группу вследствие турано-монгольского происхождения. Черно-пестрая, костромская и суксунская породы проявляли наличие существенной доли предковых компонентов трансграничных европейских пород — соответственно голштинской, бурой швицкой и красной датской. Холмогорская, ярославская, красная горбатовская и бестужевская породы характеризовались наименьшей долей интрогрессии трансграничных пород, что делает их наиболее значимым национальным генетическим ресурсом.
Вместе с тем следует отметить, что исследование современных популяций локальных российских пород скота даже с использованием такого мощного инструмента, как полногеномный SNP-анализ, не всегда позволяет однозначно интерпретировать полученные результаты с точки зрения демографической истории пород. Причина заключается в том, что аллелофонд современных популяций как российских пород, так и пород, предположительно принимавших участие в их формировании, в процессе развития подвергся существенным изменениям. С одной стороны, это вызвано влиянием меняющихся природно-климатических условий, с другой — корректировкой направлений и методов селекции. Практикуемая в последние десятилетия интенсивная селекция по ограниченному числу признаков, несомненно, способствовала закреплению в популяциях аллелей, положительно ассоциированных с селектируемыми признаками, и элиминации ряда других аллелей — нейтральных или находящихся в отрицательной зависимости по отношению к таким признакам. Еще одной причиной, затрудняющей идентификацию аутентичных генетических компонентов у современных популяций отечественного скота, стало практикуемое в последние десятилетия активное использование кроссбридинга с высокопродуктивными трансграничными породами. Так, красные голштины интенсивно используются для улучшения скота бестужевской породы (49), черно-пестрый голштинский скот — для улучшения холмогорской (54, 55) и тагильской пород (56, 57), бурый швицкий — для улучшения костромского скота (54-56) и т.д. Практически полностью поглощена голштинами отечественная черно-пестрая порода: анализ родословных более 500 быков-производителей, используемых в системе искусственного осеменения в России (57), показал наличие лишь нескольких животных, имеющих 50 % и более крови черно-пестрой породы. Другой проблемой при интерпретации данных молекулярно-генетических исследований может стать то, что некоторые исходные породы, участвующие в формировании российских пород скота, прекратили свое существование. Так произошло, например, с тирольским скотом, имевшим широкое распространение в Европе в XIX веке (58). Предполагают, что тирольский скот, массовый экспорт которого в Россию датирован 1848 годом (59), оказал существенное влияние на формирование аллелофонда бестужевской и красной горбатовской пород (60).
Информационную значимость результатов молекулярно-генетических исследований демографического происхождения пород можно существенно повысить за счет вовлечения в исследования исторических образцов ДНК. Полученные ДНК-профили таких животных могут быть использованы в качестве эталона при определении генофондного статуса современных популяций скота, а также для идентификации особей — носителей уникальных аллелей или аллельных сочетаний, свойственных историческим образцам и отсутствующих у большинства животных современных популяций. Информация о генотипах исторических образцов может быть использована при проведении полногеномных ассоциативных исследований (genome wide association study, GWAS), а также при определении мишеней для геномного редактирования. Источником ДНК для такого рода анализов могут стать образцы костной ткани черепов из краниологических коллекций (61). Краниологический метод был основным при изучении происхождения домашних животных до открытия биологических маркеров. Впервые его предложил в 1865 году профессор сравнительной анатомии из Швейцарии Ludwig Rütimeyer (1825-1898) (цит. по 62). Затем последовал ряд аналогичных работ, выполненных на различных породах скота. В России сравнительно-анатомический метод применили при обследовании крупного рогатого скота К.Э. Линдерман (1873 и 1874 годы), А.Ф. фон-Миддендорф (1884 год), П.Н. Кулешов (1888 год), Е.Ф. Лискун (1910 год), Ю.А. Филипченко (1915 и 1916 годы), В.П. Устьянцев (1915), А.А. Браунер (1919 год) (цит. по 62). Краниологический метод изучения отечественных пород скота получил наибольшее развитие в работах Е.Ф. Лискуна. Он предложил методику краниологических исследований (63), которая более 50 лет активно использовалась в изучении сельскохозяйственных животных разных видов. Е.Ф. Лискун стал создателем уникальной краниологической и остеологической коллекции домашних животных из различных районов Европы и Азии, не имеющей аналогов как в России, так и за рубежом. В 1947 году коллекция была передана в дар Московской сельскохозяйственной академии им. К.А. Тимирязева. Коллекция содержит более 700 черепов животных, в том числе 350 — крупного рогатого скота 41 породы и породной группы (64). Учитывая, что экспонаты коллекции датированы концом XIX—началом XX века, их использование в молекулярно-генетических исследованиях позволит получить эталонные профили пород скота и проследить их эволюцию за более чем 100-летний период истории.
В настоящее время разработаны методы, позволяющие получать ДНК, пригодную для проведения широкого спектра молекулярно-генетических исследований как митохондриальной, так и ядерной ДНК, включая анализ на уровне индивидуальных генов и полных геномов (65-67). Уже первые результаты молекулярно-генетических исследований с использованием в качестве ДНК-маркеров 10 локусов микросателлитов (TGLA227, BM2113, ETH10, SPS115, TGLA122, INRA23, TGLA126, BM1818, ETH225, BM1824) показали заметные генетические различия между современными и историческими образцами скота ярославской и холмогорской пород. Развитие исследований с привлечением дополнительного числа исторических образцов и расширением спектра используемых ДНК маркеров позволит получить новые данные об эволюции отечественного аллелофонда пород за последнее столетие.
Итак, результаты проведенных исследований генофондов российских пород крупного рогатого скота показывают сохранение в большинстве пород аутентичных геномных компонентов, что делает их важнейшими национальными генетическими ресурсами и резервом изменчивости, необходимым для поддержания устойчивости систем сельскохозяйственного производства в будущем. Сравнение современных и исторических образцов на геномном уровне с привлечением набора ДНК маркеров даст результаты, которые найдут применение при разработке программ сохранения пород, а также в селекции при создании органических систем производства, основанных на использовании отечественных генетических ресурсов.
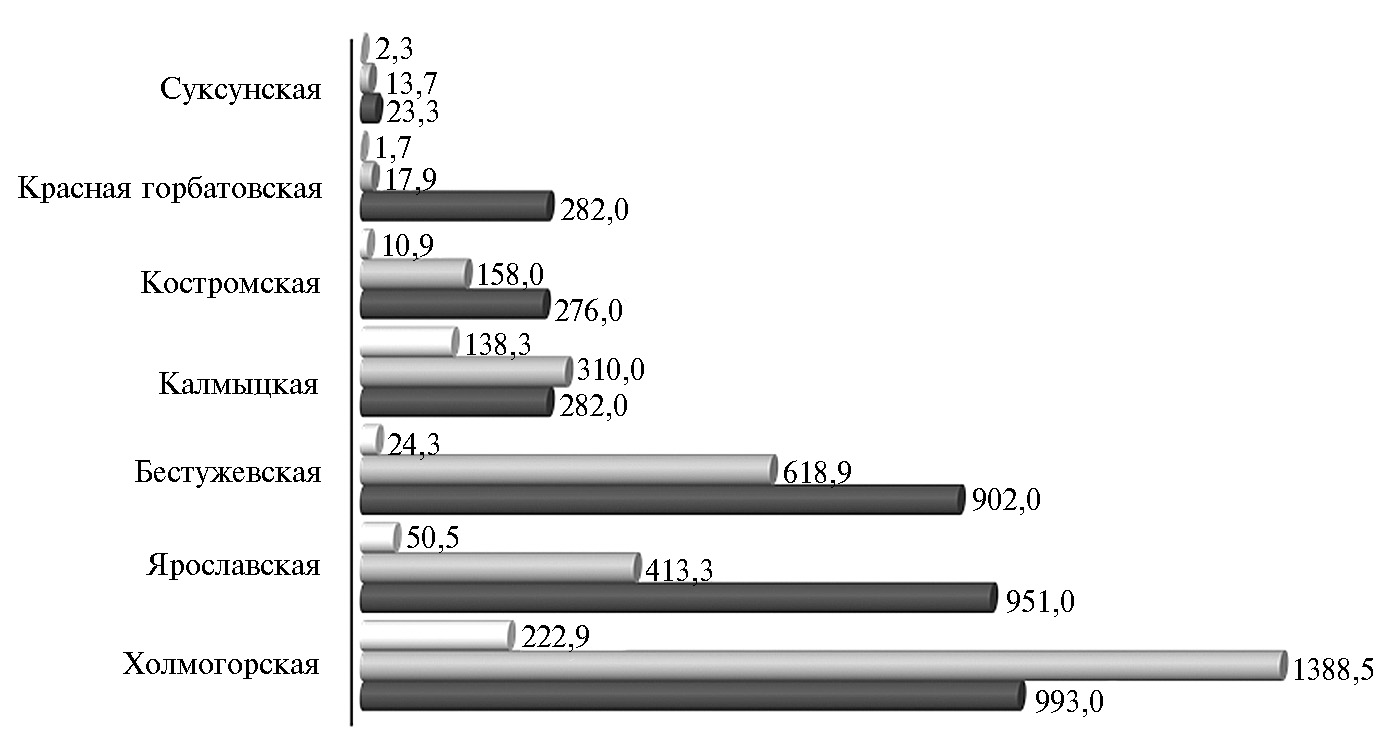
Изменение численности локальных российских пород крупного рогатого скота (тыс. гол.):  —2015 год,
—2015 год,  — 1991 год,
— 1991 год,  — 1960 год (4-6).
— 1960 год (4-6).
ЛИТЕРАТУРА
- Groeneveld L.F., Lenstra J.A., Eding H., Toro M.A., Scherf B., Pilling D., Negrini R., Finlay E.K., Jianlin H., Groeneveld E., Weigend S., GLOBALDIV Consortium. Genetic diversity in farm animals — a review. Anim. Genet., 2010, 41: 6-31 (doi: 10.1111/j.1365-2052.2010.02038.x).
- Toro M., Fernández J., Caballero A. Molecular characterization of breeds and its use in conservation. Livestock Science, 2009, 120(3): 174-195 (doi: 10.1016/j.livsci.2008.07.003).
- The state of the world’s animal genetic resources for food and agriculture /B. Rischkowsky, D. Pilling (eds.). FAO, Rome, Italy, 2007.
- Численность породного скота в колхозах и совхозах СССР на 1 января 1960 г. Статистический сборник. М., 1961.
- Ежегодник по племенной работе в молочном скотоводстве в хозяйствах Российской Федерации (1991 год). М., 1992.
- Ежегодник по племенной работе в молочном скотоводстве в хозяйствах Российской Федерации (2015 год). М., 2016.
- Yang W., Kang X., Yang Q., Lin Y., Fang M. Review on the development of genotyping methods for assessing farm animal diversity. Journal of Animal Science and Biotechnology, 2013, 4(1): 2-6 (doi: 10.1186/2049-1891-4-2).
- Rendel J. Relationships between blood groups and the fat percentage of the milk in cattle. Nature, 1961, 189: 408-409.
- Neimann-Sorensen A., Robertson A. The association between blood groups and several production characteristics in three Danish cattle breeds. Acta Agriculturae Scandinavica, 1961, 11(2): 163-196 (doi: 10.1080/00015126109433054).
- Kühn Ch., Freyer G., Weikard R., Goldammer T., Schwerin M. Detection of QTL for milk production traits in cattle by application of a specifically developed marker map of BTA6. Animal Genetics, 1999, 30(5): 333-340 (doi: 10.1046/j.1365-2052.1999.00487.x).
- Loftus R.T., MacHugh D.E., Bradley D.G., Sharp P.M., Cunningham E.P. Evidence for two independent domestications of cattle. PNASUSA, 1994, 91: 2757-2761 (doi: 10.1073/pnas.91.7.2757).
- Loftus R.T., MacHugh D.E., Ngere L.O., Balain D.S., Badi A.M., Bradley D.G., Cunningham E.P. Mitochondrial genetic variation in European, African and Indian cattle populations. Animal Genetics, 1994, 25(4): 265-271 (doi: 10.1111/j.1365-2052.1994.tb00203.x).
- Bradley D.G., MacHugh D.E., Cunningham P., Loftus R.T. Mitochondrial diversity and the origins of African and European cattle. PNAS USA, 1996, 93(10): 5131-5135 (doi: 10.1073/pnas.93.10.5131.
- MacHugh D.E., Shriver M.D., Loftus R.T., Cunningham P., Bradley D.G. Microsatellite DNA variation and the evolution, domestication and phylogeography of taurine and zebu cattle (Bos taurus and Bos indicus). Genetics, 1997, 146(3): 1071-1086.
- Loftus R.T., Ertugrul O., Harba A.H., El-Barody M.A.A., MacHugh D.E., Park S.D.E., Bradley D.G. A microsatellite survey of cattle from a centre of origin: the Near East. Molecular Ecology, 1999, 8(12): 2015-2022 (doi: 10.1046/j.1365-294x.1999.00805.x).
- Edwards C.J., Baird J.F., MacHugh D.E. Taurine and zebu admixture in Near Eastern cattle: a comparison of mitochondrial, autosomal and Y-chromosomal data. Animal Genetics, 2007, 38(5): 520-524 (doi: 10.1111/j.1365-2052.2007.01638.x).
- Cymbron T., Freeman A.R., Isabel Malheiro M., Vigne J.D., Bradley D.G. Microsatellite diversity suggests different histories for Mediterranean and Northern European cattle populations. Proc. R. Soc. B., 2005, 272: 1837-1843 (doi: 10.1098/rspb.2005.3138).
- Li M.-H., Kantanen J. Genetic structure of Eurasian cattle (Bos taurus) based on microsatellites: clarification for their breed classification. Animal Genetics, 2009, 41(2): 150-158 (doi: 10.1111/j.1365-2052.2009.01980.x).
- MacNeil M.D., Alexander L.J., Kantanen J., Ammosov I.A., Ivanova Z.I., Popov R.G., Ozerov M., Millbrooke A., Cronin M.A. Potential emigration of Siberian cattle germplasm on Chirikof Island, Alaska. Journal of Genetics, 2017, 96(1): 47-51 (doi: 10.1007/s12041-016-0739-6).
- Sharma R., Kishore A., Mukesh M., Ahlawat S., Maitra A., Kumar Pandey A., Tantia M.S. Genetic diversity and relationship of Indian cattle inferred from microsatellite and mitochondrial DNA markers. BMC Genetics, 2015, 16: 73 (doi: 10.1186/s12863-015-0221-0).
- Kantanen J., Edwards C.J., Bradley D.G., Viinalass H., Thessler S., Ivanova Z., Kiselyova T., Ćinkulov M., Popov R., Stojanović S., Ammosov I., Vilkki J. Maternal and paternal genealogy of Eurasian taurine cattle (Bos taurus). Heredity, 2009, 103(5): 404-415 (doi: 10.1038/hdy.2009.68).
- Горелов П.В., Кольцов Д.Н., Зиновьева Н.А., Гладырь Е.А. Сравнительный анализ групп крови и микросателлитов в характеристике новых типов скота бурой швицкой и сычевской пород. Сельскохозяйственная биология, 2011, 6: 37-40.
- Kiseleva T.Yu., Kantanen J., Vorobyov N.I., Podoba B.E., Terletsky V.P. Linkage disequilibrium analysis for microsatellite loci in six cattle breeds. Russian Journal of Genetics, 2014, 50: 406-414 (doi: 10.1134/S1022795414040048).
- Долматова И.Ю., Зиновьева Н.А., Горелов П.В., Ильясов А.Д., Гладырь Е.А., Траспов А.А., Сельцов В.И. Особенности аллелофонда башкирской популяции симментальского скота по микросателлитам. Сельскохозяйственная биология, 2011, 6: 70-74.
- Эрнст Л.К., Бегучев А.П., Левантин Д.Л. Скотоводство. М., 1977.
- Felius M. Cattle breeds — an encyclopedia. Misset, Doetinchem, The Netherlands, 1995.
- Столповский Ю.А., Лазебный О.Е., Столповский К.Ю., Cулимова Г.Е. Применение метода ISSR-PCR для оценки популяционной структуры идентификации и сходства генофондов пород и видов доместицированных животных. Генетика, 2010, 46(6): 1-9.
- Глазко В.И., Феофилов А.В., Бардуков Н.В., Глазко Т.Т. Видоспецифичные ISSR-PCR-маркеры и пути их формирования. Известия Тимирязевской сельскохозяйственной академии, 2012, 1: 118-125.
- Столповский Ю.А., Ахани Азари M., Евсюков А.Н., Кол Н.В., Рузина М.Н., Воронкова В.Н., Cулимова Г.Е. Сравнительный анализ полиморфизма ISSR-маркеров у пород крупного рогатого скота. Генетика, 2011, 47(2): 213-226.
- Столповский Ю.А., Ахани Азари M., Евсюков А.Н., Кол Н.В., Рузина М.Н., Воронкова Л.Н., Cулимова Г.Е. Дифференциация пород крупного рогатого скота с использованием мультилокусного межмикросателлитного анализа (ISSR-PCR). Сельскохозяйственная биология, 2011, 4: 36-45.
- Хлесткина Е.К. Молекулярные маркеры в генетических исследованиях и селекции. Вавиловский журнал генетики и селекции, 2013, 17(4/2): 1044-1054.
- Coates B.S., Sumerford D.V., Miller N.J., Kim K.S., Sappington T.W. Comparative performance of single nucleotide polymorphism and microsatellite markers for population genetic analysis. Journal of Heredity, 2009, 100(5): 556-564 (doi: 10.1093/jhered/esp028).
- Fries R., Durstewitz G. Digital DNA signatures for animal tagging. Nature Biotechnology, 2001, 19(6): 508 (doi: 10.1038/89213).
- Martınez-Arias R., Calafell F., Mateu E., Comas D., Andre´s A., Bertranpetit J. Sequence variability of a human pseudogene. Genome Research, 2001, 11(6): 1071-1085 (doi: 10.1101/gr.167701).
- Xing C., Schumacher F.R., Xing G., Lu Q., Wang T., Elston R.C. Comparison of microsatellites, single-nucleotide polymorphisms (SNPs) and composite markers derived from SNPs in linkage analysis. BMC Genetics, 2005, 6(Suppl. 1): S29 (doi: 10.1186/1471-2156-6-S1-S29).
- Morin P.A., Luikart G., Wayne R.K., Grp S.N.P.W. SNPs in ecology, evolution and conservation. Trends in Ecology & Evolution, 2004, 19(4): 208-216 (doi: 10.1016/j.tree.2004.01.009).
- Vignal A., Milan D., SanCristobal M., Eggen A. A review on SNP and other types of molecular markers and their use in animal genetics. Genetics, Selection, Evolution, 2002, 34(3): 275-305 (doi: 10.1051/gse:2002009).
- Steemers F.J., Gunderson K.L. Whole genome genotyping technologies on the BeadArrayTM platform. Biotechnology Journal, 2007, 2(1): 41-49 (doi: 10.1002/biot.200600213).
- Shen R., Fan J.B., Campbell D., Chang W., Chen J., Doucet D., Yeakley J., Bibikova M., Garcia E.W., McBride C., Steemers F., Garcia F., Kermani B.G., Gunderson K., Oliphant A. High-throughput SNP genotyping on universal bead arrays. Mutation Research, 2005, 573(1-2): 70-82 (doi: 10.1016/j.mrfmmm.2004.07.022).
- Decker J.E., Pires J.C., Contant G.C., McKay S.D., Heaton M.P., Chen K., Cooper A., Vilkki J., Seabury C.M., Caetano A.R., Johnson G.S., Brenneman R.A., Hanotte O., Eggert L.S., Wiener P., Kim J.-J., Kim K.S., Sonstegard T.S., Van Tassell C.P., Neibergs H.L., McEwan J.C., Brauning R., Coutinho L.L., Babar M.E., Wilson G.A., McClure M.C., Rolf M.M., Kim J.W., Schnabel R.D., Taylor J.F. Resolving the evolution of extant and extinct ruminants with high-throughput phylogenomics. PNAS USA, 2009, 106(44): 18644-18649 (doi: 10.1073/pnas.0904691106).
- Kuehn L.A., Keele J.W., Bennett G.L., McDaneld T.G., Smith T.P., Snelling W.M., Sonstegard T.S., Thallman R.M. Predicting breed composition using breed frequencies of 50,000 markers from the US Meat Animal Research Center 2,000 Bull Project. J. Anim. Sci., 2011, 89(6): 1742-1750 (doi: 10.2527/jas.2010-3530).
- McTavish E.J., Decker J.E., Schnabel R.D., Taylor J.F., Hillis D.M. New World cattle show ancestry from multiple independent domestication events. PNAS USA, 2013, 110(15): 1398-1406 (doi: 10.1073/pnas.1303367110).
- Decker J.E., McKay S.D., Rolf M.M., Kim J., Molina Alcalá A., Sonstegard T.S., Hanotte O., Götherström A., Seabury C.M., Praharani L., Babar M.E., Regitano L.C.A., Yildiz M.A., Heaton M.P., Wan-Sheng L., Lei C.-Z., Reecy J.M., Saif-Ur-Rehman M., Schnabel R.D., Taylor J.F. Worldwide patterns of ancestry, divergence, and admixture in domesticated cattle. PLoS Genet., 2014, 10(3): e1004254 (doi: 10.1371/journal.pgen.1004254).
- Decker J.E., Taylor J.F., Cronin M.A., Alexander L.J., Kantanen J., Millbrooke A., Schnabel R.D., MacNeil M.D. Origins of cattle on Chirikof Island, Alaska elucidated from genome-wide SNP genotypes. Journal of Heredity, 2016, 116(6): 502-505 (doi: 10.1038/hdy.2016.7).
- Iso-Touru T., Tapio M., Vilkki J., Kiseleva T., Ammosov I., Ivanova Z., Popov R., Ozerov M., Kantanen J. Genetic diversity and genomic signatures of selection among cattle breeds from Siberia, eastern and northern Europe. Animal Genetics, 2016, 47(6): 647-657 (doi: 10.1111/age.12473).
- Zinovieva N.A., Dotsev A.V., Sermyagin A.A., Wimmers K., Reyer H., Sölkner J., Deniskova T.E., Brem G. Study of genetic diversity and population structure of five Russian cattle breeds using whole genome SNP analysis. Sel’skokhozyaistvennaya biologiya, 2016, 51(6): 788-800 (doi: 10.15389/agrobiology.2016.6.788eng).
- Yurchenko A., Yudin N., Aitnazarov R., Plyusnina A., Brukhin V., Soloshenko V., Lhasaranov B., Popov R., Paronyan I.A., Plemyashov K.V., Larkin D.M. Genome-wide genotyping uncovers genetic profiles and history of the Russian cattle breeds. Heredity, 2018, 120(2): 125-137 (doi: 10.1038/s41437-017-0024-3).
- Sermyagin A.A., Dotsev A.V., Gladyr E.A., Traspov A.A., Deniskova T.E., Kostjunina O.V., Reyer H., Wimmers K., Barbato M., Paronyan I.A., Plemyashov K.V., Sölkner J., Popov R.G., Brem G., Zinovieva N.A. Whole-genome SNP analysis elucidates the genetic structure of Russian cattle and its relationship with Eurasian taurine breeds. Genetics, Selection, Evolution, 2018, 50: 37 (doi: 10.1186/s12711-018-0408-8).
- Катмаков П.С., Гавриленко В.П., Бушов А.В., Стенькин Н.И. Совершенствование генофонда бестужевской породы с использованием потенциала голштинской и красных пород европейской селекции. Вестник Ульяновской ГСХА, 2014, 1(25): 126-132.
- Кертиев Р.М., Пархоменко Л.А., Никулкин Н.С., Высоцкая В.М., Пархоменко Л.Б. Программа разведения и совершенствования крупного рогатого скота холмогорской породы. Зоотехния, 2016, 2: 14-15.
- Прожерин В.П., Ялуга В.Л. Использование национальных племенных ресурсов молочного скота. Зоотехния, 2017, 7: 6-9.
- Кавардакова О., Кузнецов В. Результаты голштинизации черно-пестрого скота Пермского края. Молочное и мясное скотоводство, 2007, 7: 37-38.
- Быданцева Е., Кавардакова О. Зависимость продуктивного долголетия коров от генетических факторов. Молочное и мясное скотоводство, 2012, 3: 17-18.
- Сулимова Г.Е., Лазебная И.В., Перчун А.В., Воронкова В.Н., Рузина М.Н., Бадин Г.А. Уникальность костромской породы крупного рогатого скота с позиции молекулярной генетики. Достижения науки и техники АПК, 2011, 9: 52-54.
- Казаков Д.С., Белокуров С.Г. Влияние быков-производителей разной селекции на продуктивное долголетие коров костромской породы. Вестник биотехнологии, 2017, 2: 11.
- Новиков В.М., Кольцов Д.Н., Цысь В.И., Леутина Д.В., Татуева О.В. Проблемные вопросы крупномасштабной селекции бурой швицкой породы крупного рогатого скота. Генетика и разведение животных, 2016, 1: 46-51.
- Специализированный портал «Быки-производители». Режим доступа: http://testfb.pli-nor.ru/plem/stat. Дата обращения: 17.03.2018.
- Moser C., Reiter M. Die Rinderrasse der Tux-Zillertaler — Ein Stück Tiroler Kultur. Verlag Edition Tirol, 1996.
- Tux Zillertaler. Режим доступа: http://en.zar.at/Cattle_breeding_in_Austria/Cattle_breeds/ Additional_Breeds/Tux_Zillertaler.html. Дата обращения: 18.03.2018.
- Лискун Е.Ф. Отечественные породы крупного рогатого скота. М., 1949.
- Боронецкая О.И., Чикурова Е.А., Никифоров А.И. Возникновение, особенности и породообразования, и практика сохранения белого паркового скота. Известия ТСХА, 2017, 6: 68-84 (doi: 10.26897/0021-342X-2017-6-68-84).
- Браунер А. Породы сельскохозяйственных животных. Крупный рогатый скот. Одесса, 1922.
- Лискун Е.Ф. Методика краниологических исследований. Доклад, сделанный на XII съезде естествоиспытателей и врачей. С.-Петербург, 1910. В кн.: Избранные труды /Под ред. Е.А. Арзуманяна. М., 1961: 42-75.
- Боронецкая О.И., Барбосова М.Е., Никифоров А.И., Быкова А.В., Михеенков В.Е., Рабаданова Г.Ш., Петрикеева Л.В., Полуротова А.И., Рукавицина Е.А. Каталог краниологической коллекции академика Е.Ф. Лискуна /Под ред. В.П. Панова. М., 2012.
- MacHugh D.E., Edwards C.J., Bailey J.F., Bancroft D.R., Bradley D.G. The extraction and analysis of ancient DNA from bone and teeth: a survey of current methodologies. Ancient Biomolecules, 2000, 3(2): 81-102.
- Beja-Pereira A., Caramelli D., Lalueza-Fox C., Vernesi C., Ferrand N., Casoli A., Goyache F., Royo L.J., Conti S., Lari M., Martini A., Ouragh L., Magid A., Atash A., Zsolnai A., Boscato P., Triantaphylidis C., Ploumi K., Sineo L., Mallegni F., Taberlet P., Erhardt G., Sampietro L., Bertranpetit J., Barbujani G., Luikart G., Bertorelle G. The origin of European cattle: evidence from modern and ancient DNA. PNASUSA, 2006, 103(21): 8113-8118 (doi: 10.1073/pnas.0509210103).
- Gargani M., Pariset L., Lenstra J.A., De Minicis E., European Cattle Genetic Diversity Consortium, Valentini A. Microsatellite genotyping of medieval cattle from central Italy suggests an old origin of Chianina and Romagnola cattle. Frontiers in Genetics, 2015, 6: Article 68 (doi: 10.3389/fgene.2015.00068).