УДК 635.649:631.523:57.088.1
МОЛЕКУЛЯРНОЕ МАРКИРОВАНИЕ ЛИНИЙ И ПОЛУЧЕННЫХ НА ИХ ОСНОВЕ СОРТОВ ПЕРЦА СЛАДКОГО Capsicum annuum L.
Н.Н. РЫЖОВА1, Е.З. КОЧИЕВА1, О.Н. ПЫШНАЯ2
На образцах перца сладкого Capsicum annum L. (три линии — Л-Золотое Чудо, Л-Беке, Л-Артек, а также полученные на их основе сорта Снегирь и Янтарь) с использованием подобранных праймеров впервые исследовали возможность применения мультилокусного RAPD-метода для анализа характера наследования родительского генетического материала при создании сортов и установления их генетической природы. По результатам сравнения ДНК-спектров оценен генетический полиморфизм и идентифицированы ДНК-маркеры, потенциально пригодные для генотипирования исследованных образцов C. annuum, показана рекомбинантная природа сорта Снегирь и подтверждено высокое сходство сорта Янтарь с родительской линией Л-Артек.
Ключевые слова: сорта перца, гибриды, молекулярные ДНК-маркеры, мультилокусный анализ, генетическое разнообразие, селекция перца.
Как известно, успех в селекции определяется не только правильным подбором родительских компонентов, выбором способа гибридизации и отбора, но и во многом зависит от того, смогут ли родительские формы обеспечить комбинирование желательных признаков, то есть генетической природой исходного материала (линий и получаемых на их основе гибридных растений) (1-2). Популяция, сформированная плановой гибридизацией сортов и линий, представляет собой генетически наиболее ценный материал для получения новых сортов и исходных форм. Отбор в гибридной популяции начинается с выявления гомозиготных и гетерозиготных растений, которые в результате оплодотворения гамет растений F1 проявляются в F2 (3).
Оценить структуру гибридной популяции и идентифицировать потенциально ценные генотипы помогают современные высокоточные и надежные системы молекулярного ДНК-маркирования (так называемая маркер-вспомогательная селекция — marker assisted selection, MAS), позволяющие значительно ускорить и упростить процедуру отбора нужного генотипа и его последующего испытания на селективном фоне (4).
Разработка и использование систем ДНК-маркирования в селекционной практике особенно актуальны для овощных культур, поскольку белковые маркеры, эффективные при селекции злаковых, у большинства овощных не могут считаться надежными: вследствие особенностей регуляции экспрессия запасных белков у них весьма зависима от внешних факторов (4-5).
Для разработки системы ДНК-маркеров может быть использован любой из современных методов молекулярной идентификации генотипов на основе ПЦР-технологии, в том числе AFLP (amplified fragment length polymorphism), DNA-RAPD (random amplification of polymorphic dna — метод произвольно амплифицированной полиморфной ДНК), ISSR (inter simple sequence repeats), SSAP (sequence-specific amplification polymorphisms), SSR (simple sequence repeats), SNP (single nucleotide polymorphism) и ряд других. RAPD-метод — один из наиболее простых и доступных. Он позволяет исследовать главным образом селективно нейтральные уникальные и умеренно повторяющиеся последовательности генома растений (6). Метод широко используется для сортовой идентификации, оценки межсортового полиморфизма, сортовой чистоты, биотипического состава сортов, а также для анализа гибридных популяций, контроля гибридизации и интрогрессии генетического материала при сортовых, линейных и межвидовых скрещиваниях (5, 7-15).
Цель настоящей работы заключалась в изучении возможности использования мультилокусного RAPD-метода для установления генетической природы сортов перца и анализа характера наследования генетического материала родительских линий в созданных сортах. В этой связи были поставлены следующие задачи: подбор праймеров, выявляющих полиморфизм родительских линий и производных сортов; маркирование родительских линий и производных сортов; оценка молекулярно-генетического полиморфизма исследованных образцов; идентификация ДНК-маркеров, потенциально пригодных для генотипирования изученных и других линий и сортов Capsicum аnnum L.
Методика. Объектом исследования служили три родительские линии перца C. annuum: Л-Золотое Чудо, Л-Беке, Л-Артек, а также Снегирь и Янтарь,полученные в результате простых скрещиваний соответственно Л-Золотое Чудо x Л-Беке и Л-Золотое Чудо x Л-Артек с последующим отбором из гибридной популяции методом педигри.
ДНК для анализа выделяли из 8-10-суточных проростков по стандартной методике (16) с дополнительной депротеинизацией смесью фенола c хлороформом. Для ПЦР-анализа использовали 20 праймеров серий OPA, OPD, OPH, OPN («Operon Technologies», США). Амплификацию проводили в реакционной смеси объемом 15 мкл, содержащей 2,5 мМ MgCl2, 0,2 мМ каждого dNTP, 0,5 мкМ праймера, 0,3 ед. Taq-полимеразы, 1x буфер из соответствующего набора («Диалат ЛТД», Россия) и 100 нг геномной ДНК, в термоциклере GeneAmp PCR System2700 («Applied Biosystems», США) в следующем режиме: денатурация — 30 с при 94 °С, отжиг праймера — 45 с при 37 °С, синтез ДНК — 1 мин при 72 °С (число циклов — 36); предварительная денатурация — 5 мин при 94 °С; заключительная элонгация — 10 мин при 72 °С.
Все реакции выполняли в двух повторностях. Продукты амплификации разделяли электрофорезом в 1,7 % агарозном геле (high resolution, «Sigma», MetaPhor, «Cambrex», США) в 1хТВЕ-буфере с последующим окрашиванием бромистым этидием.
Учитывали только четкие, воспроизводимые фрагменты размером от 300 до 2000 п.н. Полиморфизм амплифицированных фрагментов генома оценивали как отношение числа полиморфных фрагментов к общему числу полученных фрагментов, выраженное в процентах.
Результаты. С целью выявления генетических различий между родительскими формами протестировали 20 праймеров серий OPA, OPD, OPH, OPN, из которых только два (OPE1 и OPE7) позволили получить четкие воспроизводимые ДНК-спектры продуктов амплификации, содержащие специфичные фрагменты. Всего получили 34 RAPD-фрагмента ДНК, из которых 16 (47 %) были полиморфными. Длина амплифицированных RAPD-фрагментов варьировала в пределах 340-1650 п.н. При этом для пары Л-Беке—Л-Золотое Чудо фрагменты OPE11550 (A), OPE11280(B), OPE1530 (E), OPE1385 (H), OPE71150 (Aґ), OPE7800 (Bґ), OPE7720(Cґ), OPE7410(Gґ) оказались специфичными маркерами линии Л-Беке (рис.), фрагменты OPE1600 (C), OPE1550 (D), OPE1490 (F), OPE1410 (G), OPE1380 (I), OPE7630 (Dґ), OPE7410 (Gґ) — маркерами линии Л-Золотое Чудо. Соответственно для родительской пары Л-Золотое Чудо—Л-Артек фрагменты OPE1600 (C), OPE1490 (F), OPE1410 (G), OPE1380 (I), OPE7630 (Dґ) маркировали линию Л-Золотое Чудо. Для линии Л-Артек специфичных ампликонов не выявили.
Оба эти праймера (OPE1 и OPE7) использовали для последующего маркирования сортов Снегирь и Янтарь, созданных при межлинейных скрещиваниях, установления их генетической природы и проведения сравнительного мультилокусного RAPD-анализ всех пяти образцов перца.
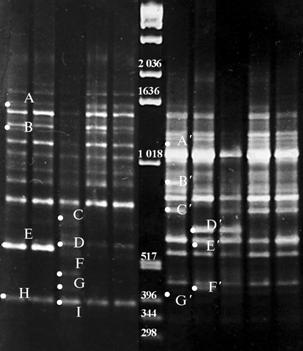 |
RAPD-спектры ДНК родительских линий перца сладкогоCapsicumannumL. Л-Беке, Л-Золотое Чудо, Л-Артек (соответственно 1, 2 и 3), а также созданных на их основе сортов Снегирь и Янтарь (соответственно 4 и 5), полученные с использованием праймеров OPE1 и OPE7 (соответственно слева и справа от маркера молекулярных масс) (М, 1 kb Ladder, «Gibco BRL», США). |
При анализе ДНК линий Л-Беке, Л-Золотое Чудои полученного на их основе сорта Снегирь оба праймера (OPE1 и OPE7) позволили детектировать различия между тремя образцами. Интересно, что в варианте с использованием праймера OPE1 RAPD-спектр у сорта Снегирьоказался более сходным со спект-ром линии Л-Беке, содержал специфичные для Л-Бекефрагменты OPE1530 (E), OPE1385 (H) и не имел ни одного фрагмента, спе-цифичного для родительской формы Л-Золотое Чудо.При использованиипраймера OPE7 RAPD-спектр у сортаСнегирь, наоборот,был похож на спектр у линии Л-Золотое Чудо и содержал ее маркерный фрагмент OPE7630 (Dґ), в то время как маркерные фрагменты линии Л-Беке OPE71150 (Aґ), OPE7800(Bґ), OPE7720 (Cґ), OPE7410 (Gґ) отсутствовали. Кроме того, следует отметить, что сорт Снегирь характеризовался наличием в RAPD-спектре своих собственных специфичных фрагментов — OPE7580 (Eґ) и OPE7430(Fґ), которые отсутствовали у исходных форм, и не содержал ряд фрагментов, имевшихся в спектрах обеих родительских линий (см. рис.), что может быть связано с рекомбинационными процессами.
Необходимо отметить, что сорта Снегирь и Янтарь были получены индивидуальным отбором растений в расщепляющихся поколениях с прослеживанием родословной отобранных растений до формирования гомозиготных линий. Известно, что расщепление по признакам происходит в потомстве каждого растения и именно поколение F2 характеризуется наибольшим числом комбинаций признаков в результате генетической рекомбинации. В процессе селекции в поколении F2 мы отбирали генотипы, которые, судя по фенотипу, содержат комбинации признаков родителей, уделяя особое внимание хозяйственно ценным. В F3 проводили отбор растений с желательными рекомбинациями признаков, причем прежде всего в потомстве, в наибольшей мере отвечающем селекционным целями, так как именно в F3 начинается формирование линий. При создании сорта Снегирь отбор из гибридной популяции по фенотипу осуществляли с учетом признаков, проявившихся только у гибридных растений, и некоторых желательных свойств, присущих родительским формам. Все это подтверждает наше предположение о рекомбинантной природе сорта Снегирь.
| Размер полиморфных RAPD-фрагментов в спектрах, полученных с праймерами OPE1 (A—I) и OPE7 (Aґ—Gґ), у изученных образцов перца сладкогоCapsicumannumL. | |||||||||
Линия, сорт |
A/Aґ |
B/Bґ |
C/Cґ |
D/Dґ |
E/Eґ |
F/Fґ |
G/Gґ |
H |
I |
П р а й м е р OPE1 |
|||||||||
Л-Беке |
1550 |
1280 |
|
|
530 |
|
|
385 |
|
Снегирь |
|
|
|
|
530 |
|
|
385 |
|
Л-Золотое Чудо |
|
|
600 |
550 |
|
490 |
410 |
|
380 |
Янтарь |
1550 |
1280 |
|
550 |
|
|
|
385 |
|
Л-Артек |
1550 |
1280 |
|
550 |
|
|
|
385 |
|
П р а й м е р OPE7 |
|||||||||
Л-Беке |
1150 |
800 |
720 |
|
|
|
410 |
|
|
Снегирь |
|
|
|
630 |
580 |
430 |
|
|
|
Л-Золотое Чудо |
|
|
|
630 |
|
|
410 |
|
|
Янтарь |
1150 |
|
720 |
|
|
|
410 |
|
|
Л-Артек |
1150 |
|
720 |
|
|
|
410 |
|
|
При сравнительном анализе RAPD-спектров родительских форм Л-ЗолотоеЧудо,Л-Артек и сорта Янтарь у последнего он оказался полностью похожим на таковой отцовского компонента (линия Л-Артек) и не содержал ни одного фрагмента, специфичного для материнской формы — линии Л-Золотое Чудо (OPE1600 — C, OPE1490 — F, OPE1410 — G, OPE1380 — I, OPE7630 — Dґ, OPE7410 — Gґ). Примечательно, что по фенотипу (габитус, форма и размер листа, форма плода, расположение плода и др.) растения сорта Янтарь действительно полностью повторяют отцовскую форму Л-Артек, за исключением признаков скороспелости и окраски плода, унаследованных от материнской линии. Таким образом, с использованием RAPD-метода выполнено молекулярное маркирование пяти образцов перца сладкого Capsicum annum L. (три линии и два полученных на их основе сорта). Подобраны RAPD-праймеры, позволяющие детектировать межсортовой полиморфизм культуры, выявлены специфичные маркерные RAPD-фрагменты образцов и показана возможность использования RAPD-маркеров для анализа гибридных комбинаций и детекции рекомбинационных процессов.
Л И Т Е Р А Т У Р А
1. М а м е д о в М.И., П ы ш н а я О.Н., Д ж о с Е.А. Селекция перца сладкого для открытого грунта Нечерноземья. Картофель и овощи, 2005, 4: 14.
2. М а м е д о в М.И., П и в о в а р о в В.Ф. Селекция томата, перца и баклажана на адаптивность. М., 2002.
3. Б у н и н М.С., М а м е д о в М.И., Ш м ы к о в а Н.А., С у п р у н о в а Т.П., Е н-
г а л ы ч е в а И.А., К о ч и е в а Е.З., Р ы ж о в а Н.Н., Д ж о с Е.А.. Межвидовая гибридизация в роде Capsicum L. и ее использование в селекции. Методика. М., 2008.
4. L e f e b v r e V. Molecular markers for genetics and breeding: development and use in pepper (Capscum spp.). In: Molecular marker systems in plant breeding and crop improvement (Н. Lörz, G. Wenzel, eds.) — Biotechnology in Agriculture and Forestry, 2004, 55: 189-214.
5. К о ч и е в а Е.З. Использование методов на основе полимеразной цепной реакции для маркирования генома растений. С.-х. биол., 1999, 1: 1-19.
6. W i l l i a m s J.G.K., K u b e l i k A.R., L i v a k K.J., R a f a l s k i J.A., T i n g e y S.V. DNA polymorphisms amplified by arbitrary primers are useful as genetic markers. Nucl. Acids Res., 1990, 18: 6531-6535.
7. C r o c k e t t P.A., B h a l l a P.L., L e e C.K., S i n g h M.B. RAPD analysis of seed purity in a commercial hybrid cabbage (Brassica oleracea var. capitata) cultivar. Genome, 2000, 43: 317-321.
8. I l b i H. RAPD markers assisted varietal identification and genetic purity test in pepper, Capsicum annuum. Scientia Horticulturae, 2003, 97(4): 211-218.
9. S u n G., W a n g - P r u s k i G., M a y i c h M., J o n g H. RAPD and pedigree-based genetic diversity estimates in cultivated diploid potato hybrids. Theor. Appl. Genet., 2003, 107: 110-115.
10. K a r i h a l o o J.L., D w i v e d i Y.K, G a i k w a d A.B. Analysis of genetic diversity of Indian mango cultivars using RAPD markers. J. Hortic. Sci. Biotechnol, 2003, 78(3): 285-289.
11. S i n g h N., S i n g h M., K u m a r S. e.a. RAPD markers for hybrid seed purity testing in tomato (Solanum lycopersicum L.). Current Science, 2007, 93(4): 462-463.
12. C h u n d e t R., C u t l e r R.W., T a s a n o n M.S. e.a. Hybrid detection in Lychee (Litchee chinensis Sonn.) cultivars using HAT-RAPD markers. ScienceAsia, 2007, 33: 307-311.
13. О n t o S., L a o s a t N., S u k s a w a t W., P o p l u e c h a i S.,
E u n g w a n i c h a
y a p a n t P.D., C h u k e a t i r o t e E. Phylogenetic analysis of Cucumis sativus using RAPD molecular markers. J. Plant Sci., 2008, 3(1): 105-110.
14. E r c i s l i S., O r h a n E., H i z a r c i Y., Y i l d i r i m N., A g a r G. Genetic diversity in grapevine germplasm resources in the Coruh Valley revealed by RAPD markers. Biochem. Genet., 2008, 46: 590-597.
15. T h a w a r o S., T e - С h a t o S. RAPD (random amplified polymorphic DNA) marker as a tool for hybrid oil palm verification from half mature zygotic embryo culture. J. Agricult. Technol., 2008, 4(2): 165-176
16. E d w a r d s K., J o h n s t o n e C., T h o m p s o n C. A simple and rapid method for the preparation of plant genomic DNA for PCR analysis. Nucl. Acids Res., 1991, 19(6): 1349.
MOLECULAR LABELING OF THE LINES AND OBTAINED ON THEIR BASIS THE VARIETIES OF Capsicum annuum L.
N.N. Ryzhova1, E.Z. Kochieva1, O.N. Pyshnaya2
On the variants of Capsicum annum L. (the L-Zolotoe Chudo, L-Beke, L-Artek lines and obtained of their basis the Snegir’ and Yantar’ varieties) with the use of appropriate primers the authors for the first time investigated the multipoint RAPD method in respect to the analysis of inheritance of parent genetic material during the varieties obtaining and establishing of their genetic nature. As result of DNA-spectra comparison the genetic polymorphism was estimated and the DNA-markers potential suitable for genetic typing of investigated variants of C. annuum were identified, the recombinant nature of the Snegir’ variety was shown and the high similarity of the Yantar’ variety with L-Artek parent line was confirmed.
Key words: pepper cultivars, hybrids, molecular DNA-markers, multiloci analysis, genetic diversity, pepper breeding.
1Центр «Биоинженерия» РАН, |
Поступила в редакцию |













