doi: 10.15389/agrobiology.2019.2.359ru
УДК 619:578:616.5:57.083.2:577.2
Работа выполнена за счет средств гранта Президента Российской Федерации для государственной поддержки молодых российских ученых № МК-2090.2018.11.
ТЕСТ-СИСТЕМА ДЛЯ ВЫЯВЛЕНИЯ ГЕНОМА ВИРУСА ЧУМЫ МЕЛКИХ ЖВАЧНЫХ ЖИВОТНЫХ МЕТОДОМ ОТ-ПЦР В РЕАЛЬНОМ ВРЕМЕНИ
Н.И. САЛЬНИКОВ, С.П. ЖИВОДЕРОВ, Д.В. ЯНЖИЕВА, А.Ю. КОЛЬЦОВ, Т.Р. УСАДОВ, М.М. СУХЕР,
Ю.П. МОРГУНОВ, А.В. ЛУНИЦИН
Чума мелких жвачных животных (ЧМЖЖ) — высококонтагиозная трансграничная инфекционная болезнь мелких жвачных, характеризующаяся лихорадкой, анорексией, истечениями из глаз и носа, эрозиями и язвами в слизистой оболочке пищеварительного тракта, диареей, а также лейкопенией с иммуносупрессией. Ввиду сложной эпизоотической обстановки по ЧМЖЖ в сопредельных с Российской Федерацией государствах (Таджикистан, Китай, Монголия, Казахстан, Афганистан) существует высокий риск проникновения данного заболевания на территории Сибирского, Уральского, Дальневосточного, Северо-Кавказского и Южного федеральных округов. В связи с этим необходимы методы молекулярно-генетической диагностики возбудителя ЧМЖЖ, а также способы стабилизации образцов биоматериала для диагностических исследований при транспортировке на дальние расстояния. Нами представлены данные по разработке тест-системы для выявления генома вируса ЧМЖЖ методом ОТ-ПЦР в реальном времени. Методика основана на амплификации фрагмента гена гемагглютинина размером 148 п.н. с использованием оригинальных олигонуклеотидных праймеров, а также флуоресцентно меченного гибридизационного зонда. Аналитическую специфичность разработанной тест-системы оценивали при исследовании панели образцов биоматериала, содержащих штаммы вируса ЧМЖЖ, чумы крупного рогатого скота, блютанга, а также образцов биоматериала от клинически здоровых животных и интактных культур клеток. При этом положительные результаты получены только при исследовании образцов, содержащих штаммы вируса ЧМЖЖ (Эпизоотический, Nigeria 75/1 и 45G37/35-K). Аналитическая чувствительность разработанной тест-системы, определенная с использованием последовательных 10-кратных разведений культурального вирусосодержащего материала, составила 0,83±0,22 lg ТЦД50/см3. Положительным контролем амплификации в разработанной тест-системе служил фрагмент гена гемагглютинина вируса ЧМЖЖ, который клонировали в составе плазмидного вектора pTZ57 R/T в клетках Escherichia coli. Установлено, что линейность амплификации в диапазоне концентраций плазмидной ДНК от 2,4×107 до 24 молекул в 1 мкл. Для оценки практической пригодности разработанной тест-системы исследовали образцы крови, отобранные от овец, экспериментально инфицированных штаммом Эпизоотический вируса ЧМЖЖ. В результате геном вируса ЧМЖЖ был выявлен в пробах крови, отобранных с 5-х по 12-е сут после заражения. Для транспортировки образцов биоматериала на дальние расстояния нами предложена методика «сухой» капли для отбора и хранения проб крови на бумажном носителе. Показано, что такие препараты стабильны при комнатной температуре в течение 1 мес и могут быть использованы для исследований методом ОТ-ПЦР в реальном времени.
Ключевые слова: чума мелких жвачных животных, вирус, культура клеток, обратная транскрипция, ПЦР, рекомбинантная плазмида, экспериментальная инфекция.
Чума мелких жвачных животных (ЧМЖЖ) — эндемичная во многих странах Африки, Ближнего Востока, Азии высококонтагиозная трансграничная инфекционная болезнь мелких жвачных, которая представляет серьезную угрозу для животноводства этих стран. Искоренение ЧМЖЖ рассматривают как необходимый компонент глобальной продовольственной безопасности и борьбы с нищетой (1, 2). ЧМЖЖ характеризуется лихорадкой, анорексией, истечениями из глаз и носа, эрозиями и язвами в слизистой оболочке пищеварительного тракта, диареей, а также лейкопенией с иммуносупрессией. У суягных овец и сукотных коз могут наблюдаться аборты (3, 4). Гибель больных животных возможна вследствие непосредственного поражения организма вирусом ЧМЖЖ, снижения иммунитета и размножения других патогенов (например, Pasteurella spp., Escherichia coli, Mycoplasma spp.). Показатели смертности значительно варьируют (от 10 до 90 %) в зависимости от вида инфицированных животных, породы, их возраста и распространенности вторичных инфекционных агентов (5).
Возбудитель болезни — РНК-содержащий вирус семейства Paramyxoviridae, рода Morbillivirus (6, 7), который антигенно родствен вирусу чумы крупного рогатого скота (ЧКРС), также относящемуся к роду Morbillivirus (8). Геном вируса ЧМЖЖ представлен одноцепочечной молекулой РНК негативной полярности размером 15948 нуклеотидов. Геномная РНК вируса ЧМЖЖ содержит открытые рамки считывания для шести структурных белков (нуклеокапсидный, фосфопротеин, матриксный, белок слияния и гемагглютинин) и двух неструктурных белков (V и C) (9). Штаммы и изоляты вируса ЧМЖЖ не подразделяются на серотипы, но на основе нуклеотидных последовательностей генов нуклеокапсидного белка и фосфопротеина сгруппированы в четыре генетические линии (10). Установлено, что штаммы и изоляты вируса ЧМЖЖ, относящиеся к I и II линиям, циркулируют в Западной Африке, к линии III — в Восточной Африке, Ближнем Востоке и на юге Индии, к линии IV — в Азии (11, 12).
Первоначально ЧМЖЖ отмечали в странах Западной Африки, затем инфекция проникла в страны Центральной и Восточной Африки, а с 1980 года и на Аравийский полуостров. В настоящее время это заболевание широко распространено на Африканском континенте и в Азии. Кроме того, это заболевание зарегистрировано в сопредельных с Российской Федерацией государствах — Турции, Афганистане, Казахстане, Таджикистане, Монголии, Китае и Грузии. Анализ эпизоотической ситуации по ЧМЖЖ в России показал, что существует высокий риск проникновения инфекции на территории Сибирского, Уральского, Дальневосточного, Северо-Кавказского и Южного федеральных округов (13, 14).
Согласно рекомендациям Международного эпизоотического бюро (МЭБ, Office International des Epizooties, OIE, Франция), диагноз ЧМЖЖ может быть поставлен при выявлении вирусного генома методом полимеразной цепной реакции (ОТ-ПЦР), выделении вируса с использованием пермиссивной линии клеток, а также при обнаружении антигенов вируса методами твердофазного иммуноферментного анализа (ИФА) и флуоресцирующих антител (МФА), в реакции иммунодиффузии в агарозном геле и в иммуноэлектрофорезе. Серологические методы выявления специфических антител (реакция нейтрализации вируса и конкурентный вариант ИФА) используются при проведении мониторинговых исследований и определении иммунного статуса животного после проведенной вакцинации. МЭБ отмечает, что методы ОТ-ПЦР и ИФА (для выявления вирусных антигенов) наиболее приемлемы для подтверждения клинических случаев болезни (15). Наиболее широко для диагностики ЧМЖЖ применяется ОТ-ПЦР с электрофоретической или гибридизационно-флуоресцентной детекцией продуктов амплификации (ОТ-ПЦР в реальном времени). Хорошо себя зарекомендовали и давно используются протоколы ОТ-ПЦР с электрофоретической детекцией, предложенные M.S. Shaila с соавт. в 1996 года (16) и E. Couacy-Hymann с соавт. в 2002 году (17), основанные соответственно на амплификации фрагментов генов нуклеокапсидного белка и фосфопротеина. В дальнейшем J. Bao с соавт. (18), O. Kwiatek с соавт. (19) и C.A. Batten с соавт. (20) предложили протоколы для высокопроизводительного и более чувствительного метода ОТ-ПЦР в реальном времени на основе амплификации высококонсервативных участков гена нуклеокапсидного белка. Эти тест-системы обладают аналитической чувствительностью от 30 до 10 копий РНК на реакцию и позволяют выявлять геномы штаммов вируса ЧМЖЖ, принадлежащих ко всем четырем линиям (18-20). V. Balamurugan с соавт. (21) опубликовал в 2010 году протокол ОТ-ПЦР в реальном времени, в которой амплифицируется фрагмент гена матриксного белка с чувствительностью в пределах 0,1-1,0 ТЦД50/см3.
При проведении диагностических исследований на наличие генома вируса ЧМЖЖ от животных отбирают смывы с конъюнктивы, носовой и ротовой полостей, а также образцы крови. Для предотвращения неспецифической деградации нуклеиновой кислоты вируса биологический материал необходимо транспортировать в охлажденном виде (22). Если нет возможности соблюдать температурный режим при доставке образцов в диагностическую лабораторию, рекомендуют использовать бумажный носитель для стабилизации образцов крови (методика «сухой» капли крови). Метод сухих пятен широко применяют в ветеринарии и медицине для взятия в домашних условиях и доставки в лаборатории проб различных биологических жидкостей для вирусологических, серологических и биохимических анализов (23, 24).
Несмотря на то, что молекулярно-генетические методы широко используются в ветеринарной практике для диагностики ЧМЖЖ, необходимо наличие подтверждающих тестов, основанных на применении альтернативных генов-мишеней. Такие тесты должны быть валидированы в соответствии со стандартами МЭБ и выявлять геном вируса ЧМЖЖ в пробах биоматериала от инфицированных животных.
В настоящем сообщении мы представляем результаты разработки тест-системы для обнаружения генома вируса ЧМЖЖ, основанной на амплификации фрагмента гена гемагглютинина с использованием методом ОТ-ПЦР в реальном времени. Эта тест-система обладает чувствительностью 0,83±0,22 lg ТЦД50/см3 и позволяет дифференцировать геном вируса ЧМЖЖ от геномов вирусов чумы крупного рогатого скота и блютанга, вызывающих у овец сходные симптомы. Экспериментально подтверждена возможность практической диагностики ЧМЖЖ с помощью предложенной тест-системы.
Нашей целью была разработка на основе ОТ-ПЦР в реальном времени тест-системы для выявления генома вируса ЧМЖЖ и ее валидация с использованием проб крови от экспериментально инфицированных овец.
Методика. Использованные штаммы вируса ЧМЖЖ (Nigeria 75/1, Эпизоотический и 45G37/35-K), чумы крупного рогатого скота (вакцинные штаммы Накамура и ЛТ 67) и блютанга (VIII серотип, штамм NET 2007), депонированы в Государственной коллекции микроорганизмов ФГБНУ ФИЦВиМ. В качестве отрицательных образцов при определении аналитической специфичности тест-системы использовали культуры клеток из коллекции культур клеток ФГБНУ ФИЦВиМ.
Вирусную РНК выделяли с помощью набора РНК-Сорб (ООО «Интерлабсервис», Россия), реакцию обратной транскрипции проводили на термоциклере Терцикл (ЗАО «ДНК-Технологии», Россия), ПЦР в реальном времени — на детектирующем амплификаторе ДТ-Прайм (ЗАО «ДНК-Технологии», Россия). Для синтеза комплементарной ДНК на матрице выделенных образцов РНК использовали реакционную смесь объемом 20 мкл, содержащую 10 мкл исследуемого образца, 1 мкл обратного праймера (10 пмоль/мкл, ЗАО «Евроген», Россия), 4 мкл 5× буфера для обратной транскрипции (ООО «Альфа-фермент», Россия), 0,3 мкл смеси дезоксинуклеозидтрифосфатов (10 ммоль/мл, ЗАО «Синтол», Россия), 0,2 мкл ревертазы (200 ед/мкл, ООО «Альфа-фермент», Россия) и 4,5 мкл бидистиллированной воды. Реакционную смесь инкубировали в течение 30 мин при температуре 42 °С. ПЦР в реальном времени с использованием полученной комплементарной ДНК проводили в реакционной смеси объемом 25 мкл, содержащей 5 мкл матрицы, по 1 мкл прямого и обратного праймеров и 0,3 мкл зонда Taq-man (10 пмоль/мкл), 2,5 мкл 10½ буфера для ПЦР (ООО «Альфа-фермент», Россия), 0,3 мкл смеси дезоксинуклеозидтрифосфатов (10 ммоль/мл), 0,5 мкл MgCl2 (25 ммоль/мл, ЗАО «Синтол», Россия), 0,1 мкл Taq MS-полимеразы (5 ед/мкл, ООО «Альфа-фермент», Россия) и 14,3 мкл бидистиллированной воды. Для ПЦР применяли следующий режим: предварительная денатурация при 94 °С в течение 2 мин, далее 40 циклов амплификации (94 °С — 15 с, 60 °С — 15 с, 72 °С — 15 с). Детекцию флуоресценции осуществляли при температуре 60 °С по каналу FAM.
При конструировании рекомбинантного контроля амплификации продукты ПЦР из реакционной смеси выделяли с помощью набора Cleanup Standard (ЗАО «Евроген», Россия). Выделенные продукты ПЦР клонировали в составе плазмидного вектора pTZ57 R/T («Thermo Fisher Scientific», США) в клетках штамма Escherichia coli Top10 («Invitrogen», США). Плазмидную ДНК выделяли с помощью набора Plasmid Miniprep (ЗАО «Евроген», Россия), концентрацию полученной ДНК определяли на микроспектрофотометре NanoDrop Lite («Thermo Fisher Scientific», США).
Для экспериментального воспроизведения ЧМЖЖ использовали трех овец романовской породы массой 45-50 кг каждая. Овец № 1 и № 2 инфицировали внутривенным введением культурального материала, содержащего вирус ЧМЖЖ (штамм Эпизоотический) в дозе 3,5 lg ТЦД50, а овцу № 3 использовали в качестве неинфицированного контроля. Все животные подвергались клиническому осмотру с ежедневной термометрией и отбором проб крови. Эксперименты с животными проводились под наблюдением Комиссии по биоэтике ФГБНУ ФИЦВиМ.
Для приготовления препаратов «сухой» капли на вырезанные из фильтровальной бумаги Whatman DE-81 («Whatman», Великобритания) кружки диаметром около 4-5 см наносили 0,05 см3 контаминированной вирусом ЧМЖЖ крови, стабилизированной в EDTA, и подсушивали при комнатной температуре. Кровь контаминировали культуральным материалом, содержащим штамм Nigeria 75/1 вируса ЧМЖЖ с титром 4,83±0,22 lg ТЦД50/см3, в соотношении 1000:1. Для выделения вирусной РНК приготовленные таким способом препараты помещали в микроцентрифужные пробирки с 800 мкл лизирующего буфера, содержащего 4 М изотиоцианат гуанидина, и инкубировали 10 мин при 56 °С. После центрифугирования при 10000 g в течение 10 мин надосадочную жидкость использовали для выделения РНК методом нуклеосорбции согласно инструкции к набору РИБО-Сорб (ЗАО «Интерлабсервис», Россия).
Полученные результаты подвергали статистической обработке общепринятыми методами. Достоверность статистической разницы между средними величинами определяли по разностному методу Стьюдента— Фишера, согласно которому для большинства биологических экспериментов при р > 0,05 закономерные различия между сравниваемыми величинами отсутствуют, а при р < 0,05, р < 0,01 и р < 0,001 различия статистически значимы. Средние арифметические (M) при числе опытов n и среднее квадратичное отклонение (s) средней арифметической рассчитывали в программе Microsoft Excel 365.
Результаты. На основании анализа доступных в базе данных GenBank (https://www.ncbi.nlm.nih.gov/) нуклеотидных последовательностей геномов различных штаммов и изолятов вируса ЧМЖЖ в качестве мишени для отжига праймеров была выбрана нуклеотидная последовательность гена гемагглютинина (H). С помощью программ BioEdit 7.0 и Oligo 6.0 (http://www.mbio.ncsu.edu/BioEdit/bioedit.html; http://www.oligo.net) осуществляли выравнивание нуклеотидных последовательностей, поиск консервативных участков и расчет структуры олигонуклеотидных праймеров. В результате были подобраны олигонуклеотидные праймеры PPRHf (5'- TCAAGATCGGGTCCAACATG-3') и PPRHr (5'-CAATCGGACTGGGTA-GAAGTAAG-3'), фланкирующие фрагмент гена гемагглютинина размером 148 п.н. Для детекции продуктов амплификации использовали гибридизационный флуоресцентно меченный зонд PPRHz 5'-(FAM)TCGCTCCTGGA-AACATCATAAGTGGC(BHQ1)-3'.
Условия постановки ОТ-ПЦР в режиме реального времени оптимизировали с использованием препаратов РНК, выделенных из культуры клеток Vero, инфицированной вирусом ЧМЖЖ (штамм Nigeria 75/1). Поскольку определенные в программе Oligo 6.0 температуры плавления праймеров составили 58-62 °С, в температурно-временном профиле реакции использовали температуру отжига 60 °С.
Аналитическую чувствительность тест-системы определяли при исследовании последовательных 10-кратных разведений культурального вируса ЧМЖЖ (штамм Nigeria 75/1) с инфекционной активностью 4,83±0,22 lg ТЦД50/см3.
Согласно представленным на рисунке 1 и в таблице 1 данным, последнее разведение вирусосодержащего материала, при котором был получен положительный результат, составило 10-4. Таким образом, аналитическая чувствительность ОТ-ПЦР в реальном времени для выявления генома вируса ЧМЖЖ составила 0,83±0,22 lg ТЦД50/см3. Рассчитанная эффективность амплификации составила 87 % при достоверности аппроксимации (R2), равной » 0,99. Внутритестовая повторяемость результатов ПЦР (значений пороговых циклов), характеризуемая средними стандартных отклонениями, находилась в пределах от 0,12 до 0,41. Коэффициенты вариаций значений пороговых циклов, полученные при повторных исследованиях одинаковых образцов, не превышали 1,5 %, что свидетельствует о высокой повторяемости результатов определения аналитической чувствительности ОТ-ПЦР.
Аналитическую специфичность тест-системы оценивали при исследовании образцов, содержащих вирусы ЧМЖЖ, ЧКРС, блютанга, образцов крови от клинически здоровых овец, а также интактных культур клеток Vero и BHK-21 (коллекция культур клеток ФГБНУ ФИЦВиМ). При этом положительный результат получили только с образцами, содержащими вирус ЧМЖЖ, чем подтверждается специфичность тест-системы.
В результате конструирования положительного контроля амплификации к тест-системе на основе клонирования фрагмента гена гемагглютинина вируса ЧМЖЖ размером 148 п.н. в составе вектора pTZ57 R/T с синтезом в ОТ-ПЦР получили препарат плазмидной ДНК, концентрация которого, определенная спектрофотометрически, составила 80 нг/мкл, что соответствует 2,4×1010 молекул на 1 мкл. Для оптимизации количества плазмидной ДНК, используемой в качестве положительного контроля, методом ОТ-ПЦР исследовали восемь 10-кратных разведений этого препарата, начиная с 10-3. Диапазон концентрации плазмидной ДНК, при котором соблюдается линейность амплификации, составил от 2,4×107 до 24 молекул ДНК/мкл, эффективность амплификации — 100 %, достоверность аппроксимации (R2) — 0,99, стандартное отклонение (s) значений Ct —0,32 (рис. 2). Рекомендуемые рабочие концентрации плазмиды в тест-системе, соответствующие диапазону аналитической чувствительности, составляют 2,4×107-2,4×104 молекул ДНК/мкл.
При экспериментальном воспроизведении ЧМЖЖ с целью проверки эффективность разработанной тест-системы в течение 15 сут у инфицированных животных не отмечали специфических клинических признаков заболевания, однако на 6-8-е сут у овец повышалась температура тела (до 40,2-40,5 °С). При этом с использованием предложенной тест-системы методом ОТ-ПЦР в реальном времени геном вируса ЧМЖЖ выявляли в пробах крови, отобранных с 5-х по 12-е сут после заражения. (табл. 2).
Наблюдаемая длительность виремии согласуется с описанной в литературе (25, 26), отсутствие четких специфических признаков может быть объяснено низкой использованной дозой заражения — 3,5 lg ТЦД50. Так, в работе M. El Harrak с соавт. (27), опубликованной в 2012 году, для воспроизведения инфекции использовался вирус ЧМЖЖ в дозе 5,1 lg ТЦД50.
При отборе и транспортировке проб биологических жидкостей в настоящее время широко используют метод «сухой» капли крови. Мы изучили применимость этого подхода в случае предложенной нами тест-системы. При отработке методики «сухой» капли крови для отбора и хранения образцов на бумажном носителе при диагностике ЧМЖЖ мы провели сравнительные исследования образцов РНК, выделенных из исходного вирусосодержащего материала и свежеприготовленных образцов «сухой» капли крови с помощью разработанной тест-системы. Результаты показали отсутствие значительных различий в пороговых циклах (Ct). При исследовании в ОТ-ПЦР образцов РНК, выделенных из препаратов «сухой» капли крови, хранившихся на бумажном носителе в течение 1 мес в плотно закрытых полиэтиленовых ZIP-пакетах при температуре 20±2 °С, и исходных препаратов, исследованных в начале эксперимента, различия в значениях Ct составили 3-5 циклов (табл. 3).
Таким образом, нами разработана высокоспецифичная и чувствительная тест-система для выявления генома вируса чумы мелких жвачных животных (ЧМЖЖ), основанная на амплификации фрагмента гена гемагглютинина методом ОТ-ПЦР в реальном времени. Разработанная тест-система обладает чувствительностью 0,83±0,22 lg ТЦД50/см3 и позволяет дифференцировать геном вируса ЧМЖЖ от вирусов, вызывающих схожие клинические признаки у овец, — вирусов чумы крупного рогатого скота и блютанга. Исследованием проб крови от экспериментально инфицированных овец подтверждена возможность практического применения разработанной тест-системы для диагностики ЧМЖЖ. Также отработан способ отбора и хранения проб крови на бумажном носителе (метод «сухой» капли крови) для выявления генома вируса ЧМЖЖ. Показано, что препараты «сухой» капли крови на бумажном носителе стабильны при комнатной температуре в течение 1 мес и могут использоваться для исследований методом ОТ-ПЦР в реальном времени.
1. Показатели аналитической чувствительности ОТ-ПЦР в реальном времени при выявлении генома вируса чумы мелких жвачных животных с использованием предложенной тест-системы (n = 3) |
||||
Разведение |
Титр вируса, |
Результат ОТ-ПЦР в реальном времени |
||
среднее значение Сt |
среднее значение s |
Сv, % |
||
Исходный материал |
4,83±0,22 |
16,53 |
0,12 |
0,75 |
10-1 |
3,83±0,22 |
19,13 |
0,29 |
1,50 |
10-2 |
2,83±0,22 |
22,83 |
0,17 |
0,74 |
10-3 |
1,83±0,22 |
26,70 |
0,14 |
0,53 |
10-4 |
0,83±0,22 |
31,07 |
0,41 |
1,35 |
10-5 |
-0,17±0,22 |
Нет значений |
|
|
2. Результаты термометрии и выявления генома вируса чумы мелких жвачных животных в пробах крови от экспериментально инфицированных овец при использовании предложенной тест-системы ОТ-ПЦР в реальном времени |
||||||
Время после заражения, сут |
Овца № 1 |
Овца № 2 |
Овца № 3 |
|||
температура |
значение Ct |
температура |
значение Ct |
температура |
значение Ct |
|
1 |
38,6 |
- |
39,5 |
- |
39,0 |
- |
2 |
39,4 |
- |
39,4 |
- |
39,3 |
- |
3 |
39,5 |
- |
39,2 |
- |
39,1 |
- |
4 |
39,6 |
- |
39,5 |
- |
39,0 |
- |
5 |
39,6 |
- |
39,6 |
- |
39,3 |
- |
6 |
40,2 |
+ (20,15) |
39,7 |
- |
38,9 |
- |
7 |
40,5 |
+ (20,30) |
40,2 |
+ (20,38) |
39,2 |
- |
8 |
40,4 |
+ (21,45) |
40,5 |
+ (20,57) |
39,3 |
- |
9 |
39,2 |
+ (21,89) |
39,9 |
+ (21,74) |
38,8 |
- |
10 |
39,0 |
+ (22,12) |
39,5 |
+ (21,90) |
39,0 |
- |
11 |
38,7 |
+ (22,90) |
39,9 |
+ (22,25) |
38,9 |
- |
12 |
38,8 |
+ (23,12) |
39,9 |
+ (22,85) |
39,1 |
- |
13 |
38,7 |
- |
39,8 |
- |
38,8 |
- |
14 |
38,8 |
- |
39,9 |
- |
38,9 |
- |
15 |
39,2 |
- |
39,7 |
- |
39,1 |
- |
Примечание. «+» — положительный результат, «-» — отрицательный результат. |
||||||
3. Выявление генома вируса чумы мелких жвачных животных в препаратах «сухой» капли крови на бумажном носителе методом ОТ-ПЦР в реальном времени |
|||
№ препарата |
Нативный препарат (кровь, |
Препарат «сухой» капли крови, |
|
свежеприготовленный |
хранившийся 1 мес |
||
1-й |
20,76 |
20,51 |
23,67 |
2-й |
20,15 |
20,83 |
24,98 |
3-й |
21,00 |
21,43 |
25,01 |
4-й |
20,35 |
21,02 |
23,73 |
Среднее |
20,64±0,64 |
20,99±0,38 |
24,55±0,62 |
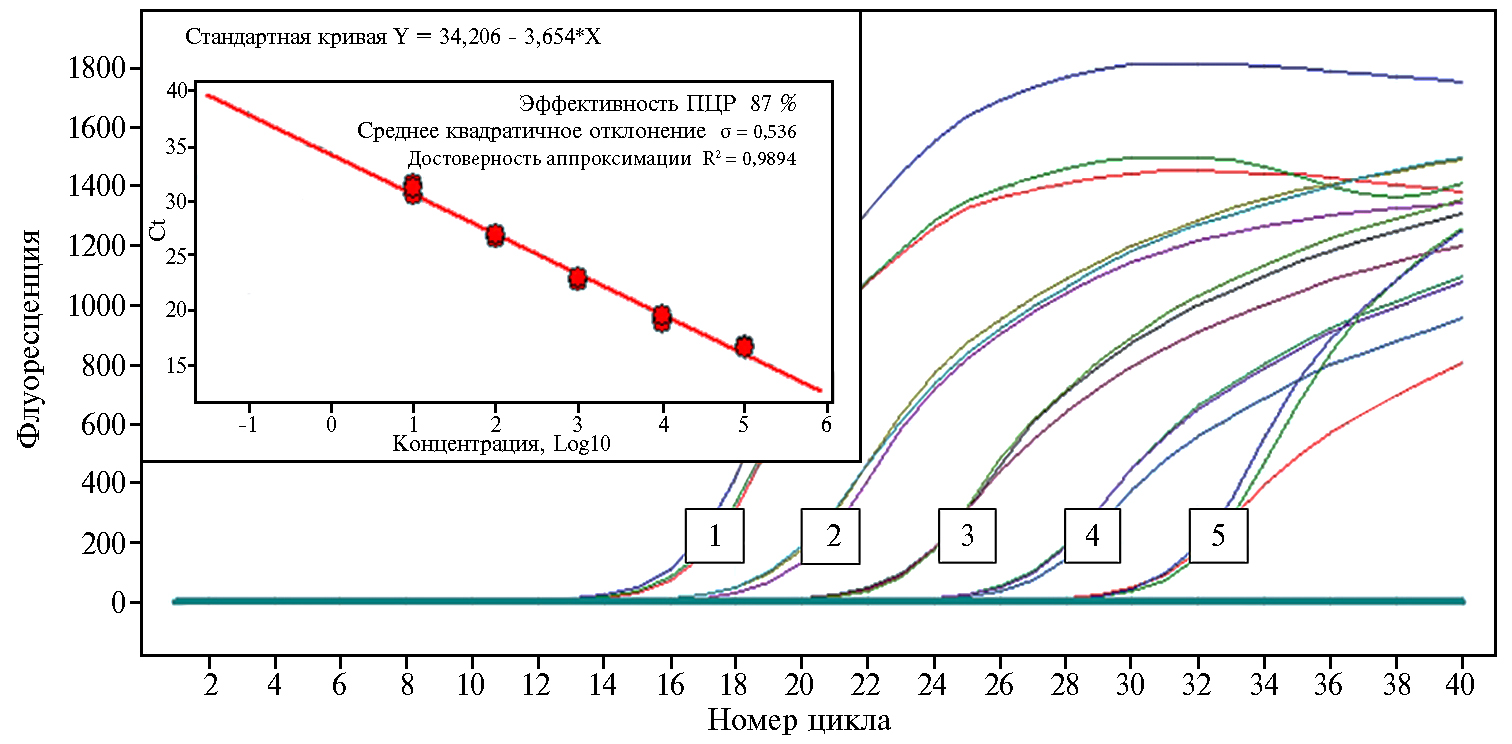
Рис. 1. Анализ последовательных 10-кратных разведений культурального материала, содержащего вирус чумы мелких жвачных животных (штамм Nigeria 75/1) с исходной активностью 4,83±0,22 lg ТЦД50/см3, с использованием предложенной тест-системы: 1 — исходный материал; 2-5 — разведения от 10-1 до 10-4.
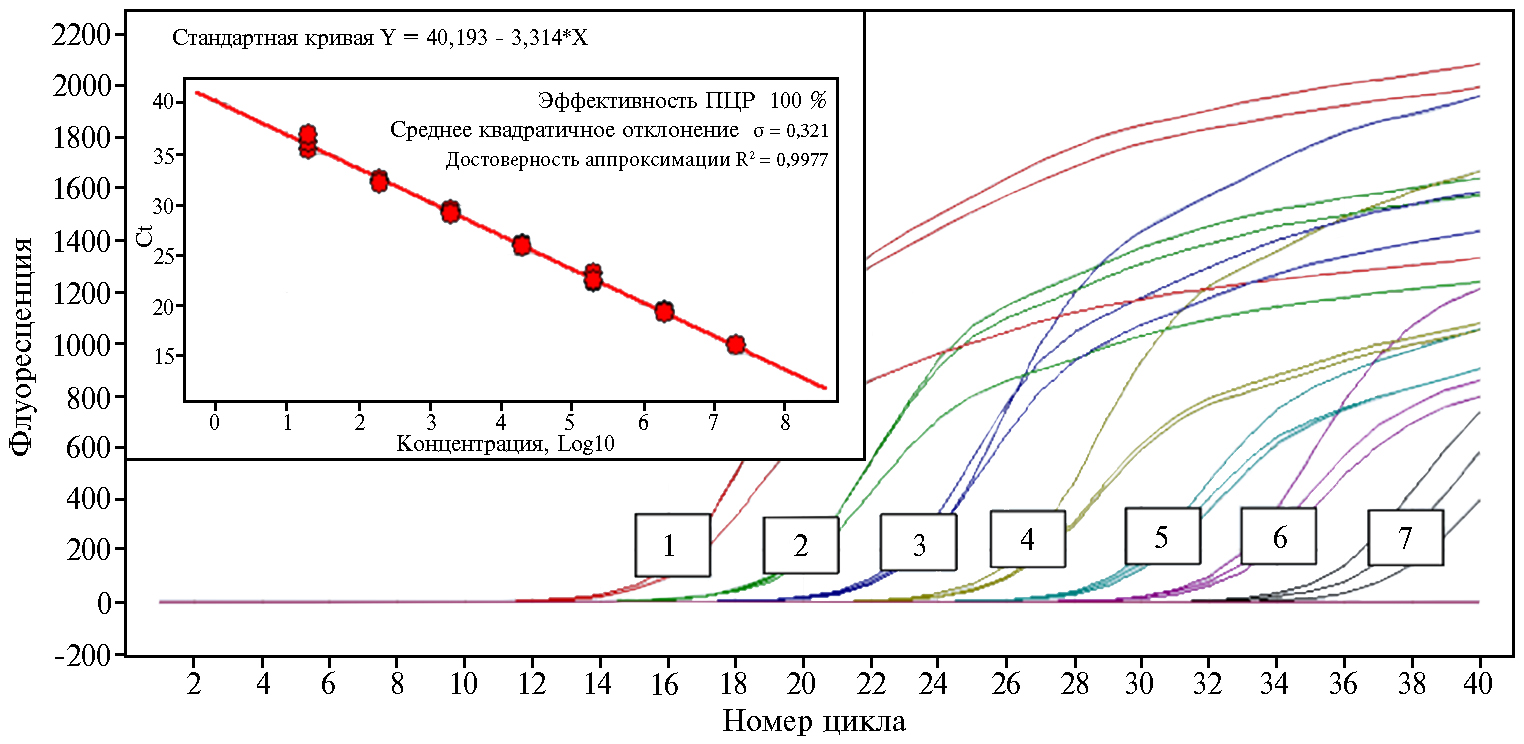
Рис. 2. Определение рабочих концентраций рекомбинантного положительного контроля амплификации (число копий/мкл)к тест-системе для выявления генома вируса чумы мелких жвачных животных методом ОТ-ПЦР в реальном времени: 1 — 2,4×107, 2 — 2×106, 3 — 2,4×105, 4 — 2,4×104, 5 — 2,4×103, 6 —2,4×102, 7 — 24.
ЛИТЕРАТУРА
- Закутский Н.И. Чума мелких жвачных животных (современное состояние, эпизоотология, специфическая профилактика и меры борьбы). Научный журнал КубГАУ, 2012, 83: 10-24.
- Сюрин В.Н., Самуйленко А.Я., Соловьёв Б.В., Фомина Н.В. Вирусные болезни животных. М., 1998.
- Kumar N., Maherchandani S., Kashyap S.K., Singh S.V., Sharma S., Chaubey K.K., Ly H. Peste des petits ruminants virus infection of small ruminants: a comprehensive review. Viruses, 2014, 6: 2287-2327 (doi: 10.3390/v6062287).
- Khan A., Saleemi M.K., Ali F., Abubakar M., Hussain R., Abbas R.Z., Khan I.A. Pathophysiology of peste des petits ruminants in sheep (Dorper & Kajli) and goats (Boer & Beetal). Microbial Pathogenesis,2018, 117: 139-147 (doi: 10.1016/j.micpath.2018.02.009).
- Wohlsein P., Saliki J. Rinderpest and peste des petits ruminants virus: plague of large and small ruminants — the diseases: clinical sciences and pathology. In: Rinderpest and peste des petits ruminants virus: plague of large and small ruminants /T. Barrett, P.P. Pastoret, W.P. Taylor (eds.).Academic Press, London, UK, 2006: 68-83.
- Baron M.D., Diallo A., Lancelot R., Libeau G. Peste des petits ruminants virus. Adv.Virus Res., 2016, 95: 1-42 (doi: 10.1016/bs.aivir.2016.02.001).
- Dundon W.G., Adombi C.M., Kanu S., Loitsch A., Cattoli G., Diallo A. Complete genome sequence of a lineage II peste des petits ruminants virus from Sierra Leone. Genome Announc., 2018, 6(1): e01417-17 (doi: 10.1128/genomeA.01417-17).
- Gibbs E.P., Taylor W.P., Lawman, M.J., Bryant, J. Classification of peste des petits ruminants virus as the fourth member of the genus morbillivirus. Intervirology, 1979, 11: 268-274.
- Samal S.K. Paramyxoviruses of animals. In: Encyclopedia of virology /B.W.J. Mahy, M.H.V. Van Regenmortel (eds.). San Diego, United States, Elsevier Science Publishing Co Inc., 2008: 40-47 (doi: 10.1016/B978-012374410-4.00460-X).
- Kwiatek O., Ali Y.H., Saeed I.K., Khalafalla A.I., Mohamed O.I., Obeida A.A., Abdelrahman M.B., Osman H.M., Taha K.M., Abbas Z., El Harrak M., Lhor Y., Diallo A., Lancelot R., Albina E., Libeau G. Asian lineage of peste des petits ruminants virus, Africa. Emerging Infectious Diseases, 2011, 17: 1223-1231 (doi: 10.3201/eid1707.101216).
- Dhar P, Sreenivasa B.P., Barrett T., Corteyn M., Singh R.P., Bandyopadhyay S.K. Recent epidemiology of peste des petits ruminants virus (PPRV). Veterinary Microbiology, 2002, 88: 153-159 (doi: 10.1016/S0378-1135(02)00102-5).
- Muniraju M., Munir M., Parthiban A.B.R., Banyard A.C., Bao J., Wang Z., Ayebazibwe C., Ayelet G., Harrak M.E., Mahapatra M., Libeau G., Batten C., Parida S. Molecular evolution of peste des petits ruminants virus. Emerging Infectious Diseases, 2014, 20: 2023-2033 (doi: 10.3201/eid2012.140684).
- Парилов С.В., Книзе А.В., Балышев В.М. Анализ и прогноз мировой эпизоотической ситуации по оспе овец и коз и чумы мелких жвачных животных в 2011-2015 гг.). Научный журнал КубГАУ, 2011, 69: 423-432.
- Щербинин C.В., Караулов А.К., Захаров В.М. Анализ угрозы заноса чумы мелких жвачных на территорию Российской Федерации. Ветеринария сегодня, 2017, 4: 17-22.
- Office International des Epizooties (OIE). Manual of diagnostic tests and vaccines for terrestrial animals 2017. Chapter 2.07.10. Peste des petits ruminants (infection with peste des petits ruminants virus). Available http://www.oie.int/fileadmin/Home/eng/. No date.
- Couacy-Hymann E., Bodjo S.C., Koffi M.Y., Kouakou C., Danho T. The early detection of peste-des-petits-ruminants (PPR) virus antigens and nucleic acid from experimentally infected goats using RT-PCR and immunocapture ELISA techniques. Research in Veterinary Science, 2009, 87: 332-335 (doi: 10.1016/j.rvsc.2009.03.002).
- Shaila M.S., Shamaki D., Forsyth M.A., Diallo A., Goatley L., Kitching R.P., Barrett T. Geographic distribution and epidemiology of Peste des Petits Ruminants viruses. Virus Research, 1996, 43: 149-153.
- Bao J., Li L., Wang Z., Barrett T., Suo L., Zhao W., Liu Y., Liu C., Li J. Development of one-step real-time RT-PCR assay for detection and quantitation of Peste des Petits Ruminants virus. Journal of Virological Methods, 2008, 148: 232-236 (doi: 10.1016/j.jviromet.2007.12.003).
- Kwiatek O., Keita D., Gil P., Fernandez-Pinero J., Clavero M.A., Albina E., Libeau G. Quantitative one-step real-time RT-PCR for the fast detection of the four genotypes of PPRV. Journal of Virological Methods, 2010, 165: 168-177 (doi: 10.1016/j.jviromet.2010.01.014).
- Batten C.A., Banyard A.C., King D.P., Henstock M.R., Edwards L., Sanders A., Buczkowski H., Oura C.C.L., Barrett T. A real time RT-PCR assay for the specific detection of Peste des petits ruminants virus. Journal of Virological Methods, 2011, 171: 401-404 (doi: 10.1016/j.jviromet.2010.11.022).
- Balamurugan V., Sen A., Venkatesan G., Yadav V., Bhanot V., Bhanuprakash V., Singh R.K. Application of semi-quantitative M gene-based hydrolysis probe (TaqMan) real-time RT-PCR assay for the detection of Peste des petitis ruminants virus in the clinical samples for investigation into clinical prevalence of disease. Transboundary and Emerging Diseases, 2010, 57: 383-395 (doi: 10.1111/j.1865-1682.2010.01160.x).
- Berg Sh., Bøtner A., Browman H., De Koeijer A., Depner K., Domingo M., Ducrot Ch., Edwards S., Fourichon Ch., Koenen F., More S., Raj M., Sihvonen L., Spoolder H., Stege-man J.A., Thulke H.H., Vågsholm I., Velarde A., Willeberg P., Zientara S. Scientific opinion on peste des petits ruminants. EFSA AHAW Panel (EFSA Panel on Animal Health and Welfare). EFSA Journal, 2015, 13(1): 3985 (doi: 10.2903/j.efsa.2015.39).
- Bhuiyan A.R., Chowdhury E.H., Kwiatek O., Parvin R., Rahman M.M., Islam M.R., Albina E., Libeau G. Dried fluid spots for peste des petits ruminants virus load evaluation allowing for non-invasive diagnosis and genotyping. BMC Vet. Res., 2014, 10: 247 (doi: 10.1186/s12917-014-0247-y).
- Bolais P.F., Vignoles P., Pereira P.F., Keim R., Aroussi A., Ismail K., Dardé M.L., Amendoeira M.R., Mercier A. Toxoplasma gondii survey in cats from two environments of the city of Rio de Janeiro, Brazil by Modified Agglutination Test on sera and filter-paper. Parasite Vectors, 2017, 10: 88 (doi: 10.1186/s13071-017-2017-8).
- Truong T., Boshra H., Embury-Hyatt C., Nfon C., Gerdts V., Tikoo S., Babiuk L.A., Kara P., Chetty T., Mather A. Peste des petits ruminants virus tissue tropism and pathogenesis in sheep and goats following experimental infection. PLoS ONE, 2014, 9: e87145 (doi: 10.1371/journal.pone.0087145).
- Couacy-Hymann E., Bodjo C., Danho T., Libeau G., Diallo A. Evaluation of the virulence of some strains of peste-des-petits-ruminants virus (PPRV) in experimentally infected West African dwarf goats. The Veterinary Journal, 2007, 173: 178-183 (doi: 10.1016/j.tvjl.2005.08.020).
- Harrak M.E., Touil N., Loutfi C., Hammouchi M., Parida S., Sebbar G., Chaffai N., Harif B., Messoudi N., Batten C., Oura C.A.L. A reliable and reproducible experimental challenge model for peste des petits ruminants virus. Journal of Clinical Microbiology, 2012, 50: 3738-3740 (doi: 10.1128/JCM.01785-12).