doi: 10.15389/agrobiology.2021.1.32rus
УДК 635.21:575.1:577.21
Для выращивания растений картофеля использовали экспериментальную установку искусственного климата ЭУИК (Институт биоинженерии ФИЦ Биотехнологии РАН). Работа выполнена при поддержке РФФИ (грант 18-29-07007) и ФНТП развития сельского хозяйства РФ на 2017-2025 годы (подпрограмма «Развитие селекции и семеноводства картофеля в Российской Федерации»).
ВАРИАБЕЛЬНОСТЬ ГЕНОМНЫХ RGA-ЛОКУСОВ СОВРЕМЕННЫХ ОТЕЧЕСТВЕННЫХ СОРТОВ КАРТОФЕЛЯ: ДАННЫЕ NBS-МАРКИРОВАНИЯ
Е.А. ДЬЯЧЕНКО ✉, А.В. КУЛАКОВА, Е.З. КОЧИЕВА, А.В. ЩЕННИКОВА
Свойственный растениям иммунитет направлен на защиту от биотических и абиотических стрессов и, как следствие, на адаптацию к изменяющимся условиям среды. На первом уровне защиты используется широкий спектр рецепторов фитопатогенов, кодируемых R-генами устойчивости. Присутствие в рецепторах консервативного NBS-домена дает возможность маркировать геном растения с помощью амплификации аналогов R-генов. На этом основано мультилокусное NBS-маркирование, которое позволяет эффективно охарактеризовать геном растения с точки зрения представленности и вариабельности семейства R-генов, продукты которых имеют NBS-домен. Этот метод применяется для изучения разнообразия локусов R-генов устойчивости сельскохозяйственных культур и родственных им дикорастущих видов, явлений интрогрессивной гибридизации и эволюции R-генов у видов растений с различной степенью резистентности к патогенам. Также с помощью NBS-маркирования проводится генотипирование коллекций генбанков, разработка кодоминантных маркеров и насыщение генетических карт. Существующая необходимость паспортизации и сравнения генотипов сортов и ограниченное число таких работ в России делают актуальными исследования по молекулярному маркированию сортов агрокультур отечественной и зарубежной селекции, возделываемых на территории Российской Федерации. В настоящей работе NBS-маркирование было использовано для генотипирования 60 сортов и пяти перспективных селекционных клонов картофеля Solanum tuberosum отечественной и зарубежной селекции, а также образца родственного вида Solanum stoloniferum (в качестве внешней группы). С помощью двух комбинаций праймер/фермент (NBS7/MseI и NBS9/MseI) было сгенерировано 204 NBS-фрагмента, включая 144 (70,6 %) полиморфных и один уникальный для сорта Гала. Для каждого сорта был определен индивидуальный, специфичный спектр NBS-фрагментов. Анализ матрицы генетических расстояний между сортами показал, что исследуемая выборка сортов высокополиморфна (GD = 0,18-0,45 при среднем значении 0,33). Генетические расстояния внутри анализируемой выборки варьировали больше, чем между сортами S. tuberosum и образцом S. stoloniferum (0,27-0,40). Наиболее близкими оказались сорта селекции Всероссийского НИИ картофельного хозяйства им. А.Г. Лорха Солнечный/Памяти Рогачева (GD = 0,18) и Великан/Вымпел (GD = 0,19), а наиболее удаленными — сорта Чароит/Ред Скарлетт (GD = 0,45). Был проведен статистический анализ результатов NBS-маркирования, который позволил кластеризовать изучаемые образцы картофеля по различным признакам, связанным с родословной и устойчивостью к фитопатогенам. На дендрограмме и графиках, построенных с помощью программ PAST и Structure 2.3.4, наблюдалась слабая тенденция к группировке сортов по признакам устойчивости к Y вирусу картофеля (Potato virus Y, Potyvirus, Potyviridae) и вирусу скручивания листьев картофеля (Potato leafroll virus, Polerovirus, Luteoviridae). Продемонстрировано, что использованные в настоящей работе системы праймер/фермент для NBS-анализа могут быть применены в дальнейшем для исследования механизмов устойчивости сортов картофеля к биотическим стрессам.
Ключевые слова: Solanum tuberosum, российские сорта картофеля, сорта зарубежной селекции, геномный полиморфизм, NBS-LRR-маркирование, RGA-анализ.
Иммунная система растений, направленная на защиту от биотических угроз и адаптацию, содержит широкий спектр R-генов устойчивости, кодирующих рецепторы фитопатогенов нескольких классов, самый многочисленный среди которых — NLR (nucleotide binding leucine-rich repeats proteins) (1, 2). Присутствие консервативных доменов в продуктах R-генов дает возможность анализировать геном растения с помощью амплификации аналогов R-генов (RGA — Resistance Gene Analog), число которых в геноме может достигать сотен за счет тандемных и эктопических дупликаций с последующей локальной перестройкой и конверсией генов (2). NBS-LRR-рецепторы имеют в своем составе два основных консервативных домена: центральный — NBS (Nucleotide Binding Site) и С-концевой — LRRs (Leucine-Rich Repeats) (1, 3). Семейства генов, кодирующих NBS-LRR-рецепторы, идентифицированы у различных видов растений в результате полногеномного секвенирования: 57 генов у Cucumis sativus, 149 — у Arabidopsis thaliana, 653 — у Oryza sativa. В геноме картофеля Solanum tuberosum L. обнаружено 435 генов и 179 псевдогенов семейства NBS-LRR (1).
В то время как последовательность LRR-домена относительно полиморфна, NBS-домен состоит из ряда высококонсервативных и строго упорядоченных участков P-loop, Kinase-2 и Gly-Leu-Pro-Leu (GLPL), мутации, затрагивающие эти участки, приводят к нарушению функции рецепторов (1). Мультилокусное NBS-маркирование (NBS-profiling) позволяет эффективно охарактеризовать геном растений с точки зрения представленности и вариабельности семейства R-генов устойчивости, продукт которых имеет NBS-домен. Метод основан на амплификации последовательностей геномной ДНК, фланкирующих участок, кодирующий NBS-домен R-генов, с помощью вырожденных праймеров, комплементарных высококонсервативным участкам NBS-домена (4).
В настоящее время метод NBS-маркирования широко применяется для исследования разнообразия RGA-локусов сельскохозяйственных культур (картофель, томат, пшеница, салат и др.) (5-7) и родственных им дикорастущих видов (2, 4, 8). Также NBS-маркирование позволяет исследовать явления интрогрессивной гибридизации между культурными и дикорастущими видами (6). Более того, NBS-метод применяется в исследованиях эволюции R-генов у видов растений с различной степенью устойчивости к патогенам, для генотипирования коллекций генбанков, разработки кодоминантных маркеров и насыщения генетических карт (4, 6, 7).
В настоящей работе проведено NBS-маркирование сортов и перспективных селекционных клонов картофеля преимущественно современной российской селекции.
Цель работы состояла в оценке полиморфизма NBS-LRR генов устойчивости и выявлении возможной корреляции между кластеризацией на основе данных NBS-анализа и признаками устойчивости к различным фитопатогенам картофеля.
Методика. Для NBS-LRR-маркирования отобрали 60 сортов и пять перспективных селекционных клонов картофеля Solanum tuberosum отечественной и зарубежной селекции, а также в качестве внешней группы — образец родственного вида Solanum stoloniferum, широко применяемого при селекции сортов (в том числе как источник устойчивости к различным фитопатогенам). Выбор сортов был обусловлен их использованием в исследованиях в рамках Федеральной научно-технической программы развития селекции и семеноводства картофеля в Российской Федерации. Большинство сортов выборки (59 из 65; 90,77 %) зарегистрировано в российском «Государственном реестре селекционных достижений, допущенных к использованию» (2020 год) (http://reestr.gossortrf.ru/reestr/culture/159.html). Посевной материал (клубни), предоставленный Всероссийским НИИ картофельного хозяйства им. А.Г. Лорха (ВНИИКХ), проращивали в стандартных условиях теплицы (23 °С /25 °С, 16 ч/8 ч день/ночь).
Геномную ДНК выделяли из ткани 5-суточных проростков с помощью СТАВ-метода (9, 10) в двух аналитических повторах.
NBS-маркирование проводили по стандартной методике (4). Для анализа 350 нг геномной ДНК образцов гидролизовали с использованием рестриктазы MseI («Thermo Fisher Scientific», США). Полученные фрагменты лигировали с соответствующими адаптерами и использовали для амплификации (амплификатор T100tm, ThermoCycler, «Bio-Rad», США), которую проводили в два раунда. Первый раунд ПЦР осуществлялся в течение 30 циклов (денатурация 30 с при 95 °С, отжиг праймеров 1 мин при 55-60 °С, синтез ДНК 2 мин при 72 °С) с применением NBS-праймеров NBS7, NBS9 (4, 11). Аликвоту ПЦР-смеси (0,5 мкл) использовали для второго раунда ПЦР (в тех же условиях) с NBS-праймером, меченным флуорофорами IRD 700/800, и MseI-адаптерным (4) праймером. Продукты амплификации разделяли в денатурирующем 6 % полиакриламидном геле и визуализировали с использованием гель-анализатора LI-COR 4300 (LI-COR operator manual; «LI-COR», США).
Для статистической обработки результатов NBS-маркирования полученные спектры NBS-фрагментов документировали в виде бинарных матриц (Microsoft Excel). Матрицы использовали для идентификации сортоспецифичных ДНК маркеров, определения коэффициентов попарного генетического сходства/различия между образцами (GS) и генетических расстояний (GD = 1 - GS), кластерного анализа методом ближайшего соседа (NJ) и методом главных координат (PCA) и выявления групп генетически сходных образцов (пакет программ PAST, http://www.nhm.uio.no/english/research/infrastructure/past/) (12). Геномную структуру исследуемых образцов анализировали с помощью программы Structure v. 2.3.4 (https://web.stan-ford.edu/group/pritchardlab/structure_software/release_versions/v2.3.4/html/structure.html), позволяющей находить общие генетические блоки и определять их соотношение в каждом образце (13, 14).
Результаты. Характеристика изученных сортов и образцов представлена в таблице 1 (полностью см. на http://www.agrobiology.ru).
NBS-маркирование. В настоящей работе был выполнен анализ адаптивно значимого семейства растительных R-генов у 60 сортов и пяти перспективных селекционных клонов картофеля S. tuberosum и образца S. stoloniferum.
Для амплификации были взяты вырожденные праймеры NBS7 и NBS9, комплементарные последовательности двух основных консервативных мотивов NBS-домена — P-loop и GLPL, что позволило маркировать как 5´-, так и 3´-области генов семейства NBS-LRR (рис. 1).
Использовав две комбинации NBS-праймер/фермент — NBS7/MseI и NBS9/MseI для каждого из анализируемых сортов, были получены индивидуальные спектры NBS-фрагментов размером 70-550 п.н., которые предположительно могут быть ассоциированы со специфичной устойчивость анализируемых сортов к патогенам.
Всего было получено 204 NBS-фрагмента, включая 144 полиморфных (70,6 %) и только один уникальный — для сорта Гала (табл. 2). Образцы картофеля с наиболее различающимися спектрами R-генов могут рассматриваться в качестве потенциально перспективных доноров генов устойчивости для селекции новых сортов.
Оставшиеся 60 (29,4 %) мономорфных NBS-фрагментов присутствовали у всех анализируемых образцов картофеля, что может отражать наличие неких общих генов устойчивости к некоторым фитопатогенам или их аллельных вариантов. Например, все исследуемые в представляемой работе сорта S. tuberosumи образец S. stoloniferum невосприимчивы к возбудителю рака картофеля Synchytrium endobioticum. Устойчивость к раку определяется двумя R-генами Sen1 и Sen2, локализованными на 11-й хромосоме (15). Оба гена — Sen1 и Sen2 моделируют резистентность к патотипу 1 S. endobioticum, который преимущественно распространен в России, тогда как Sen2 — еще и к другим семи патотипам из 40 распространенных в Европе (15-18). Проведенный нами анализ доступных в базе NCBI (https://www.ncbi.nlm.nih.gov/) последовательностей белков SEN1 (XP_006342849.1) и SEN2 (PGSC0003DMT400082374) показал, что они относятся к разным семействам, и белок SEN1 содержит P-loop (элемент домена NBS). Поэтому NBS-спектры, полученные в этом исследовании с NBS7-праймером, могут содержать фрагменты, имеющие отношение к гену Sen1 и одинаковые для всех анализируемых образцов картофеля.
Выявленный нами уровень полиморфизма R-генов у изученных сортов картофеля оказался крайне высоким, что свидетельствует об удачном подборе системы праймер/фермент для NBS-маркирования. Так, в сравнении с полученными нами 70,6 % полиморфных фрагментов, NBS-мар-кирование образцов твердой пшеницы (Triticum durum L.) с использованием рестриктаз MseI и AluI детектировало только соответственно 34 и 22 % полиморфных фрагментов (7). Помимо прочего, высокий уровень полиморфизма полученных NBS-спектров можно объяснить использованием в нашей работе LI-COR, который имеет высокую разрешающую способность при проведении фрагментного анализа. Так, при NBS-анализе образцов овса (Avena strigosa и A. wiestii) было продемонстрировано, что применение LI-COR значительно повышает процент детектируемых полиморфных фрагментов в NBS-спектрах (19).
Статистический анализ NBS-спектров. По данным маркирования семейства NBS-LRR генов устойчивости были составлены бинарные матрицы, которые использовались для кластерного анализа.
Значения генетических расстояний (GD) между анализируемыми сортами варьировали в пределах 0,18-0,45 (GDср. = 0,33). Наиболее близкими по результатам маркирования оказались сорта селекции ВНИИКХ: Солнечный и Памяти Рогачева (GD = 0,18), Великан и Вымпел (GD = 0,19). Максимальные значения генетических дистанций были выявлены между сортами Чароит и Ред Скарлетт (GD = 0,45). Стоит отметить, что GD между сортами картофеля и образцом S. stoloniferum варьировали незначительно (GD = 0,27-0,40). Это может быть связано с интенсивным использованием S. stoloniferum в селекции сортов картофеля в качестве донора ценных аллельных вариантов генов устойчивости (прежде всего к вирусным патогенам) (20). Большинство исследуемых сортов — современные, поэтому были созданы, скорее всего, на основе межвидовой гибридизации с участием нескольких дикорастущих видов, как это было показано (21).
Сведения о вариабельности семейства R-генов устойчивости у представителей рода Solanum ограничены (в том числе у сортов картофеля), в связи с этим представляет интерес сравнить полученные данные с таковыми у других видов растений. Так, NBS-маркирование 32 российских и зарубежных сортов яблони (Malus domestica Borkh.) показало, что уровень полиморфизма их R-генов (49 % полиморфных фрагментов, GDср. = 0,14) (22) значительно ниже значений, выявленных нами для сортов картофеля (GDср. = 0,33). NBS-генотипирование сортов пшеницы, выращиваемых в Турции, Казахстане и Европе, детектировало уровень полиморфизма R-генов (GDср. = 0,30) (23), сравнимый с показанным в настоящей работе. Анализ сортов перца (Capsicum annuum L.) показал, что межсортовой полиморфизм (GD до 0,26) в 2 раза ниже в сравнении с полиморфизмом у дикорастущих образцов того же вида (GD до 0,58) (8). Таким образом, можно предположить, что уровень полиморфизма генов устойчивости у сортов зависит как от числа и вариабельности R-генов в геноме вида, так и от степени вовлеченности дикорастущего материала в селекцию сортов.
В настоящее время NBS-маркирование все чаще применяют не только для генотипирования, но и для идентификации генов устойчивости к различным заболеваниям у сортов и видов растений (2, 24). Поэтому описанные нами праймерные системы для NBS-анализа могут быть применены в дальнейшем для исследования механизмов устойчивости сортов картофеля к биотическим стрессам.
На основе полученных NBS-спектров была построена дендрограмма, на которой анализируемые образцы формируют высокополиморфный кластер, разделенный на группы с низкой бутстрэп-поддержкой (рис. 2).
Представлялось интересным оценить возможные связи кластеризации образцов с определенными признаками, а также с оригинатором (см. табл. 1). Четкой кластеризации исследуемых сортов по принадлежности к селекционным центрам выявлено не было, что может объясняться интенсивным обменом селекционным материалом. Шесть зарубежных сортов сгруппировались попарно — Ред Скарлетт/Сатурна, Ньютон/Леди Клэр и Гала/Импала, однако это не было специфично для конкретного зарубежного селекционного центра (см. рис. 2). Тем не менее на дендрограмме можно выделить четыре кластера, объединяющие в основном сорта селекции ВНИИКХ (см. рис. 2).
Была также проанализирована возможная кластеризация образцов по резистентности к различным фитопатогенам. По устойчивости к Globodera rostochiensis на дендрограмме выделились несколько небольших кластеров (см. рис. 2), что может быть связано с использованием в селекции сортов разных доноров, включая в том числе различные дикорастущие виды, например Solanum acaule, S. spegazzinii и S. vernei (25). Из пяти известных патотипов G. rostochiensis (26) в Российской Федерации распространен один — Ro1 (27). Устойчивость к нему определяется доминантными аллелями генов Н1 (5-я хромосома) и Gro1 (7-я хромосома) (17, 28-32), локусы которых содержат большое число копий RGA (33, 34). Ген Н1 интрогрессирован в сорта от единичных нематодоустойчивых образцов S. tuberosum subsp. andigenum, S. vernei (26, 35), а ген Gro1-4 — от образцов S. spegazzinii (28).
Основными донорами устойчивости к возбудителю фитофтороза служат образцы Solanum demissum (25). По этому признаку анализируемые сорта рассеяны по дендрограмме без очевидной кластеризации (см. рис. 2). Возможное объяснение заключается в том, что белки BAK1/SERK3, моделирующие защитный ответ растений на возбудителя фитофтороза, относятся к рецептор-подобным киназам с лейцин-богатыми повторами (LRR-RLK), у которых нет NBS-домена (36, 37).
Y-вирус картофеля (PVY, Potato virus Y, Potyvirus, Potyviridae) относится к наиболее экономически важным патогенам (38). При ответе на заражение вирусный эффектор распознается в растении NBS-LRR-геном Ny-1 (9-я хромосома) (38, 39). На дендрограмме анализируемые сорта картофеля, устойчивые к PVY, формируют 4 группы (см. рис. 2).
Вторым по значимости после PVY считается вирус скручивания листьев картофеля (PLRV, Potato leafroll virus, Polerovirus, Luteoviridae). Три QTL, ассоциированных с устойчивостью к накоплению PLRV, локализованы на 11-й и 5-й хромосомах (40, 41). Интересно, что кластеризация анализируемых нами сортов картофеля по признаку устойчивости к PLRV пересекается с кластеризацией по устойчивости к PVY (см. рис. 2). Основными донорами устойчивости к обоим вирусам служат дикорастущие виды S. demissum, S. acaule, S. chacoense и S. stoloniferum (25). Возможно, поэтому на дендрограмме образец S. stoloniferum группировался с сортами, устойчивыми и к PVY, и к PLRV (см. рис. 2).
Довольно любопытной оказалась кластеризация двух сортов — Фиолетовый и Василек. Только они в изучаемой выборке формируют клубни с сине-фиолетовой окраской кожуры (а сорт Фиолетовый — еще и мякоти) за счет повышенного содержания антоцианов. Оба сорта устойчивы к фитофторе и Y-вирусу картофеля. Отметим, что при этом сорта имеют разные родословные. И хотя ранее была показана связь уровня защитного ответа растений на возбудителя фитофтороза с интенсивностью накопления антоцианов (42), объединение указанных сортов в одну группу отражает сходство их паттернов R-генов и никак не связано с окраской. Интересно отметить, что сведения по устойчивости анализируемых сортов, предоставленные в Госсортреестр оригинаторами сортов, не всегда совпадают с данными молекулярного маркирования (30-32, 43, 44), что, по всей вероятности, связано с трудностями визуального определения симптоматики ответа на заражение.
Выполненный кластерный анализ был дополнен анализом методом главных компонент (PCA), который подтвердил наличие единой полиморфной группы образцов сортов и линий российской и зарубежной селекции (рис. 3). Образцы группировались на PCA-графике несколько по-иному, чем на дендрограмме. Наблюдалась некоторая, хотя и крайне слабо выраженная тенденция к сближению образцов по признакам устойчивости к нематоде и вирусам PVY/PLRV и по принадлежности к селекционным центрам. Зарубежные сорта сместились в крайнюю правую часть графика, а образец S. stoloniferum расположился почти по центру.
Также по данным NBS-маркирования был проведен анализ геномной структуры 65 анализируемых образцов картофеля с использованием программы Structure 2.3.4. Были проверены варианты разделения на подгруппы от k = 2 до k = 15, и наилучший результат был получен для k = 10 (LnLike = -75339,2). На рисунке 4 данные NBS-маркирования анализируемых образцов представлены в виде различных соотношений блоков с наборами генов устойчивости.
Проведенный анализ позволил идентифицировать 10 сформированных образцами кластеров, в пяти из них преобладал один из блоков в геноме. Также была выделена одна группа, где отсутствовали мажорные блоки, причем образцы этой группы отличались от остальных наибольшим полиморфизмом. Проведенный анализ не выявил ни одного образца, который был бы представлен одним или двумя блоками (см. рис. 4).
Итак, в результате мультилокусного анализа с использованием метода NBS-маркирования впервые была охарактеризована коллекция из 60 сортов и пяти селекционных клонов (Solanum tuberosum) российской и зарубежной селекции. В целом выявлен высокий уровень полиморфизма NBS-LRR генов у российских сортов картофеля. Определены группы образцов с наиболее схожими и различающимися паттернами NBS-LRR генов. Обнаружена слабо выраженная тенденция к сближению образцов по признакам устойчивости к нематоде и вирусам PVY (Potato virus Y)/PLRV (Potato leafroll virus) и по принадлежности к селекционным центрам. Показано, что использованные в настоящей работе системы праймер/фермент могут быть применены в дальнейшем для NBS-генотипирования сортов картофеля при первичном скрининге коллекций по признакам устойчивости образцов к различным биотическим стрессам, а также для исследования механизмов такой устойчивости к биотическим стрессам.

2. Характеристика полиморфизма, выявленного в результате NBS-маркирования 65 сортов и селекционных клонов картофеля и образца Solanum stoloniferum |
||||
Комбинация праймер/фермент |
Число NBS-фрагментов |
|||
общее |
полиморфные |
уникальные |
||
всего |
% |
|||
NBS7/MseI |
108 |
70 |
64,8 |
1 |
NBS9/MseI |
96 |
74 |
77,1 |
0 |
Всего |
204 |
144 |
70,6 |
1 |
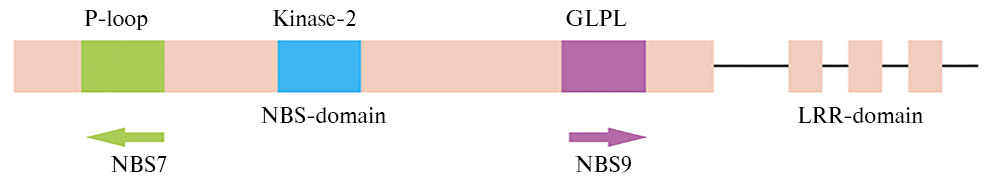
Рис. 1. Схема расположения праймеров NBS7 и NBS9 для проведения NBS-маркирования: NBS — Nucleotide Binding Site, LRR — Leucine-Rich Repeats, GLPL — Gly-Leu-Pro-Leu.
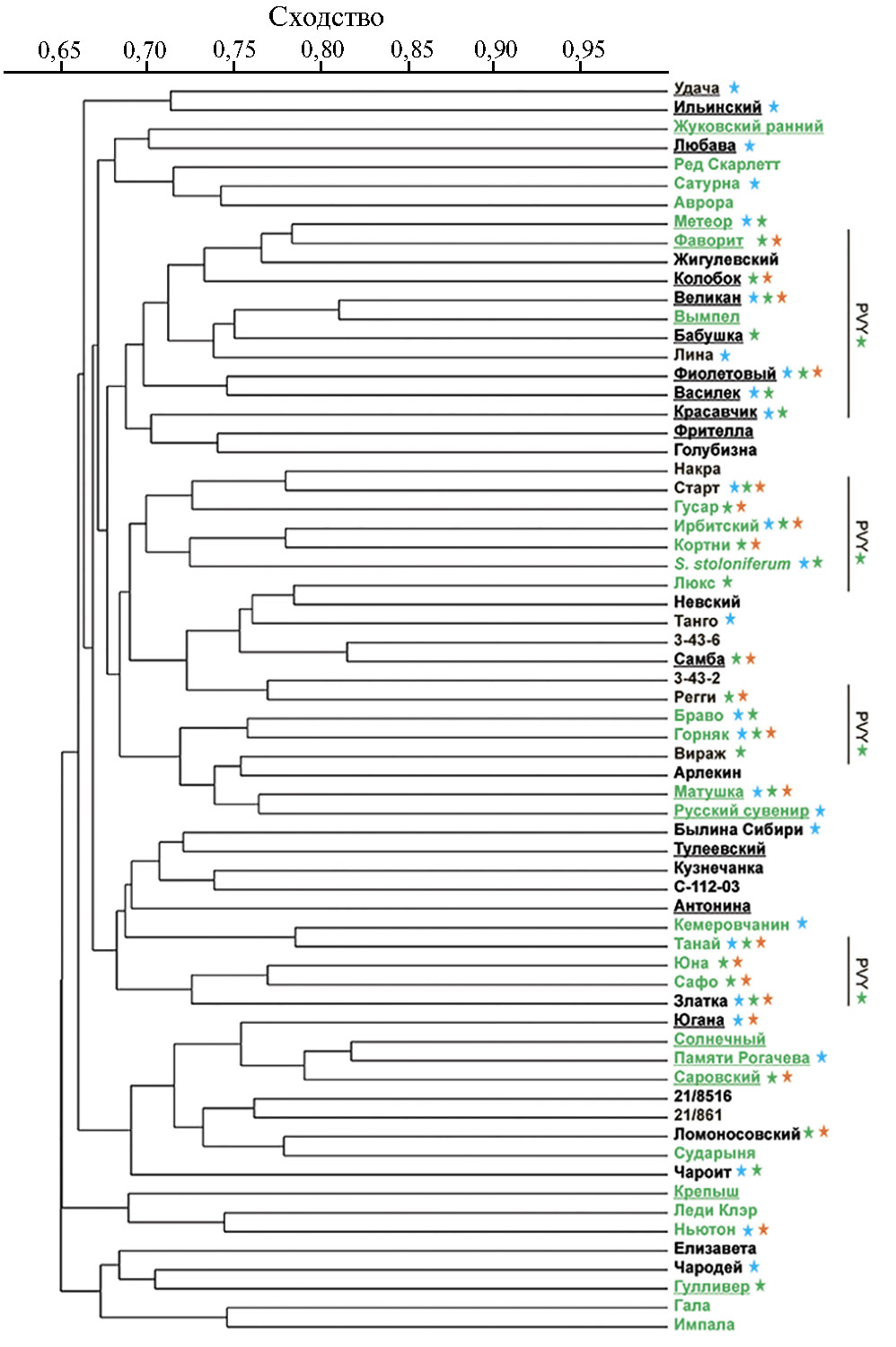
Рис. 2. Дендрограмма генетических различий 65 изученных сортов и селекционных клонов картофеля и вида Solanum stoloniferum, построенная методом ближайшего соседа (NJ, программа PAST) по данным NBS-маркирования. Выделены сорта, устойчивые к нематоде (зеленый шрифт), фитофторе (синяя звездочка рядом с названием), PVY (Potato virus Y, зеленая звездочка) и PLRV (Potato leafroll virus, оранжевая звездочка). Названия сортов селекции Всроссийского НИИ картофельного хозяйства им. А.Г. Лорха отмечены полужирным шрифтом и подчеркнуты.
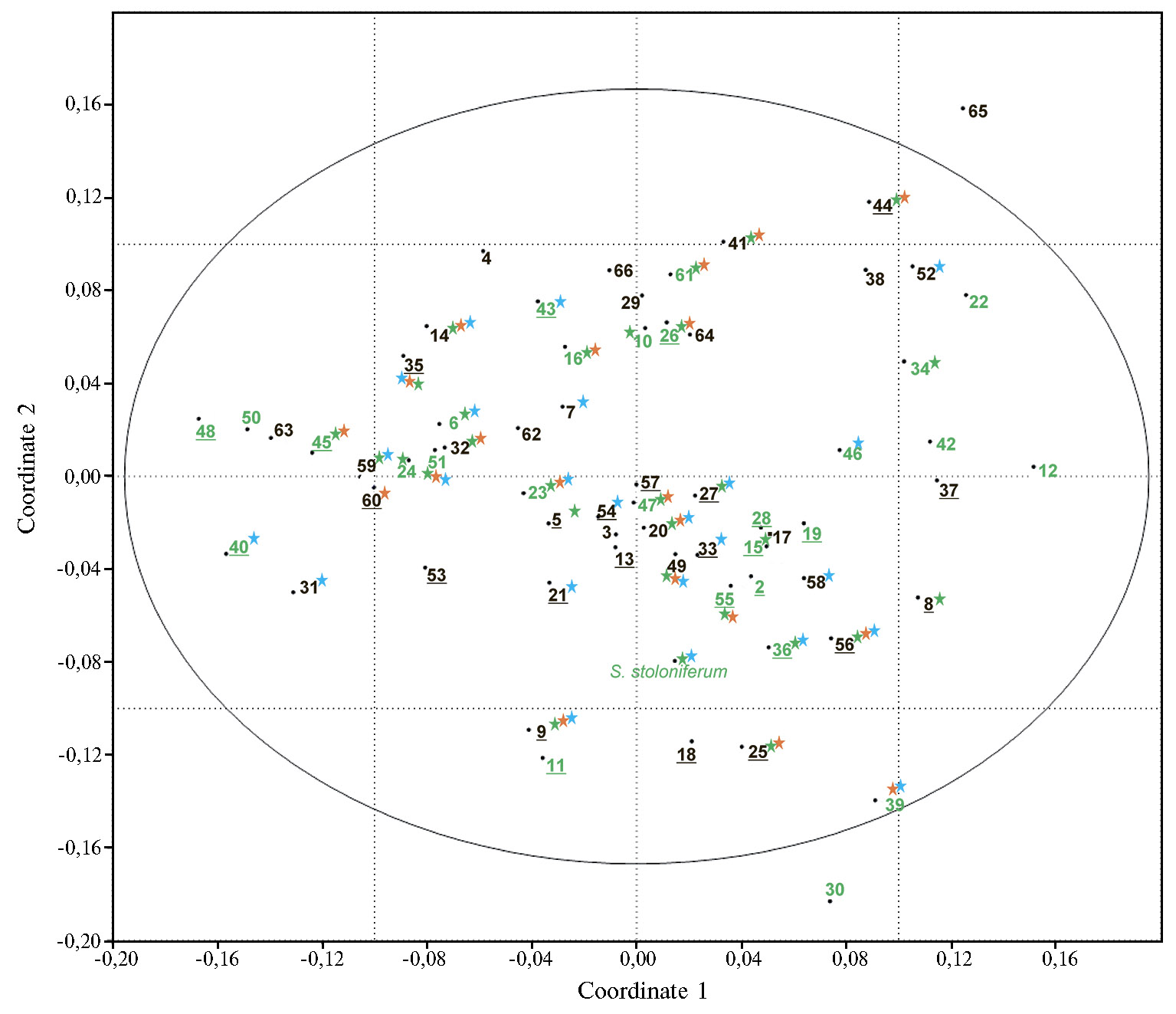
Рис. 3. График PCA-анализа данных NBS-маркирования 65 изученных сортов и селекционных клонов картофеля и вида Solanum stoloniferum. Цифры соответствуют нумерации образцов в таблице 1. Выделены сорта, устойчивые к нематоде (цифры зеленого цвета), фитофторе (синяя звездочка рядом с номером), PVY (Potato virus Y, зеленая звездочка рядом с номером) и PLRV (Potato leafroll virus, оранжевая звездочка рядом с номером). Номера сортов селекции Всерос-сийского НИИ картофельного хозяйства им. А.Г. Лорха подчеркнуты.
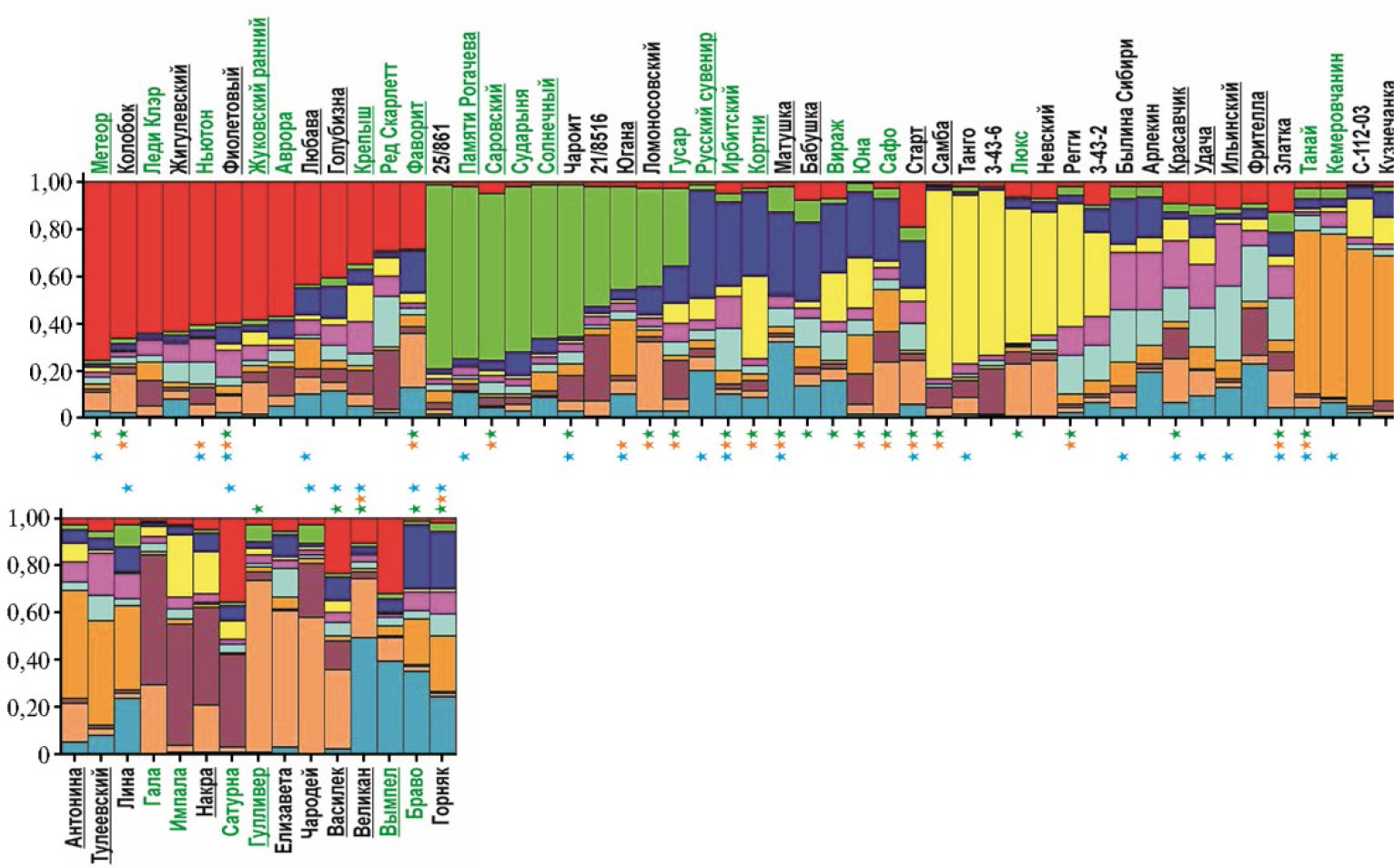
Рис. 4. Анализ геномной структуры 65 изученных сортов и селекционных клонов картофеля по данным NBS-маркиро-вания (k = 10, программа Structure 2.3.4). Анализ прово-дился без сравнения с данными по виду Solanum stoloniferum. Выделены названия сортов, устойчивых к нематоде (шрифт зеленого цвета), PVY (Potato virus Y, зеленая звездочка), PLRV (Potato leafroll virus, оранжевая звездочка) и фито-фторозу (синяя звездочка). Названия сортов селекции Все-российского НИИ картофельного хозяйства им. А.Г. Лор-ха подчеркнуты.
ЛИТЕРАТУРА
- Marone D., Russo M.A., Laidò G., De Leonardis A.M., Mastrangelo A.M. Plant nucleotide binding site-leucine-rich repeat (NBS-LRR) genes: active guardians in host defense responses. International Journal of Molecular Sciences, 2013, 14(4): 7302-7326 (doi: 10.3390/ijms14047302).
- Vossen J.H., Dezhsetan S., Esselink D., Arens M., Sanz M.J., Verweij W., Verzaux E., van der Linden C.G. Novel applications of motif-directed profiling to identify disease resistance genes in plants. Plant Methods, 2013, 9(1): 37 (doi: 10.1186/1746-4811-9-37).
- Glowacki S., Macioszek V.K., Kononowicz A.K. R proteins as fundamentals of plant innate immunity. Cellular & Molecular Biology Letters, 2010, 16(1): 1-24 (doi: 10.2478/s11658-010-0024-2).
- van der Linden C.G., Wouters D.C., Mihalka V., Kochieva E.Z., Smulders M.J., Vosman B. Efficient targeting of plant disease resistance loci using NBS profiling. Theoretical and Applied Genetics, 2004, 109(2): 384-393 (doi: 10.1007/s00122-004-1642-8).
- Calenge F., van der Linden C.G., van de Weg E., Schouten H.J., van Arkel G., Denancé C., Durel C.E. Resistance gene analogues identified through the NBS-profiling method map close to major genes and QTL for disease resistance in apple. Theoretical and Applied Genetics, 2005, 110(4): 660-668 (doi: 10.1007/s00122-004-1891-6).
- Syed N.H., Sørensen A.P., Antonise R., van de Wiel C., van der Linden C.G., van 't Westende W., Hooftman D.A., den Nijs H.C., Flavell A.J. A detailed linkage map of lettuce based on SSAP, AFLP and NBS markers. Theoretical and Applied Genetics, 2006, 112(3): 517-527 (doi: 10.1007/s00122-005-0155-4).
- Mantovani P., van der Linden G., Maccaferri M., Sanguineti M.C., Tuberosa R. Nucleotide-binding site (NBS) profiling of genetic diversity in durum wheat. Genome, 2006, 49(11): 1473-1480 (doi: 10.1139/g06-100).
- Кочиева Е.З., Рыжова Н.Н. Анализ вариабельности семейства генов устойчивости у представителей вида перца Capsicumannuum. Доклады академии наук, 2009, 425(2): 256-258.
- Puchooa D. A simple, rapid and efficient method for the extraction of genomic DNA from lychee (Litchi chinensis Sonn.). African Journal of Biotechnology,2004, 3: 253-255 (doi: 10.5897/AJB2004.000-2046).
- Gavrilenko T., Antonova O., Shuvalova A., Krylova E., Alpatyeva N., Spooner D., Novikova L. Genetic diversity and origin of cultivated potatoes based on plastid microsatellite polymorphism. Genetic Resources and Crop Evolution, 2013, 60: 1997-2015 (doi: 10.1007/s10722-013-9968-1).
- Brugmans B., Wouters D., van Os H., Hutten R., van der Linden G., Visser R.G., van Eck H.J., van der Vossen E.A. Genetic mapping and transcription analyses of resistance gene loci in potato using NBS profiling. Theoretical and Applied Genetics, 2008, 117: 1379-1388 (doi: 10.1007/s00122-008-0871-7).
- Hammer O., Harper D.A.T., Ryan P.D. PAST: Paleontological statistics software package for education and data analysis. Palaeontologia Electronica, 2001, 4(1): 1-9 (http://palaeo-electronica.org/2001_1/past/issue1_01.htm).
- Pritchard J.K., Stephens M., Donnelly P. Inference of population structure using multilocus genotype data. Genetics, 2000, 155(2): 945-959 (https://www.genetics.org/content/155/2/945).
- Hubisz M.J., Falush D., Stephens M., Pritchard J.K. Inferring weak population structure with the assistance of sample group information. Molecular Ecology Resources, 2009, 9(5): 1322-1332 (doi: 10.1111/j.1755-0998.2009.02591.x).
- Plich J., Przetakiewicz J., Śliwka J., Flis B., Wasilewicz-Flis I., Zimnoch-Guzowska E. Novel gene Sen2 conferring broad-spectrum resistance to Synchytrium endobioticum mapped to potato chromosome XI. Theoretical and Applied Genetics, 2018, 131(11): 2321-2331 (doi: 10.1007/s00122-018-3154-y).
- Hehl R., Faurie E., Hesselbach J., Salamini F., Witham S. TMV resistance gene N homologues are linked to Synchytrium endobioticum resistance in potato. Theoretical and Applied Genetics, 1999, 98: 379-386 (doi: 10.1007/s001220051083).
- Ballvora A., Hesselbach J., Niewohner J., Leister D., Salamini F., Gebhardt C. Marker enrichment and high-resolution map of the segment of potato chromosome VII harbouring the nematode resistance gene Gro1. Molecular and General Genetics, 1995, 249: 82-90 (doi: 10.1007/BF00290239).
- Khiutti A., Afanasenko O., Antonova O., Shuvalov O., Novikova L., Krylova E., Chalaya N., Mironenko N., Spooner D.M., Gavrilenko T. Characterization of resistance to Synchytrium endobioticum in cultivated potato accessions from the collection of Vavilov Institute of Plant Industry (VIR) collection. Plant Breeding, 2012, 131: 744-750 (doi: 10.1111/j.1439-0523.2012.02005.x).
- Sanz M.J., Loarce Y., Fominaya A., Vossen J.H., Ferrer E. Identification of RFLP and NBS/PK profiling markers for disease resistance loci in genetic maps of oats. Theoretical and Applied Genetics, 2013, 126(1): 203-218 (doi: 10.1007/s00122-012-1974-8).
- Рогозина Е.В., Хавкин Э.Е. Межвидовые гибриды картофеля как доноры долговременной устойчивости к патогенам. Вавиловский журнал генетики и селекции, 2017, 21(1): 30-41 (doi: 10.18699/VJ17.221).
- Антонова О.Ю., Швачко Н.А., Новикова Л.Ю., Шувалов О.Ю., Костина Л.И., Клименко Н.С., Шувалова А.Р., Гавриленко Т.А. Генетическое разнообразие сортов картофеля российской селекции и стран ближнего зарубежья по данным полиморфизма SSR-локусов и маркеров R-генов устойчивости. Вавиловский журнал генетики и селекции, 2016, 20(5): 596-606 (doi: 10.18699/VJ16.181).
- Савельева Е.Н., Борис К.В., Кочиева Е.З., Кудрявцев А.М. Полиморфизм NBS-LRR генов устойчивости сортов яблони (Malusdomestica Borkh.) по данным NBS-профайлинга. Генетика, 2016, 52(12): 1463-1468 (doi: 10.1134/S1022795416120115).
- Sayar-Turet M., Dreisigacker S., Braun H.J., Hede A., MacCormack R., Boyd L.A. Genetic variation within and between winter wheat genotypes from Turkey, Kazakhstan, and Europe as determined by nucleotide-binding-site profiling. Genome, 2011, 54(5): 419-430 (doi: 10.1139/g11-008).
- Ma F.F., Wu M., Liu Y.N., Feng X.Y., Wu X.Z., Chen J.Q., Wang B. Molecular characterization of NBS-LRR genes in the soybean Rsv3 locus reveals several divergent alleles that likely confer resistance to the soybean mosaic virus. Theoretical and Applied Genetics, 2018, 131(2): 253-265 (doi: 10.1007/s00122-017-2999-9).
- Machida-Hirano R. Diversity of potato genetic resources. Breeding Science, 2015, 65(1): 26-40 (doi: 10.1270/jsbbs.65.26).
- Kort J., Ross H., Rumpenhorst H.J., Stone A.R. An international scheme for the identification of pathotypes of potato cyst nematodes Globodera rostochiensis and G. pallida. Nematologica, 1977, 23: 333-339 (doi: 10.1163/187529277X00057).
- Limantseva L., Mironenko N., Shuvalov O., Antonova O., Khiutti A., Novikova L., Afanasenko O., Spooner D., Gavrilenko T. Characterization of resistance to Globodera rostochiensis pathotype Ro1 in cultivated and wild potato species accessions. Plant Breeding, 2014, 133(5): 660-665 (doi: 10.1111/pbr.12195).
- Barone A., Ritter E., Schachtschabel U., Debener T., Salamini F., Gebhardt C. Localization by restriction fragment length polymorphism mapping in potato of a major dominant gene conferring resistance to the potato cyst nematode Globodera rostochiensis. Molecular Genetics and Genomics, 1990, 224(2): 177-182 (doi: 10.1007/bf00271550).
- Bakker E., Achenbach U., Bakker J., van Vliet J., Peleman J., Segers B., van der Heijden S., van der Linde P., Graveland R., Hutten R., van Eck H., Coppoolse E., van der Vossen E., Bakker J., Goverse A. A high-resolution map of the H1 locus harboring resistance to the potato cyst nematode Globodera rostochiensis. Theoretical and Applied Genetics, 2004, 109(1): 146-152 (doi: 10.1007/s00122-004-1606-z).
- Клименко Н.С., Антонова О.Ю., Костина Л.И., Мамадбокирова Ф.Т., Гавриленко Т.А. Маркер-опосредованная селекция отечественных сортов картофеля с маркерами генов устойчивости к золотистой картофельной нематоде (патотип RO1). Труды по прикладной ботанике, генетике и селекции, 2017, 178(4): 66-75 (doi: 10.30901/2227-8834-2017-4-66-75).
- Гавриленко Т.А., Клименко Н.С., Антонова О.Ю., Лебедева В.А., Евдокимова З.З., Гаджиев Н.М., Апаликова О.В., Алпатьева Н.В., Костина Л.И., Зотеева Н.М., Мамадбокирова Ф.Т., Егорова К.В. Молекулярный скрининг сортов и гибридов картофеля северо-западной зоны Российской Федерации. Вавиловский журнал генетики и селекции, 2018, 22(1): 35-45 (doi: 10.18699/VJ18.329).
- Бирюкова В.А., Шмыгля И.В., Абросимова С.Б., Запекина Т.И., Мелешин А.А., Митюшкин А.В., Мананков В.В. Поиск источников генов устойчивости к патогенам среди образцов селекционно-генетических коллекций ВНИИКХ с использованием молекулярных маркеров. Защита картофеля, 2015, 1: 3-7.
- Paal J., Henselewski H., Muth J., Meksem K., Menendez C., Salamini F., Ballvora A., Gebhardt C. Molecular cloning of the potato Gro 1-4 gene conferring resistance to pathotype Ro1 of the root cyst nematode Globodera rostochiensis, based on a candidate gene approach. The Plant Journal,2004, 38(2): 285-297 (doi: 10.1111/j.1365-313X.2004.02047.x).
- Finkers-Tomczak A., Bakker E., Boer J., Vossen E., Achenbach U., Golas T., Suryaningrat S., Smant G., Bakker J., Goverse A. Comparative sequence analysis of the potato cyst nematode resistance locus H1 reveals a major lack of co-linearity between three haplotypes in potato (Solanum tuberosum ssp.). Theoretical and Applied Genetics, 2011, 122: 595-608 (doi: 10.1007/s00122-010-1472-9).
- Ellenby C. Tuber forming species and varieties of the genus Solanum tested for resistance to the potato root eelworm Heterodera rostochiensis Wollenweber. Euphytica, 1954, 3: 195-202 (doi: 10.1007/BF00055593).
- Chaparro-Garcia A., Wilkinson R.C., Gimenez-Ibanez S., Findlay K., Coffey M.D., Zipfel C., Rathjen J.P., Kamoun S., Schornack S. The receptor-like kinase SERK3/BAK1 is required for basal resistance against the late blight pathogen Phytophthora infestans in Nicotiana benthamiana. PLoS ONE, 2011, 6(1): e16608 (doi: 10.1371/journal.pone.0016608).
- Leesutthiphonchai W., Vu A.L., Ah-Fong A.M.V., Judelson H.S. How does Phytophthora infestans evade control efforts? Modern insight into the late blight disease. Phytopathology, 2018, 108(8): 916-924 (doi: 10.1094/PHYTO-04-18-0130-IA).
- Baebler Š., Coll A., Gruden K. Plant molecular responses to Potato Virus Y: a continuum of outcomes from sensitivity and tolerance to resistance. Viruses, 2020, 12(2): pii:E217 (doi 10.3390/v12020217).
- Szajko K., Strzelczyk-Żyta D., Marczewski W. Ny-1 and Ny-2 genes conferring hypersensitive response to potato virus Y (PVY) in cultivated potatoes: mapping and marker-assisted selection validation for PVY resistance in potato breeding. MolecularBreeding, 2014, 34: 267-271 (doi: 10.1007/s11032-014-0024-4).
- Marczewski W., Flis B., Syller J., Strzelczyk-Zyta D., Hennig J., Gebhardt C. Two allelic or tightly linked genetic factors at the PLRV.4 locus on potato chromosome XI control resistance to potato leafroll virus accumulation. Theoretical and Applied Genetics,2004, 109(8): 1604-1609 (doi: 10.1007/s00122-004-1780-z).
- Velásquez A.C., Mihovilovich E., Bonierbale M. Genetic characterization and mapping of major gene resistance to potato leafroll virus in Solanum tuberosum ssp. andigena. Theoretical and Applied Genetics, 2007, 114(6): 1051-1058. (doi: 10.1007/s00122-006-0498-5).
- de Vries S., von Dahlen J.K., Schnake A., Ginschel S., Schulz B., Rose L.E. Broad-spectrum inhibition of Phytophthora infestans by fungal endophytes. FEMS Microbiology Ecology,2018, 94(4): fiy037 (doi: 10.1093/femsec/fiy037).
- Клименко Н.С., Антонова О.Ю., Желтова В.В., Фомина Н.А., Костина Л.И., Мамадбокирова Ф.Т., Гавриленко Т.А. Скрининг сортов картофеля (Solanum tuberosum L.) российской селекции с помощью маркеров R-генов устойчивости к Y-вирусу картофеля. Сельскохозяйственная биология, 2019, 54(5): 958-969 (doi: 10.15389/agrobiology.2019.5.958rus).
- Бирюкова В.А., Шмыгля И.В., Жарова В.А., Бекетова М.П., Рогозина Е.В., Митюшкин А.В., Мелёшин А.А. Молекулярные маркеры генов экстремальной устойчивости к y вирусу картофеля в сортах и гибридах Solanum tuberosum L. Российская сельскохозяйственная наука, 2019, 5: 17-22 (doi: 10.31857/S2500-26272019517-22).