doi: 10.15389/agrobiology.2021.1.20rus
УДК 633.853.55:575.1:577.21
Работа выполнена при финансовой поддержке Российского научного фонда, Соглашение от 21 июля 2017 г. № 17-74-10233 по научному проекту: «Адаптация элементов технологии геномного редактирования CRISPR/Cas9 для улучшения генома клещевины обыкновенной (Ricinus communis L.)».
СЕКВЕНИРОВАНИЕ ПРОМОТОРОВ ГЕНОВ U6 КЛЕЩЕВИНЫ (Ricinus communis L.) И СОЗДАНИЕ НА ИХ ОСНОВЕ ВЕКТОРОВ ДЛЯ ГЕНОМНОГО РЕДАКТИРОВАНИЯ С ПОМОЩЬЮ СИСТЕМЫ CRISPR/Cas9
О.С. АЛЕКСАНДРОВ ✉, Г.И. КАРЛОВ
Клещевина — важная сельскохозяйственная культура, которую выращивают во многих странах мира и используют в основном для получения касторового масла, широко применяемого в различных отраслях промышленности. Жмыхи и шроты, которые остаются после отжима масла, богаты белком и перспективны для использования в качестве протеиновых добавок при производстве кормов, однако содержат токсины — белок рицин и алкалоид рицинин. Одним из способов детоксификации клещевины может стать геномное редактирование с помощью системы CRISPR/Cas9. Эта технология заключается в разрезании целевого участка интактной ДНК ферментом Cas9 при содействии короткого направляющего фрагмента РНК. Доставка в клетку редактируемого растения генов, кодирующих Cas9 и направляющую РНК, часто осуществляется с помощью плазмидных векторов. Для эффективного синтеза направляющей РНК в таких векторах обычно используют промоторы генов, кодирующих малые ядерные РНК. При редактировании двудольных растений чаще всего используют промотор U6 гена арабидопсиса, однако эффективность редактирования повышается, если использовать векторные конструкции с промотором редактируемого растения. В настоящей работе впервые проведена амплификация, секвенирование и анализ промоторов гена U6 клещевины. Найденные последовательности были проанализированы и использованы в конструировании CRISPR/Cas9 векторов для дальнейшего редактирования генов рицина. Нашей целью было изучение промоторов гена U6 клещевины с помощью биоинформационных и молекулярно-генетических подходов. В работе использовали растения клещевины (Ricinus communis L.) сортов Занзибар Грин и Гибзонская. ДНК выделяли из молодых листьев. Обработку последовательностей, выравнивание и определение степени гомологии проводили в программе GenDoc (http://www.nrbsc.org/gfx/genedoc/index.html). Праймеры для амплификации промоторов гена U6 клещевины подбирали с помощью программы Primer3 (http://bioinfo.ut.ee/primer3-0.4.0/). ПЦР проводили на амплификаторе C-100 («Bio-Rad Laboratories, Inc.», США). Продукты ПЦР разделяли в 1,5 % агарозном геле при 6 В/см в камере для горизонтального электрофореза Sub-Cell GT («Bio-Rad Laboratories, Inc.», США). Ампликоны очищали с помощью набора GeneJET PCR Purification Kit («Thermo Fisher Scientific, Inc.», США). Очищенные ампликоны клонировали с помощью вектора pAL2-T («Евроген», Россия) согласно инструкции производителя. При конструировании CRISPR/Cas9 векторов использовали плазмиду pRGE31, эндонуклеазы рестрикции HindIII и SbfI («Сибэнзим», Россия) и необходимые олигонуклеотиды. При вырезании продуктов рестрикции из геля их очищали с помощью набора GeneJET Gel Extraction Kit («Thermo Fisher Scientific, Inc.», США). Был проведен биоинформационный поиск в базе генетических данных GenBank и обнаружено 12 скаффолдов, содержащих последовательность гена U6 клещевины. Шесть промоторов, включающих неповрежденные регуляторные элементы USE и ТАТА-бокс, были использованы при подборе праймеров для амплификации на матрице ДНК клещевины сортов Занзибар Грин и Гибзонская. Продукты ПЦР, состоящие из единичных фрагментов, были клонированы и секвенированы. Анализ полученных последовательностей ампликонов показал, что промоторные области полностью совпадали между сортами. Степень идентичности промоторов из разных ампликонов варьировала в пределах 51-77 %. При сравнении этих же промоторов с промоторами гена U6 других растений степень гомологии составляла 42-64 %. Последовательности промоторов, содержащие неповрежденные мотивы USE и ТАТА-бокс, были использованы для конструирования CRISPR/Cas9 векторов, которые могут применяться для эффективного редактирования генов клещевины, участвующих в синтезе рицина и рицинина.
Ключевые слова: клещевина, промотор, U6 гены, секвенирование, геномное редактирование, CRISPR/Cas9, конструирование векторов.
Клещевина (Ricinus communis L.) — это сельскохозяйственное растение семейства Молочайные (Euphorbiaceae), культивируемое во многих странах мира. Возделывается в основном ради семян, из которых получают ценный продукт — касторовое масло, используемое в различных отраслях народного хозяйства (1). Касторовое масло практически не высыхает и сохраняет свойства в широком диапазоне рабочих температур, поэтому находит применение в системе смазки машин в пищевой промышленности и двигателей для авиамоделирования, а также служит компонентом пластичных смазок (2). В нем содержатся ценные для химического синтеза вещества — ундециленовая и себациновая кислоты, компоненты эпоксидных и алкидных смол и др. В медицине касторовое масло издавна применяется как эффективное слабительное средство (3, 4).
После извлечения масла из семян клещевины остаются богатые протеином остатки — жмыхи и шроты, перспективные для использования в качестве белковых добавок при производстве кормов. Однако в этих остатках содержатся токсичные вещества — белок рицин и алкалоид рицинин (5). Рицин представляет собой кумулятивный яд, обладающий сильным местным прижигающим действием, что приводит к развитию геморрагического гастроэнтерита. После всасывания рицина в кровеносную систему происходит агглютинация и разрушение эритроцитов, возрастает риск тромбозов, изъязвляются стенки сосудов. Кроме того, рицин негативно воздействует на центральную нервную систему, вызывая судороги, парезы и параличи (6). Токсичность рицинина намного меньше, чем у рицина, но его наличие в кормах также нежелательно. Детоксификацию жмыхов и шротов клещевины проводят чаще всего с помощью жестких термических и химических обработок, что значительно ухудшает качество белка. Снижается его переваримость и усвояемость, происходит гидролиз лизина и других дефицитных аминокислот (5). В связи с этим актуален поиск альтернативных подходов к детоксификации жмыхов и шротов клещевины, которые позволили бы отказаться от традиционных обработок.
Несомненный теоретический и практический интерес представляет получение растений клещевины, в которых не вырабатывались бы рицин и рицинин. Для создания таких растений необходимо осуществить нокаут генов, участвующих в синтезе токсинов. Один из эффективных современных способов нокаутирования генов — технология геномного редактирования с помощью системы CRISPR/Cas9. Ее компонентами служат фермент Cas9 и направляющая, или гидовая, РНК (7). Гены фермента Cas9 и направляющей РНК вводятся в клетку с помощью плазмидных векторов под соответствующими промоторами. Для гена гидовой РНК обычно используют промоторы генов малых ядерных РНК U6 или U3 (8). При геномном редактировании у однодольных, как правило, используют последовательности U3 промоторов риса, в случае двудольных — U6 промоторов арабидопсиса. Однако в ряде работ было показано, что эффективность редактирования возрастает при использовании промоторов редактируемого вида, и их введение в CRISPR/Cas9 векторы стало распространенным трендом (9-13). При изучении промоторов гена U6 у разных видов было установлено, что они содержат два сайта узнавания РНК-полимеразы III — Upstream Sequence Element (USE) и TATA-бокс (11, 13, 14). Сайт USE имеет консенсусную последовательность RTCCCACATCG. На основе этой последовательности и последовательности самого гена U6 арабидопсиса C. Marshallsay с соавт. (14) предложили систему праймеров для амплификации промоторов гена U6. Эти праймеры пригодны для изучения промоторов гена U6 у других видов, геном которых еще не секвенирован.
На сегодняшний день секвенированы геномы ряда представителей семейства Euphorbiaceae (Jatropha curcas, Manihot esculentum, Hevea brasiliensis, Euphorbia esula), в том числе и геном клещевины (15-19). Также секвенированы и изучаются кодирующие рицин последовательности (20-23). Они не содержат интронов, поэтому с них транслируется большой прекурсор, называемый препрорицин, который содержит сигнальный пептид и цепи рицина A и B, разделенные линкерным пептидом из 12 аминокислот (20). Наиболее подходят в качестве мишеней для редактирования части, кодирующие сигнальный пептид или начало цепи А (23, 24).
В настоящей работе впервые проведена амплификация, секвенирование и анализ промоторов гена U6 клещевины. Найденные последовательности проанализированы и использованы в конструировании CRISPR/Cas9 векторов для дальнейшего редактирования генов рицина.
Нашей целью было изучение промоторов гена U6 клещевины с помощью биоинформационных и молекулярно-генетических подходов.
Методика. В работе были использованы растения клещевины сортов Занзибар Грин («Гавриш», Россия) и Гибзонская («Гавриш», Россия).
ДНК выделяли из молодых листьев согласно методике J.J. Doyle с соавт. (25) с некоторыми модификациями (26). Обработку последовательностей, выравнивание и определение степени гомологии проводили в программе GenDoc (http://www.nrbsc.org/gfx/genedoc/index.html).
Праймеры для амплификации промоторов гена U6 клещевины подбирали с помощью программы Primer3 (http://bioinfo.ut.ee/primer3-0.4.0/). Условия ПЦР на амплификаторе C-100 («Bio-Rad Laboratories, Inc.», США) были следующими: 5 мин при 95 °C; 30 с при 95 °C, 30 с при 50 °C, 1 мин при 72 °C (35 циклов); 10 мин при 72 °C (терминальная элонгация).
Продукты ПЦР разделяли в 1,5 % агарозном геле при 6 В/см в камере для горизонтального электрофореза Sub-Cell GT («Bio-Rad Laboratories, Inc.», США). Результаты электрофореза визуализировали с помощью системы гель-документирования Gel Doc™ XR+ («Bio-Rad Laboratories, Inc.», США). Ампликоны очищали с использованием набора GeneJET PCR Purification Kit («Thermo Fisher Scientific, Inc.», США).
Очищенные ампликоны клонировали с помощью вектора pAL2-T («Евроген», Россия) согласно инструкции производителя.
При конструировании CRISPR/Cas9 векторов использовали плазмиду pRGE31, эндонуклеазы рестрикции HindIII и SbfI («Сибэнзим», Россия) и необходимые олигонуклеотиды. При вырезании продуктов рестрикции из геля проводили очистку с помощью набора GeneJET Gel Extraction Kit («Thermo Fisher Scientific, Inc.», США).
Результаты. В базе генетических данных GenBank было обнаружено 12 скаффолдов, содержащих последовательность гена U6 клещевины, — AASG02000063, AASG02000163, AASG02000719, AASG02001323, AASG02002949, AASG02003223, AASG02003842, AASG02006600, AASG02019053, AASG02021200, AASG02020516, AASG02025904. Однако характерные USE и ТАТА элементы были выявлены в промоторной области гена U6 только у шести из этих скаффолдов — AASG02021200, AASG02020516, AASG02006600, AASG02000719, AASG02025904, AASG02002949. Сравнение полученных результатов и результатов поиска промоторов гена U6, имеющих USE и TATA-элементы, в геномах других представителей семейства Euphorbiaceae показало, что их количество может быть разным. В геномах Jatropha curcas и Manihot esculentum (сорт W14) так же, как и у клещевины, имеется 6 таких промоторов, однако у Hevea brasiliensis и Euphorbia esula их больше (соответственно 9 и 23).
Последовательности скаффолдов AASG02021200, AASG02020516, AASG02006600, AASG02000719, AASG02025904, AASG02002949, а также одного скаффолда без USE и TATA-элементов AASG02000063 использовали для подбора праймеров (табл. 1) и последующей амплификации фрагментов, содержащих up-stream область соответствующих генов U6 (рис. 1). Во всех случаях, кроме AASG02000063, амплифицировались единичные фрагменты ожидаемой длины (ампликоны Rc200, Rc516, Rc600, Rc719, Rc904 и Rc949). Поскольку в случае с AASG02000063 помимо целевого амплифицировались еще два дополнительных фрагмента, этот вариант up-stream области далее не изучался. Единичные фрагменты остальных вариантов были клонированы и секвенированы.
Анализ последовательностей ампликонов, полученных у сортов Занзибар Грин (Rc200ZG, Rc516ZG, Rc600ZG, Rc719ZG, Rc904ZG, Rc949ZG) и Гибзонская (Rc200Gib, Rc516Gib, Rc600Gib, Rc719Gib, Rc904Gib, Rc949Gib), показал, что у этих сортов промоторные области полностью совпадали между собой, а также с соответствующими последовательностями в скаффолдах AASG02021200, AASG02020516, AASG02006600, AASG02000719, AASG02025904, AASG02002949. Однако вне промоторных областей, содержащих регуляторные элементы, было обнаружено два полиморфных сайта: G/А замена в положении -228 от начала гена U6 у ампликона Rc200Gib и Т/А замена в положении -351 от начала гена U6 у Rc904Gib. Очевидно, что изучаемые промоторные области достаточно консервативны у сортов одного вида.
Похожая ситуация наблюдается и у сортов маниока AM560-2 и КU50. Промоторные области генов U6 из их скаффолдов LTYI01019634 (нуклеотиды 16035-16336) и JPQF01078381 (нуклеотиды 1216-1517), а также LTYI01021841 (нуклеотиды 27091-27392) и JPQF01070438 (нуклеотиды 16559-16860) полностью совпадают.
Сравнение секвенированных промоторов генов U6 клещевины между собой показало среднюю гомологию, варьирующую в пределах 51-77 % (табл. 2). Сравнение этих же промоторов со случайно выбранными промоторами генов U6 ряда других случайно выбранных видов растений из разных семейств демонстрировало сходные показатели степени гомологии — 42-64 % (табл. 3, 4).
Во многом выявленное сходство обеспечивалось наличием достаточно консервативных USE и ТАТА элементов, в то время как гомология остальных участков была низкой (рис. 2).
Анализ последовательностей USE у изученных промоторов показал, что у клещевины имеется несколько вариантов этого регуляторного элемента: вариант A (GааCCACATCG) из ампликонов AASG02021200ZG и AASG02021200Gib, вариант B (A-CCCACATCG) из ампликонов AASG02020516ZG и AASG02020516Gib, вариант D (AT-CCACATCG) из ампликонов AASG02000719ZG и AASG02000719Gib. В остальных трех случаях мотивы USE (С, Е и F, см. рис. 2) соответствовали консенсусной последовательности RTCCCACATCG арабидопсиса.
На основании найденных промоторов генов U6 клещевины были созданы векторные конструкции для проведения CRISPR/Cas9 редактирования гена рицина.
При конструировании CRISPR/Cas9 векторов использовали плазмиду pRGE31, содержащую ген Cas9 под CaMV 35S промотором, а также конструкцию для синтеза гидовой РНК: U3 промотор риса (OsU3), мотив из двух разнонаправленных сайтов эндонуклеазы рестрикции Bso31I (для встраивания сайта-мишени после гидролиза плазмиды этим ферментом, 2xBso31I) и последовательность, кодирующую часть гидовой РНК, которая взаимодействует с белком Cas9 (gRNAgene).
В первую очередь вели поиск сайтов для эндонуклеаз рестрикции в исходной плазмиде pRGE31 с таким расчетом, чтобы один сайт находился перед началом промотора U3, а второй — после него. В то же время оба сайта должны были отсутствовать внутри конструкции, которой предстояло лигироваться с pRGE31. В итоге были выбраны сайты HindIII и SbfI. Поскольку участок между ними полностью вмещал в себя конструкцию OsU3-2xBso31I-gRNAgene, то необходимо было синтезировать последовательность вставки, включающую 2xBso31I-gRNAgene под соответствующим промотором клещевины.
Для этого были синтезированы 6 олигонуклеотидов (forward-n), содержащих RcU6(n)-2xBso31I-(20bp)gRNAgene, где n — соответствующий вариант промотора U6 клещевины, (20bp)gRNAgene — первые 20 нуклеотидов, кодирующие начало части гидовой РНК, которая взаимодействует с белком Cas9 (табл. 5). Кроме того, был синтезирован олигонуклеотид, обратный gRNAgene (reverse). После одного цикла ПЦР (5 мин при 95 °C, 30 с при 50 °C, 10 мин при 72 °C) для каждого из олигонуклеотидов forward c олигонуклеотидом reverse были получены двуцепочечные последовательности RcU6(n)-2xBso31I-gRNAgene. Такой подход был выбран как более экономичный по сравнению с полным синтезом прямых и обратных конструкций RcU6(n)-2xBso31I-gRNAgene. К тому же он позволял осуществлять экономичный синтез конструкций с любым другим промотором при замене только прямого олигонуклеотида.
Введение в последовательность вставки сайтов рестрикции HindIII и SbfI осуществляли с помощью ПЦР. Амплификацию проводили с праймерами, подобранными на бордеры конструкций RcU6(n)-2xBso31I-gRNAgene и имеющими соответствующие сайты рестрикции в 5¢-области (см. табл. 5). Продукты ПЦР очищали, клонировали в АТ-векторе pAL2-T и секвенировали для проверки на отсутствие ошибок синтеза. После обработки плазмиды pRGE31 и плазмид pAL2-T c конструкциями RcU6(n)-2xBso31I-gRNAgene эднонуклеазами рестрикции HindIII и SbfI продукты разделяли с помощью электрофореза и вырезали целевые фрагменты. Очищенные фрагменты лигировали и использовали для трансформации компетентных клеток Escherichia coli. Отбор клонов проводили методом ПЦР с праймерами, подобранными на бордеры конструкций RcU6(n)-2xBso31I-gRNAgene. Выделенные из отобранных клонов плазмиды тестировали с помощью эндонуклеаз рестрикции HindIII (линеризует целевую плазмиду) и Bso31I (не расщепляет целевую плазмиду), после чего секвенировали с использованием праймеров на вставку (SbfI-reverse) и на ключевые регионы — ген Cas9 (35Spr-F), ген устойчивости к ампициллину (Amp-R), ori (pBR322ori-F) (см. табл. 5).
В дальнейших экспериментах созданные векторные конструкции будут сравниваться по эффективности между собой и с векторами, содержащими промотор гена U6 арабидопсиса. Проведение таких экспериментов целесообразно, поскольку на картофеле, например, было показано увеличение выхода мутантных форм при замене промотора для гена направляющей РНК с AtU6 (U6 промотор Arabidopsis thaliana, GenBank accession no. X52527.1) на StU6 (U6 промотор Solanum tuberosum, GenBank accession no. Z17290.1) в 2 раза (9).
К таким же выводам пришли L. Long с соавт. (11) в работе по оптимизации технологии CRISPR/Cas9 геномного редактирования хлопка. Экспрессия гидовой РНК при включении в векторную конструкцию CRISPR/Cas9 эндогенного промотора хлопка GhU6.3 (U6 промотор Gossypium hirsutum) вместо промотора AtU6-29 (U6 промотор Arabidopsis thaliana) увеличивалась в 6-7 раз. В результате эффективность внесения мутаций системой CRISPR/Cas9 повышалась в 4-6 раз (11). В свою очередь, X. Sun с соавт. (10) получили схожие результаты при сравнении эндогенных и экзогенных промоторов в кассетах CRISPR/Cas9 для редактирования генома сои. Установлено, что степень экспрессии гидовой РНК под эндогенным промотором GmU6 (U6 промотор Glycine max) была в 2 раза выше, чем под экзогенным промотором AtU6-26 (U6 промотор Arabidopsis thaliana). Эффективность редактирования генов в первом случае варьировала в пределах 14,7-20,2 %, во втором — 3,2-9,7 % (10). Приведенные результаты, полученные при редактировании у неродственных видов растений из разных семейств, наглядно демонстрируют преимущества использования эндогенных промоторов в векторах CRISPR/Cas9. Однако важны сравнения не только эндогенных промоторов с экзогенными, но и эндогенных промоторов друг с другом. В нашей работе были найдены различные промоторы U6 клещевины, которые имеют необходимые для нормального функционирования регуляторные элементы USE и ТАТА, и целесообразно будет провести их сравнение между собой в экспериментах по редактированию у клещевины, так как ранее отмечалось, что не все эндогенные промоторы одинаково эффективны в управлении экспрессией генов (27-30).
Таким образом, в геноме клещевины имеется шесть промоторов генов U6 с регуляторными элементами USE и ТАТА. Секвенирование найденных промоторов у сортов Занзибар Грин и Гибзонская показало их высокую консервативность. Между собой эти промоторы имеют средний уровень гомологии, как и с промоторами других видов. В найденных промоторах были обнаружены USE-элементы, которые как соответствуют (у трех промоторов), так и не соответствуют (у трех других промоторов) консенсусной последовательности USE-элемента арабидопсиса. Сконструированные на основе найденных промоторов U6 клещевины CRISPR/Cas9 станут ценными инструментами для дальнейших экспериментов по геномному редактированию генома этого вида и определению эффективности различных промоторов для синтеза направляющей РНК.
1. Праймеры, подобранные для амплификации промоторной области гена U6 у анализируемых скаффолдов клещевины (Ricinus communis L.) |
||||
Праймер |
Последовательность |
Скаффолд |
Ампликон |
Длина апликона, п.н. |
RcU6-200f |
5'-TGGATAAGAGGAGATTCTTGAATTG-3' |
AASG02021200 |
Rc200 |
392 |
RcU6-200r |
5'-AGGGGCCATGCTAATCTTCT-3' |
|||
RcU6-516f |
5'-GTTGGCAGCCTTCAGATTTC-3' |
AASG02020516 |
Rc516 |
590 |
RcU6-516r |
5'-AGGGGCCATGCTAATCTTCT-3' |
|||
RcU6-600f |
5'-CTCCGAATTATATTTGGGGTTTT-3 |
AASG02006600 |
Rc600 |
280 |
RcU6-600r |
5'-AAAAATTTGGACCATTTCTCG-3' |
|||
RcU6-719f |
5'-ACCTGTGAGGTGGCTTTCTG-3' |
AASG02000719 |
Rc719 |
346 |
RcU6-719r |
5'-CGGTGTCTGTTTGCCCTAAT-3' |
|||
RcU6-904f |
5'-AGCCCATTTTGGGGTGTTAT-3' |
AASG02025904 |
Rc904 |
512 |
RcU6-904r |
5'-AGGGGCCATGCTAATCTTCT-3' |
|||
RcU6-949f |
5'-TTCTGGGAGGTATGCATCAA-3' |
AASG02002949 |
Rc949 |
401 |
RcU6-949r |
5'-AAAAATTTGGACCATTTCTCG-3' |
|||
RcU6-063f |
5'-CAACCCGACTCCTTCATCAT-3' |
AASG02000063 |
Rc063 |
584 |
RcU6-063r |
5'-AGGGGCCCTACTTTGATACC-3' |
|||
2. Степень гомологии (%) между шестью выбранными промоторами U6 генов клещевины (Ricinus communis L.) |
||||||
Промотор |
Rc200ZG |
Rc516ZG |
Rc600ZG |
Rc719ZG |
Rc904ZG |
Rc949ZG |
Rc200ZG |
100 |
56 |
53 |
77 |
57 |
50 |
Rc516ZG |
100 |
63 |
57 |
67 |
62 |
|
Rc600ZG |
100 |
54 |
63 |
54 |
||
Rc719ZG |
100 |
53 |
51 |
|||
Rc904ZG |
100 |
59 |
||||
Rc949ZG |
100 |
|||||
3. Нуклеотидные последовательности случайно выбранных промоторов у видов растений из разных семейств, использованные для |
||||
Шифр |
Последовательность |
GenBank acc. no. |
Первый нуклеотид |
Последний нуклеотид |
abrus1 |
5´-GCCCCACATCGAACAGTATTATCAAAGCATGACACAATATATAGCAAAAGAAACACGCAGAGAGT-3´ |
XM_027482675 |
708 |
644 |
arachis1 |
5´-GTACCACATCGAGTAGCATCATATAACTCTGACAATATATATAGCAGAGGGTGCAAAGAAGGCTC-3´ |
XM_025790263 |
575 |
511 |
arachis2 |
5´-GTCCCACATCGCTTAGTATCAGACCACTCTGACAGAATATATATCAAAGGAAACACAAAAGGCTC-3´ |
XR_003810923 |
117 |
181 |
arachis4 |
5´-GTCTCACATCGCCCGAGTTTTGAGAAACCAATAACTTATATATCAGAGGCGAAGCAAAGGCTC-3´ |
XM_025845620 |
218 |
281 |
bras_ol1 |
5´-GTCCCACATCGCTCAGGTGAAGAGAAGGAGCTGCGTTTATATAGCGATGAAGTCACGAAAGTGATT-3´ |
LR031877 |
48176336 |
48176271 |
bras_rap1 |
5´-CTCCCACATCGCTCAGCGAAGCAAAAGAAGCTCCTGTTTATATACTTTCAGAGTCAAGAAGATGATT-3´ |
LR031575 |
1103139 |
1103205 |
bras_rap2 |
5´-CTCCCACATCGTTTATCAGAGAAGCAGAAGCCGAGTTTATATAGGGACGGAGTGACGAAGGAGATT-3´ |
LR031575 |
1143184 |
1143119 |
cicer1 |
5´-GTCCCACATCGAATACATGTATCCCATTTTCCATATTTATATAACGCAGGTTAACCATGCAGTAT-3´ |
CP039335 |
30486401 |
30486452 |
cicer2 |
5’-GTCCCACATCGAATACATTTATCCCTTTTTCCGTATTTATATAACGCAGGTTAACCATGGAGTTT-3´ |
CP039335 |
30494676 |
30494740 |
cicer3 |
5´-GTCCCACACCGAATCATCTATCATTTTTTTCGTCTTTATATAACCCATGTTAATCATTAGGTTT-3´ |
CP039335 |
30512524 |
30512587 |
cicer4 |
5´-GTCCCACATCGTCTAAATATTCGAATATTTAATATTTATATACAATGTTCGAGCAGTATAGTAT-3´ |
CP039333 |
18318744 |
18318807 |
cicer7 |
5´-GTCCCACACCGCGTACGCATAACATGTGTTCAGTGTTTATAATACCCTCGCACACATCATCAAC-3´ |
CP039333 |
31682334 |
31682398 |
cicer8 |
5´-GTTCCACATCGTCTACATCTATCATTATTTACGTCTTTATATTCAACCGATGAGCCATAAGGCTT-3´ |
CP039333 |
39009683 |
39009747 |
cichorium1 |
5´-CGTCCCATACCGACCAGTAAAGTACTTCCCGTCGCCTTATATAGCGCAGCTCGGCGACTATCATC-3´ |
MK455779 |
235 |
299 |
cichorium2 |
5´-GTCCCATACCGACCAGTAAAGTACTTCCCGTCGCCTTATATAGCGCAGCTCGGCGACTATCATC-3´ |
MK455779 |
236 |
299 |
cichorium3 |
5´-TTCCCACATCGCTCTTTGAAGCAACATCGCCATGCTTTATATAGCTTGGCTTCCAAACATATATC-3´ |
MK455773 |
235 |
299 |
cichorium4 |
5´-TTCCCACATCGATGATTGAAACGATTCCTCGGTGTTTTATATAGCCTGGCTTCCAATCAAATATC-3´ |
MK455776 |
238 |
302 |
cichorium5 |
5´-CTCCCACATCGATGATCGGAACGGTTGTTTCGTGCTTTATATAGCTCGGGTTCCAACCATTTATC-3´ |
MK455775 |
238 |
302 |
citrullus1 |
5´-GTCCCACATCGGTAAGTTTTGATTCTAGTTTACGCTTTATATAACTAAGACTGCAGTACAAGGCTT-3´ |
VOOL01000005 |
2250542 |
2250477 |
cynara1 |
5´-AATCCCACATCGCCTTTAACGATATCCAGTGCTAGCTTTATATGGCGGAGGTCGGCAGCTAAGATC-3´ |
XR_003069239 |
1 |
65 |
goss_raim1 |
5´-ATCCCATATCGCTAAAGAACTATAACACAGGAGCGTTTATATAAGCGAAAGAAGCAGCAAATGATT-3´ |
CP032562 |
3130166 |
3130101 |
goss_raim2 |
5´-ATCCCACATAGCTAAAGAATTAGGAAAATTTATTGTTTATAAAGGCAAAGGAAGAAACTTATTATT-3´ |
CP032562 |
3328533 |
3328468 |
goss_raim3 |
5´-ATCCCGCATCGCTAAAGAATTGAAAAAATTTTATTGTTTATATAGGAAAAACAAGCTGACTATGATT-3´ |
CP032562 |
3351518 |
3351452 |
goss_raim4 |
5´-ATCCCGCATCGCTAAAGAATTGAAAAAATTTTATTGTTTATATAGGAAAAACAAGCTGACTATGATT-3´ |
CP032562 |
3351518 |
3351452 |
gossipioides1 |
5´-ATCCCACATCGCTAAAGAACTAAAATGCCGAAGTATTTATATAAGCGAAAGAAACAGCATTAGTGT-3´ |
CP032252 |
2333488 |
2333423 |
gossipioides2 |
5´-ATCCCACATTGCTAAAGAATTAACAAATACTATTGTTTATATAGGCAAAAGAAACACCGTAGCAGT-3´ |
CP032252 |
2341665 |
2341600 |
gossipioides3 |
5´-ATCCCACATCTCTAAAGAATTAAAAAACACTATTGTTTATATGGGCAAAAGAAGCACCGTTGTATT-3´ |
CP032252 |
2343934 |
2343869 |
ipomea1 |
5´-CTCCCACATCGGGCGATGAAGCAGCTCTCTTCCAGTACACATACTCCGCCATTGAAGAAGAAGAAC-3´ |
CP025668 |
7620112 |
7620047 |
ipomea2 |
5´-CTCCCACATCGGCCAATGAGCCATCTTACTTCCAGTACATATACTCCGCCATGGAAGCTCTTATC-3´ |
XR_004100417 |
33 |
97 |
ipomea3 |
5´-CTCCCACATCGGCTGATGAAACAACTTGCTTCCAGTATACATACTCTATCATGGAAGCACTGAGC-3´ |
XM_031272161 |
1 |
65 |
ipomea4 |
5´-GTCCCACATCGGCCAATGAGCCATCTTACTTCCAGTACATATACTCCGCCATTGGAGCACTTAGC-3´ |
CP025668 |
7980996 |
7981060 |
ipomea5 |
5´-TTCCCACATCGGGCGATGAAGCAGCTCTCTTCCATTACACATACTCCGCCATTGAGGAAGGAGAAT-3´ |
CP025668 |
7984209 |
7984274 |
lotus1 |
5´-GTCCCACACCGGATAAACATACAGAAATATGAGTGTTTATAAGCAAATAGTCAGCAATAAGGTTC-3´ |
AP010923 |
70411 |
70347 |
lotus2 |
5´-GTTCCACATCGGCTATGTTGATTAAGATTTTATAGTTCATATATCACTACAGAACAGCAAGTATT-3´ |
AP010923 |
68417 |
68353 |
tea1 |
5´-GTCCCACATCGAAACTTCGACGTTATAGACATGGAGTTTATAAGAAAGAAGAAAAGAGAAGACGTT-3´ |
XR_003649102 |
150 |
215 |
tomato1 |
5´-CTCCCTCATCGCTTACAGAAAAAAGCTATATGCTGTTTATATTGCGAAATCTAACAGTGTAGTTT-3´ |
XM_004230407 |
25 |
89 |
tomato2 |
5´-CTCCCTCATCGCTTACAGAAAAAAGCTATATGCTGTTTATATTGCGAATCTAACAGTGTAGTTT-3´ |
X51447 |
198 |
261 |
vigna1 |
5´-GTCCCACATCGTCCAAACATGTCACAACTTCCATGTTTAAAAACGCACGCCTACTCGCTGCTGTT-3´ |
CP039354 |
53496613 |
53496677 |
vigna2 |
5´-GTCCCACACCGTATACTTTCACTAGAGGTTTAGTGTTTATATAGATACAGACTGCATCCAAGCTT-3´ |
CP039350 |
33742544 |
33742608 |
4. Степень гомологии (%) между шестью выбранными промоторами U6 генов клещевины (Ricinus communis L.) |
||||||
Шифр |
Промотор |
|||||
Rc200ZG |
Rc516ZG |
Rc600ZG |
Rc719ZG |
Rc904ZG |
Rc949ZG |
|
abrus1 |
48 |
53 |
56 |
46 |
60 |
62 |
arachis1 |
52 |
53 |
57 |
47 |
55 |
49 |
arachis2 |
54 |
57 |
57 |
51 |
56 |
58 |
arachis4 |
54 |
54 |
63 |
52 |
49 |
54 |
bras_ol1 |
53 |
54 |
57 |
48 |
52 |
53 |
bras_rap1 |
54 |
55 |
57 |
56 |
54 |
52 |
bras_rap2 |
45 |
54 |
54 |
46 |
52 |
54 |
cicer1 |
56 |
54 |
53 |
53 |
55 |
52 |
cicer2 |
58 |
52 |
53 |
57 |
55 |
49 |
cicer3 |
59 |
53 |
50 |
54 |
51 |
46 |
cicer4 |
50 |
56 |
59 |
53 |
56 |
55 |
cicer7 |
47 |
64 |
53 |
50 |
50 |
55 |
cicer8 |
51 |
57 |
58 |
54 |
55 |
55 |
cichorium1 |
42 |
47 |
55 |
45 |
47 |
50 |
cichorium2 |
43 |
48 |
55 |
46 |
48 |
51 |
cichorium3 |
55 |
53 |
50 |
52 |
49 |
52 |
cichorium4 |
49 |
48 |
50 |
51 |
50 |
45 |
cichorium5 |
55 |
51 |
54 |
55 |
47 |
44 |
citrullus1 |
57 |
60 |
59 |
58 |
56 |
59 |
cynara1 |
52 |
55 |
63 |
55 |
56 |
51 |
goss_raim1 |
48 |
62 |
57 |
51 |
61 |
59 |
goss_raim2 |
47 |
58 |
53 |
50 |
58 |
56 |
goss_raim3 |
51 |
61 |
50 |
52 |
65 |
63 |
goss_raim4 |
50 |
46 |
50 |
52 |
65 |
63 |
gossipioides1 |
49 |
55 |
57 |
54 |
62 |
58 |
gossipioides2 |
51 |
60 |
54 |
51 |
62 |
61 |
gossipioides3 |
52 |
60 |
57 |
52 |
60 |
60 |
ipomea1 |
42 |
52 |
47 |
46 |
44 |
47 |
ipomea2 |
48 |
58 |
52 |
44 |
50 |
49 |
ipomea3 |
46 |
58 |
54 |
43 |
52 |
52 |
ipomea4 |
50 |
58 |
51 |
48 |
48 |
49 |
ipomea5 |
45 |
52 |
47 |
43 |
47 |
47 |
lotus1 |
46 |
55 |
58 |
52 |
63 |
59 |
lotus2 |
59 |
59 |
62 |
59 |
56 |
58 |
tea1 |
57 |
53 |
56 |
58 |
57 |
58 |
tomato1 |
48 |
47 |
53 |
48 |
53 |
51 |
tomato2 |
48 |
47 |
53 |
47 |
52 |
50 |
vigna1 |
52 |
60 |
57 |
46 |
53 |
52 |
vigna2 |
55 |
66 |
56 |
53 |
61 |
55 |
5. Олигонуклеотиды, использованные при создании и тестировании CRISPR/Cas9 векторов с промоторами гена U6 клещевины |
|
Праймер |
Последовательность праймера |
Олигонуклеотиды для синтеза двуцепочечных конструкций RcU6(n)-2xBso31I-gRNAgene |
|
forward-A |
5'-GAACCACATCGATTCGTTGTAGCTTTTTAGAATTCTTTATATGAATTAGGGCAAACAGCTGGTTGGGGAGACCGAGGTCTCGGTTTTAGAGCTAGAAATAGC-3' |
forward-B |
5'-AACCCACATCGTCTAGTTACGCATAACTTTAGAGTTTATAAACGCCTACAAGCAAGCACCATGCTGGAGACCGAGGTCTCGGTTTTAGAGCTAGAAATAGC-3' |
forward-C |
5'-GTCCCACATCGTCTGGTTACGTGAGACTAATGATCCTTATATGCAAACGGGAGGCAGCATGATGCTGGAGACCGAGGTCTCGGTTTTAGAGCTAGAAATAGC-3' |
forward-D |
5'-AATCCACATCGGTTATTTGTCGGTCTTTAGAAGTCTTTATATGAATTAGGGCAAACAGACACCGTTGGAGACCGAGGTCTCGGTTTTAGAGCTAGAAATAGC-3' |
forward-E |
5'-ATCCCACATCGTTTAGTTACATAAAATATTAGACTTTATAAGAAAATCAAGCTAGCTTCTGAAGTGGAGACCGAGGTCTCGGTTTTAGAGCTAGAAATAGC-3' |
forward-F |
5'-ATCCCACATCGACAAGGAATACAAATTTAAAACGGTTTATAAGTAAACAAAAACGACATGCTACTGGAGACCGAGGTCTCGGTTTTAGAGCTAGAAATAGC-3' |
reverse |
5'-AAAACAAAAAAGCACCGACTCGGTGCCACTTTTTCAAGTTGATAACGGACTAGCCTTATTTTAACTTGCTATTTCTAGCTCTAAAAC-3' |
Олигонуклеотиды для введения в RcU6(n)-2xBso31I-gRNAgene сайтов рестрикции |
|
HindIII-A |
5'-AAGCTTGAACCACATCGATTC-3' |
HindIII-B |
5'-AAGCTTAACCCACATCGTCTA-3' |
HindIII-C |
5'-AAGCTTGTCCCACATCGTCTG-3' |
HindIII-D |
5'-AAGCTTAATCCACATCGGTTA-3' |
HindIII-E |
5'-AAGCTTATCCCACATCGTTTA-3' |
HindIII-F |
5'-AAGCTTATCCCACATCGACAA-3' |
SbfI-reverse |
5'-CCTGCAGGAAAACAAAAAAGCAC-3' |
Олигонуклеотиды для проверки векторов |
|
35Spr-F |
5'-CTATCCTTCGCAAGACCCTTC-3' |
Amp-R |
5'-ATAATACCGCGCCACATAGC-3' |
pBR322ori-F |
5'-GGGAAACGCCTGGTATCTTT-3' |
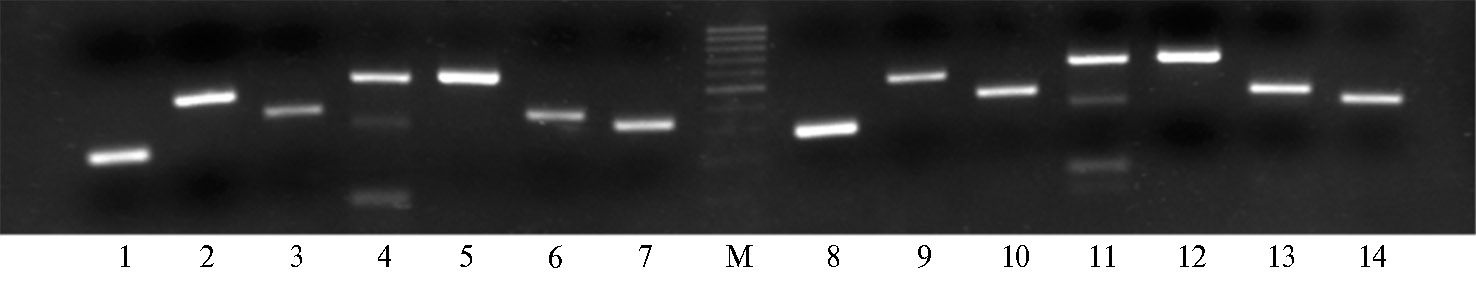
Рис. 1. Электрофореграмма продуктов ПЦР-амплификации на матрице ДНК клещевины (Ricinus communis L.) сортов Занзибар Грин (1-7) и Гибзонская (8-14) при использовании праймеров, подобранными для амплификации промоторной области гена U6: 1 и 8 — Rc600f/Rc600r, 2 и 9 — Rc904f/Rc904r, 3 и 10 — Rc949f/Rc949r, 4 и 11 — Rc063f/Rc063r, 5 и 12 — Rc516f/Rc516r, 6 и 13 — Rc200f/Rc200r, 7 и 14 — Rc719f/Rc719r; М — маркер молекулярных масс с шагом 100 п.н. (100 bp DNA Ladder, M-214S, «Jena Biosience GmbH», Германия).

Рис. 2. Выравнивание вариантов промоторов гена U6 клещевины (Ricinus communis L.): А — промотор из ампликонов AASG02021200ZG и AASG02021200Gib, B — из ампликонов AASG02020516ZG и AASG02020516Gib, С — из ампликонов AASG02006600ZG и AASG02006600Gib, D — из ампликонов AASG02000719ZG и AASG02000719Gib, Е — из ампликонов AASG02025904ZG и AASG02025904Gib, F — из ампликонов AASG02002949ZG и AASG02002949Gib. Красным цветом выделены варианты USE, которые совпадают с консенсусной последовательностью RTCCCACATCG арабидопсиса. Зеленым цветом выделена часть гена U6, которая амплифицируется с праймера Oligo3 (14).
ЛИТЕРАТУРА
- Мошкин В.А. Клещевина. М., 1980.
- Азнаурьян М.П., Елошвили Н.Г., Назаров Н.В., Якубович H.H., Мещеряков C.B., Гирсин Ю.А. Использование отходов масложирового производства для приготовления пластичных смазок. Известия вузов, Пищевая технология, 1993, 1-2(212-213): 87-89.
- Naughton F.C. Production, chemistry and commercial applications of various chemicals from castor oil. Journal of the American Oil Chemists' Society, 1974, 51(3): 65-71 (doi: 10.1007/BF00000015).
- Шульпекова Ю.О. Запор и методы его лечения. Приложение РМЖ «Болезни органов пищеварения», 2006, 8(2): 90-96.
- Anandan S., Kumar G.K.A., Ghosh J., Ramachandra K.S. Effect of different physical and chemical treatments on detoxification of ricin in castor cake. Animal Feed Science and Technology, 2005, 120(1-2): 159-168 (doi: 10.1016/j.anifeedsci.2004.10.002).
- Гусынин И.А. Токсикология ядовитых растений. М., 1962.
- Ishino Y., Krupovic M., Forterre P. History of CRISPR-Cas from encounter with a mysterious repeated sequence to genome editing technology. Journal of Bacteriology, 2018, 200(7): e00580-17 (doi: 10.1128/JB.00580-17).
- Злобин Н.Е., Терновой В.В., Гребенкина Н.А., Таранов В.В. Сделать сложное проще: современный инструментарий для редактирования генома растений. Вавиловский журнал генетики и селекции, 2017, 21(1): 104-111 (doi: 10.18699/VJ17.228).
- Andersson M., Turesson H., Nicolia A., Fält A.S., Samuelsson M., Hofvander P. Efficient targeted multiallelic mutagenesis in tetraploid potato (Solanum tuberosum) by transient CRISPR-Cas9 expression in protoplasts. Plant Cell Reports, 2017, 36(1): 117-128 (doi: 10.1007/s00299-016-2062-3).
- Sun X., Hu Z., Chen R., Jiang Q., Song G., Zhang H., Xi Y. Targeted mutagenesis in soybean using the CRISPR-Cas9 system. Scientific Reports, 2015, 5: 10342 (doi: 10.1038/srep10342).
- Long L., Guo D.D., Gao W., Yang W.W., Hou L.P., Ma X.N., Miao Y.C., Botella J.R., Song C.P. Optimization of CRISPR/Cas9 genome editing in cotton by improved sgRNA expression. Plant Methods, 2018, 14: 85 (doi: 10.1186/s13007-018-0353-0).
- Yan P.U., Chao L.I.U., JiYang L.I., TaShi А., Yan H.U., XiaoDong L.I.U. Different SlU6 promoters cloning and establishment of CRISPR/Cas9 mediated gene editing system in tomato. Scientia Agricultura Sinica, 51(2): 315-326.
- Bernard G., Gagneul D., Dos Santos H.A., Etienne A., Hilbert J.-L., Rambaud C. Efficient genome editing using CRISPR/Cas9 technology in chicory. International Journal of Molecular Sciences, 2019, 20(5): 1155 (doi: 10.3390/ijms20051155).
- Marshallsay C., Kiss T., Filipowicz W. Amplification of plant U3 and U6 snRNA gene sequences using primers specific for an upstream promoter element and conserved intragenic regions. Nucleic Acids Research, 1990, 18(12): 3459-3466 (doi: 10.1093/nar/18.12.3459 ).
- Chan A.P., Crabtree J., Zhao Q., Lorenzi H., Orvis J., Puiu D., Melake-Berhan A., Jones K.M., Redman J., Chen G., Cahoon E.B., Gedil M., Stanke M., Haas B.J., Wortman J.R., Fraser-Liggett C.M., Ravel J., Rabinowicz P.D. Draft genome sequence of the oilseed species Ricinus communis. Nature Biotechnology, 2010, 28: 951-956 (doi: 10.1038/nbt.1674).
- Horvath D.P., Patel S., Doğramaci M., Chao W.S., Anderson J.V., Foley M.E., Scheffler B., Lazo G., Dorn K., Yan C., Childers A., Schatz M., Marcus S. Gene space and transcriptome assemblies of leafy spurge (Euphorbia esula) identify promoter sequences, repetitive elements, high-quality markers, and a full-length chloroplast genome. Weed Science, 2018, 66(3): 355-367 (doi: 10.1017/wsc.2018.2).
- Rahman A.Y., Usharraj A.O., Misra B.B., Thottathil G.P., Jayasekaran K., Feng Y., Hou S., Ong S.Y., Ng F.L., Lee L.S., Tan H.S., Sakaff M.K., Teh B.S., Khoo B.F., Badai S.S., Aziz N.A., Yuryev A., Knudsen B., Dionne-Laporte A., Mchunu N.P., Yu Q., Langston B.J., Freitas T.A., Young A.G., Chen R., Wang L., Najimudin N., Saito J.A., Alam M. Draft genome sequence of the rubber tree Hevea brasiliensis. BMC Genomics, 2013, 14: 75 (doi: 10.1186/1471-2164-14-75).
- Wang W., Feng B., Xiao J., Xia Z., Zhou X., Li P., Zhang W., Wang Y., Møller B.L., Zhang P., Luo M.C., Xiao G., Liu J., Yang J., Chen S., Rabinowicz P.D., Chen X., Zhang H.B., Ceballos H., Lou Q., Zou M., Carvalho L.J., Zeng C., Xia J., Sun S., Fu Y., Wang H., Lu C., Ruan M., Zhou S., Wu Z., Liu H., Kannangara R.M., Jørgensen K., Neale R.L., Bonde M., Heinz N., Zhu W., Wang S., Zhang Y., Pan K., Wen M., Ma P.A., Li Z., Hu M., Liao W., Hu W., Zhang S., Pei J., Guo A., Guo J., Zhang J., Zhang Z., Ye J., Ou W., Ma Y., Liu X., Tallon L.J., Galens K., Ott S., Huang J., Xue J., An F., Yao Q., Lu X., Fregene M., López-Lavalle L.A., Wu J., You F.M., Chen M., Hu S., Wu G., Zhong S., Ling P., Chen Y., Wang Q., Liu G., Liu B., Li K., Peng M. Cassava genome from a wild ancestor to cultivated varieties. Nature Communications, 2014, 5: 5110 (doi: 10.1038/ncomms6110).
- Wu P., Zhou C., Cheng S., Wu Z., Lu W., Han J., Chen Y., Chen Y., Ni P., Wang Y., Xu X., Huang Y., Song C., Wang Z., Shi N., Zhang X., Fang X., Yang Q., Jiang H., Chen Y., Li M., Wang Y., Chen F., Wang J., Wu G. Integrated genome sequence and linkage map of physic nut (Jatropha curcas L.), a biodiesel plant. Plant Journal, 2015, 81(5): 810-821 (doi: 10.1111/tpj.12761).
- Halling K.C., Halling A.C., Murray E.E., Ladin B.F., Houston L.L., Weaver R.F. Genomic cloning and characterization of a ricin gene from Ricinus communis. Nucleic Acids Research, 1985, 13(22): 8019-8033 (doi: 10.1093/nar/13.22.8019).
- Tregear J.W., Roberts L.M. The lectin gene family of Ricinus communis: cloning of a functional ricin gene and three lectin pseudogenes. Plant Molecular Biology, 1992, 18(3): 515-525 (doi: 10.1007/BF00040667).
- Ladin B.F., Murray E.E., Halling A.C., Halling K.C., Tilakaratne N., Long G.L., Houston L.L., Weaver R.F. Characterization of a cDNA encoding ricin E, a hybrid ricin-Ricinus communis agglutinin gene from the castor plant Ricinus communis. Plant Molecular Biology, 1987, 9(3): 287-295 (doi: 10.1007/BF00166464).
- Alexandrov O. Study of the upstream ricin gene sequences in different castor (Ricinus communis) varieties as a preliminary step in CRISPR/Cas9 editing. Research on Crops, 2020, 21(2): 344-348 (doi: 10.31830/2348-7542.2020.058).
- Александров О.С. Биоинформатический анализ гена рицина Ricinuscommunis L. и поиск участков-мишеней для его редактирования с помощью технологии CRISPR/Cas9. Актуальная биотехнология, 2018, 3(26): 161-162.
- Doyle J.J., Doyle J.L. Isolation of plant DNA from fresh tissue. Focus, 1990, 12(1): 13-15.
- Razumova O.V., Alexandrov O.S., Divashuk M.G., Sukhorada T.I., Karlov G.I. Molecular cytogenetic analysis of monoecious hemp (Cannabis sativa L.) cultivars reveals its karyotype variations and sex chromosomes constitution. Protoplasma, 2016, 253(3): 895-901 (doi: 10.1007/s00709-015-0851-0).
- Wang M.B., Helliwell C.A., Wu L.M., Waterhouse P.M., Peacock W.J., Dennis E.S. Hairpin RNAs derived from RNA polymerase II and polymerase III promoter-directed transgenes are processed differently in plants. RNA, 2008, 14(5): 903-913 (doi: 10.1261/rna.760908).
- Mikami M., Toki S., Endo M. Comparison of CRISPR/Cas9 expression constructs for efficient targeted mutagenesis in rice. Plant Molecular Biology, 2015, 88(6): 561-572 (doi: 10.1007/s11103-015-0342-x).
- Domitrovich A.M., Kunkel G.R. Multiple, dispersed human U6 small nuclear RNA genes with varied transcriptional efficiencies. Nucleic Acids Research, 2003, 31(9): 2344-2352 (doi: 10.1093/nar/gkg331).
- Wang P., Zhang J., Sun L., Ma Y., Xu J., Liang S., Deng J., Tan J., Zhang Q., Tu L., Daniell H., Jin S., Zhang X. High efficient multisites genome editing in allotetraploid cotton (Gossypium hirsutum) using CRISPR/Cas9 system. Plant Biotechnology Journal, 2018, 16(1): 137-150 (doi: 10.1111/pbi.12755).