УДК 633.8:575.113:575.174.015.3:57.088
ОЦЕНКА ВНУТРИ- И МЕЖПОПУЛЯЦИОННОГО ГЕНЕТИЧЕСКОГО РАЗНООБРАЗИЯ ЛЕКАРСТВЕННОГО ВИДА Adonis vernalis L. С ИСПОЛЬЗОВАНИЕМ IRAP-МАРКЕРОВ
С.В. БОРОННИКОВА, Н.Н. ТИХОМИРОВА, О.А. КРАВЧЕНКО
У растений шести популяций редкого для Пермского края лекарственного вида Adonis vernalis L. IRAP-методом изучили полиморфизм ДНК и оценили генетическое разнообразие по 127 выявленным IRAP-маркерам. Среди факторов, обусловливающих высокую степень генетической изменчивости, реликтовость вида, вероятно, имеет решающее значение.
Ключевые слова: IRAP-маркеры, полиморфизм, генетическое разнообразие, охрана генофондов, отбор с помощью молекулярных маркеров.
В настоящее время насчитывается более 15 различных типов маркеров (1-3), используемых для молекулярно-генетического анализа генома, в частности у редких и находящихся под угрозой исчезновения видов, а также для выявления фармацевтического потенциала лекарственных растений. Значительная часть генома растений представлена повторяющимися последовательностями, к которым относятся микросателлиты и мобильные генетические элементы ретротранспозоны. В некоторых случаях на ретротранспозоны может приходиться более 70 % ядерного генома (4). IRAP-метод (inter-retrotransposon amplified polymorphism — анализ полиморфных участков ДНК, амплифицированных между ретротранспозонами) наиболее эффективно выявляет полиморфизм ДНК (5) и успешно применялся для филогенетических исследований и изучения биоразнообразия у видов Hordeum (5, 6), зерновых культур (7), сортов Oryza (8), Pisum (9), редких и ресурсных видов растений при антропогенных воздействиях (10, 11).
Adonis vernalis L. (адонис весенний, сем. Ranunculaceae Juss.) относится к высокодекоративным лекарственным видам, внесен в Красную книгу Пермского края с категорией редкости 3 — редкий (12) и является ксеротермическим реликтом (13). A. vernalis содержит гликозиды сердечной группы, широко используется при лечении сердечно-сосудистой недостаточности и неврологических заболеваний (14).
Целью работы был анализ генетического разнообразия природных популяций редкого лекарственного вида растений Adonis vernalis с помощью выявленных IRAP-маркеров.
Методика. Объектом исследований, проводившихся в 2002-2008 годах, служили растения A. vernalis шести популяций — Av1, Av2, Av3, Av4, Av5 и Av6, произрастающие в Пермском крае соответственно около с. Михино Ординского района, на Спасской горе около с. Плеханово Кунгурского района, около дер. Мерекай Ординского района, около дер. Иштеряки, около с. Богородск и около дер. Ишимово Октябрского района. С 30 случайно выбранных растений из каждой популяции, находящихся на расстоянии 30-50 м друг от друга, собирали фрагменты листьев, из которых выделяли ДНК методом A.M. Torres с соавт. (15) с незначительными модификациями. Проанализировали 186 проб ДНК. Для выявления генетического полиморфизма ДНК применяли IRAP-метод (5). Нуклеотидные последовательности ДНК определяли на капиллярном секвенаторе ABI3700 («Biosystems», США), дизайн праймеров проводили в лаборатории растительной геномики Института биотехнологии Университета г. Хельсинки (Финляндия) (11), синтез 70 праймеров — в «MWG Biotech AG» (Германия). Выполняли подбор молекулярных маркеров, условий проведения полимеразной цепной реакции (ПЦР), апробацию праймеров. Амплификацию ДНК осуществляли в термоциклерах MJ Mini-Cycler («Bio-Rad», США) и «Терцик» (НПФ «ДНК-Технология», Россия) по следующему протоколу: предварительная денатурация — 95 °C, 3 мин; 30 циклов при 95 °С по 20 с; отжиг — 60 °С, 1 мин; элонгация — 68 °C, 1 мин, завершающая элонгация — 72 °C, 5 мин. Температура отжига в зависимости от состава праймеров по G/C варьировала от 55 до 68 °С. Повторность опыта по каждому образцу — не менее чем 2-кратная. Амплифицированные продукты разделяли электрофорезом в 1,7 % агарозном геле с бромистым этидием. Гели сканировали в системе Gel-Doc («Bio-Rad», США). Для определения длины фрагментов ДНК использовали маркер молекулярной массы (100 bp + 1.5 + 3 Кb DNA Ladder, «ООО-СибЭнзим-М», Россия) (программа Quantity One, фирма «Bio-Rad», США).
С помощью компьютерных программ POPGENE1.31 (16) и GenAlEx6 (17) оценивали долю полиморфных локусов (P95) (18), общее (Na) и эффективное (Ne) число аллелей (19), ожидаемую гетерозиготность (He) (20). Подразделенность популяций анализировали по индексу Шеннона (21, 22) и показателю подразделенности популяций (Gst) (23), для расчета которого определяли общее генное разнообразие в суммарной выборке (HT) и среднее выборочное генное разнообразие по всем локусам (Hs). На основе матриц бинарных признаков получили матрицы генетических различий (24), использовавшиеся далее для построения дендрограмм невзвешенным парно-групповым методом (UPGMA — unweighed pair-grup method using arithmetic average) при помощи компьютерной программы POPGENE1.31. Статистическую обработку данных молекулярно-генетического анализа проводили с применением стандартных для популяционно-генетических исследований методов (25-27).
Результаты. На редких реликтовых видах растений апробированы три метода IRAP-анализа полиморфизма ДНК (28-30). Для популяционно-генетических исследований редких видов растений, по нашему мнению, бо-
| 1. Характеристика праймеров и амплифицированых с их помощью фрагментов ДНК у растений шести популяций Adonis vernalis(Пермский край) | ||||
IRAP- |
Нуклеотидная последовательность |
Размер фраг-ментов, п.н. |
Число фрагментов |
|
учитываемых |
полиморфных |
|||
2175 |
TTAGACCCGGAACCGCCGTG |
2400-190 |
24 |
23 (0,9583) |
2198 |
ATCCTTCGCGTAGATCAAGCGCCG |
2470-310 |
30 |
26 (0,8666) |
2202 |
TGGCGCTTGATCTACGCGAAGGA |
1650-300 |
19 |
17 (0,8947) |
2200 |
ATGTGACAGTCGACTAACCAC |
2500-360 |
23 |
22 (0,9565) |
2197 |
GAAGTACCGATTTACTTCCGTGTA |
2430-340 |
31 |
30 (0,9677) |
Всего |
|
|
127 |
118 (0,9291) |
лее эффективно применение единичного праймера из областей LTR (long terminal repeats) (11), когда продукты амплификации образуются между двумя инвертированными LTR с одинаковой последовательностью. В этом случае при превышении центральной частью ретротранспозона обычной длины продуктов ПЦР (около 3 п.н.) ПЦР будет проходить только между двумя соседними инвертированными LTR (5).
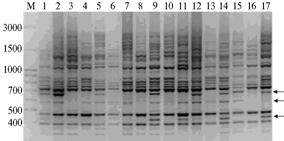 |
Рис. 1. IRAP-спектр, полученный с праймером 2197 и пробами ДНК растений Adonis vernalis из популяции Av5 (Пермский край, описание популяций см. в разделе «Методика»): 1-17 — номера проб, М — маркер молекулярной массы (100 bp + 1.5 + 3 Кb DNA Ladder, «ООО-СибЭнзим-М», Россия). Стрелками обозначены некоторые полиморфные фрагменты ДНК. |
Из 70 праймеров мы отобрали пять наиболее эффективных для A. vernalis (29). С их помощью в изученных популяциях A. vernalis выявили 127 фрагментов ДНК, из которых 118 были полиморфными. Число амплифицированных фрагментов ДНК в суммарной выборке растений варьировало от 19 до 31 в зависимости от праймера (соответственно праймеры 2202 и 2197) (табл. 1). В среднем при IRAP-анализе один праймер инициировал синтез 25 фрагментов ДНК. Число полиморфных фрагментов в суммарной выборке растений A. vernalis варьировало от 17 до 30, их размер — от 190 до 2500 п.н. (рис. 1). Доля полиморфных фрагментов ДНК в суммарной выборке в зависимости от IRAP-праймера колебалась от 0,8666 до 0,9677 и в расчете на выборку составила 0,9291.
| 2. Показатели генетического разнообразия, оцененного IRAP-методом, в популяциях Adonis vernalis (Пермский край) | |||||||
Показа- |
Av1 |
Av2 |
Av3 |
Av4 |
Av5 |
Av6 |
На выборку |
N |
89 |
86 |
97 |
112 |
110 |
100 |
127 |
P95 |
74 (0,5826) |
82 (0,6457) |
80 (0,6299) |
69 (0,5433) |
98 (0,7717) |
98 (0,7717) |
118 (0,9291) |
R |
5 (0,0394) |
2 (0,0157) |
4 (0,0315) |
13 (0,1024) |
8 (0,0629) |
3 (0,0236) |
13 (0,1021) |
Na |
1,6172 |
1,5781 |
1,6719 |
1,8594 |
1,8594 |
1,7266 |
1,9922 |
Ne |
1,2808 |
1,3399 |
1,3127 |
1,4077 |
1,4449 |
1,4419 |
1,4966 |
He |
0,1770 |
0,1978 |
0,1965 |
0,2504 |
0,2700 |
0,2581 |
0,2905 |
П р и м е ч а н и е. Av1-Av6 — популяции (описание см. в разделе «Методика»); N — число амплифицированных фрагментов ДНК, Р95 и R — соответственно число (доля) полиморфных и редких фрагментов ДНК, Na и Ne — соответственно абсолютное и эффективное число аллелей на локус, He — ожидаемая гетерозиготность. |
|||||||
3. Показатели внутри- и межпопуляционного генетического разнообразия, оцененного IRAP-методом по индексу Шеннона, у Adonis vernalis (Пермский край) |
||||||||||
IRAP- |
Но |
Hsp |
Hpop |
Hpop/ |
(Hsp - Hpop)/ |
|||||
Av1 |
Av2 |
Av3 |
Av4 |
Av5 |
Av6 |
|||||
2175 |
0,3080 |
0,3538 |
0,3547 |
0,5004 |
0,3808 |
0,3582 |
0,4626 |
0,3733 |
0,8069 |
0,1930 |
2198 |
0,3111 |
0,3446 |
0,3624 |
0,3183 |
0,3126 |
0,3501 |
0,4217 |
0,3300 |
0,7825 |
0,2174 |
2202 |
0,2719 |
0,3537 |
0,3415 |
0,4140 |
0,4228 |
0,4292 |
0,5380 |
0,3683 |
0,6845 |
0,3154 |
2200 |
0,3322 |
0,2108 |
0,2161 |
0,4274 |
0,4139 |
0,2961 |
0,4528 |
0,3117 |
0,6884 |
0,3116 |
2197 |
0,1818 |
0,2363 |
0,2891 |
0,3198 |
0,5303 |
0,4800 |
0,4893 |
0,3350 |
0,6846 |
0,3153 |
Сред- |
0,2761 |
0,2966 |
0,3067 |
0,3866 |
0,4126 |
0,3850 |
0,4689 |
0,3383 |
0,7215 |
0,2785 |
П р и м е ч а н и е. Av1-Av6 — популяции (описание см. в разделе «Методика»); Ho и Hsp — индекс разнообразия Шеннона соответственно для популяции и суммарной выборки; Hpop — среднее значение индекса разнообразия Шеннона для популяций; Hpop/Hsp и (Hsp - Hpop)/Hsp — соответственно внутри- и межпопуляционное разнообразие. |
||||||||||
Число выявленных амплифицированных фрагментов ДНК варьировало по популяциям от 86 (Av2) до 112 (Av4) (табл. 2). Доля полиморфных фрагментов ДНК, амплифицированных со всеми IRAP-праймерами, колебалась по популяциям от 0,5433 (Av4) до 0,7717 (Av5 и Av6), число редких фрагментов ДНК (с частотой не более 0,05) — от 2 (Av2) до 13 (Av4) (в среднем на выборку — 8, или 6,29 %). Уникальные молекулярные маркеры ДНК отмечали в популяции Av2 (29). Ожидаемая гетерозиготность по локусам в общей выборке A. vernalis составила 0,2905 (при максимальном значении в Av5 и минимальном — в Av1) (см. табл. 2). Абсолютное и эффективное число аллелей на локус (в нашем случае на фрагмент ДНК) на общую выборку A. vernalis составило соответственно 1,9922 и 1,4966.
Ожидаемая доля гетерозиготных генотипов (или показатель общего генного разнообразия) на общую популяцию (HT) равна 0,3045, а в субпопуляциях (это показатель среднего выборочного генного разнообразия Hs) — 0,2250. Таким образом, средняя гетерозиготность растений A. vernalis в изученных популяциях ниже, чем в суммарной выборке. Определенный коэффициент подразделенности популяций (Gst) показал, что на межпопуляционную компоненту генетического разнообразия A. vernalis приходится 26,13 % разнообразия.
Оценка внутри- и межпопуляционного разнообразия на основе информационного индекса Шеннона (табл. 3) продемонстрировала, что среднее значение индексов разнообразия Шеннона (Hpop) в изученных популяциях A. vernalis, рассчитанное по IRAP-праймерам, составило 0,3383; наибольший показатель (Ho) — у растений Av5, в общей выборке (Hsp) — 0,4689. На долю внутри- и межпопуляционного разнообразия приходится соответственно 72,15 и 27,85 %, что хорошо согласуется с данными подразделенности популяций (Gst). Число редких аллелей (R) было выше у растений из Av4, уникальные молекулярные маркеры отмечали в Av2.
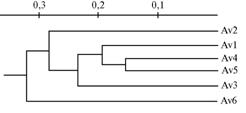 |
Рис. 2. UPGMA-дендрограмма генетического сходства популяцийAdonisvernalis(Av1-Av6), построенная на основании полиморфизма IRAP-маркеров. |
Наименьшее генетическое расстояние между исследуемыми популяциями A. vernalis, рассчитанное по M. Nei (20), обнаружили между Av4 и Av5 (0,0475), наиболее генетически удаленными оказались популяции Av3 и Av6 (0,1707) (рис. 2).
Итак, у растений шести изученных популяций Adonisvernalis выявлено 127 молекулярных IRAP-маркеров. Доля полиморфных фрагментов ДНК оказалась достаточно высокой (0,9291), что, на наш взгляд, обусловлено реликтовостью вида. Наименьшие показатели генетического разнообразия отмечены в популяции Av1 (P95 = 58,26 %; Ho = 0,2761; He = 0,1770), наибольшие — в Av5 (P95 = 77,17 %; Ho = 0,4126; He = 0,2700). Значительные число и доля редких аллелей характерны для популяции Av4, уникальные молекулярные маркеры — для Av2. Растения популяции Av6подвергаются сильному антропогенному воздействию, что проявляется в сокращении ее численности (29). Поскольку на долю межпопуляционного разнообразия приходится 27,85 %, изученные популяции сильно дифференцированы, что в основном характерно для редких видов растений. Отметим, что применение стабильных высокополиморфных IRAP-маркеров рекомендуется для изучения геномов ресурсных, продовольственных и лекарственных растений, перспективно для изучения геномов реликтовых видов, а также мобильных генетических элементов — ретротранспозонов.
Л И Т Е Р А Т У Р А
1. D e V i e n n e D. Molecular markers in plant genetics and biotechnology. Enfield, NH, USA, 2003.
2. Г о с т и м с к и й С.А., К о к а е в а З.Г., К о н о в а л о в Ф.А. Изучение организации и изменчивости генома растений с помощью молекулярных маркеров. Генетика, 2005, 41(4): 480-490.
3. С а л и н а Е.А., Е г о р о в а Е.М., A д о н и н а И.Г. и др. ДНК-маркеры для генотипирования линий мягкой пшеницы (Triticum aestivum L.) с генетическим материалом Aegilops speltoides Tausch и Triticum timopheeviiZhuk. Вест. ВОГИС, 2008, 12(4): 620-629.
4. K u m a r A., B e n n e t z e n J. Plant retrotransposons. Annu. Rev. Genet., 1999, 33: 479-532.
5. K a l e n d a r R., G r o b T., R e g i n a M. e.a. IRAP and REMAP: Two new retrotransposon-based DNA fingerprinting techniques. Theor. Appl. Genet., 1999, 98: 704-711.
6. K a l e n d a r R., T a n s k a n e n J., I m m o n e n S. e.a. Genome evolution of wild barley (Hordeum spontaneum) by BARE-1 retrotransposon dynamics in response to sharp microclimatic 7. divergence. PNAS USA, 2000, 97(12): 6603-6607.
7. Г л а з к о Т.Т., Г л а з к о В.И. Молекулярно-генетические подходы в селекции зерновых. Изв. ТСХА, 2006, 4: 100-107.
8. Ц в е т к о в И.А., И в а н о в А.Н., Г л а з к о В.И. Генетическая дифференциация сортов риса по IRAP-маркерам. Изв. ТСХА, 2006, 4: 155-159.
9. К о к а е в а З.Г., Г о с т и м с к и й С.А. Изучение генетического разнообразия сортов, линий и мутантов гороха посевного с помощью ДНК-маркеров, полученных на основе ретротранспозонов. С.-х. биол., 2006, 5: 1-5.
10. Б о р о н н и к о в а С.В., К о к а е в а З.Г., Т и х о м и р о в а Н.Н. Генетическое разнообразие популяций некоторых редких видов растений Пермского края при антропогенных воздействиях. Мат. Межд. науч.-практ. конф. «Антропогенная динамика природной среды». Пермь, 2006, т. II: 107-112.
11. К а л е н д а р ь Р.Н., Б о р о н н и к о в а С.В. Анализ молекулярно-генетического полиморфизма природных популяций редких видов растений Урала с помощью ретротранспозонов. Мат. IV Московского межд. конгр. «Биотехнология: состояние и перспективы развития». М., 2007, ч. 2: 121.
12. Красная книга Пермского края /Под науч. ред. А.И. Шепель. Пермь, 2008.
13. Б е л к о в с к а я Т.П., О в е с н о в С.А. Охраняемые виды растений Пермской области. В кн: Растительный мир Прикамья. Пермь, 1988: 155-161.
14. П о ш к у р л а т А.П. Род горицвет — Adonis L. Систематика, распространение, биология. М., 2000.
15. T o r r e s A.M., W e e d e n N.F., M a r t i n A. Linkage among sozyme, RFLP and RAPD markers in Vicia faba. Theor. Appl. Genet., 1993, 5: 937-945.
16. Y e h F.C., Y o u n g R.C., M a o J. e.a. POPGENE, the Microsoft Windows-based user-friendly software for population genetic analysis of co-dominant and dominant markers and quantitative traits. Edmonton, Alta, 1999.
17. P e a k a l l R., S m o u s e P.E. GenAlEx6: Genetic analysis in Excel. Population genetic software for teaching and research. Mol. Ecol. Not., 2006, 6: 288-295.
18. W i l l i a m s J.G.K., K u b e l i k A.R., L i v a k K.J. e.a. DNA polymorphisms amplified by arbitrary primers are useful as genetic markers. Nucl. Acids Res., 1990, 18: 6531-6535.
19. K i m u r a M., C r o w J.F. The number of alleles that can be maintained in a finite population. Genetics (US), 1964, 49: 725-738.
20. N e i M. Molecular evolutionary genetics. N.Y., 1987.
21. S c h a n n o n C.E., W e a v e r W. The mathematical theory of communication. Urbana Univ. of Illinois Press, 1949.
22. C h a l m e r s K.J., W a u g h R., S p r e n t J.I. e.a. Detection of genetic variation between and within populations of Gliricidia sepium and G. maculata using RAPD markers. Heredity, 1992, 69: 465-472.
23. N e i M. Molecular population genetics and evolution. Amsterdam, 1975.
24. N e i M. Genetic distance between populations. Amer. Natur., 1972, 106: 283-292.
25. Ж и в о т о в с к и й Л.А. Статистические методы анализа частот генов в природных популяциях. В сб.: Итоги науки и техники. Общая генетика (ВИНИТИ АН СССР). М., 1983, 8: 76-104.
26. Ж и в о т о в с к и й Л.А. Популяционная биометрия. М., 1991.
27. Л а к и н Г.Ф. Биометрия. Уч. пос. 4-е изд., перераб. и доп. М., 1990.
28. К о к а е в а З.Г., Б о р о н н и к о в а С.В., Т и х о м и р о в а Н.Н. и др. Анализ генетического разнообразия популяционных систем редких и ресурсных видов растений. Мат. IV Межд. науч. конф. «Биотехнология — охране окружающей среды» (сб. МОИП). М., 2006: 75-79.
29. Б о р о н н и к о в а С.В. Молекулярно-генетическая идентификация и паспортизация редких и находящихся под угрозой исчезновения видов растений. Пермь, 2008.
30. Б е л ь т ю к о в а Н.Н., Б о р о н н и к о в а С.В., К а р и е в а Л.Г. Исследование генетического полиморфизма лекарственного вида Digitalisgrandiflora Mill. c использованием анализа ретротранспозонов. Аграрная Россия, 2009, 4: 20-23.
ESTIMATION OF INTRA- AND INTERPOPULATION GENETIC VARIETY OF Adonis vernalis L. MEDICINAL SPECIES WITH USE OF IRAP-MARKERS
S.V. Boronnikova, N.N. Tikhomirova, O.A. Kravchenko
In plants of six populations of Adonis vernalis L. of rare for Permskii area medicinal species the authors studied a DNA polymorphism by the IRAP-method and estimated their genetic variety on 127 isolated IRAP-markers. Among the factors, which determined a high level of genetic variety, the relic properties of the species have the decisive importance, probably.
Key words: IRAP markers, polymorphism, genetic diversity, the preservation of gene pool, molecular marker selection.
ФГОУ ВПО Пермский государственный университет, |
Поступила в редакцию |













